Вернуться на страницу семестра
Построение разных форм ДНК
С помощью программы fiber пакета 3DNA я построила A-, B- и Z-форму дуплекса ДНК, последовательность одной из нитей которого представляет собой 5 раз повторенную последовательность "gatc".
Результаты вы можете увидеть в апплете Jmol.
PDB файл A-формы
PDB файл B-формы
PDB файл Z-формы
Параметры разных форм ДНК, ориентация атомов азотистых оснований по отношению к бороздкам
Определим ориентацию атомов азотистых оснований по отношению к большой и малой бороздкам ДНК в B-форме, как и в предыдущей работе, на примере тимина (увидеть рассматриваемый остаток вы можете в апплете).
В сторону большой бороздки обращены атомы T7.N3, T7.C4, T7.O4, T7.C5, T7.C6, T7.C7
В сторону малой бороздки обращены атомы T7.O2, T7.C2, T7.N1.
На рисунке 1 выделены красным цветом атомы, смотрящие в сторону большой бороздки, синим - в сторону малой.
Рисунок 1. Тимин с отмеченной ориентацией атомов относительно бороздок
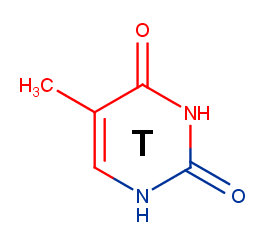
Таблица 1. Параметры разных форм ДНК
| A-форма | B-форма | Z-форма |
| Тип спирали | Правозакрученная | Правозакрученная | Левозакрученная |
| Шаг спирали (Å) | 28.03 | 33.75 | 43.5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки (Å) | 16.81 (7-36) | 17.21 (7-31) | 16.08 (12-28) |
| Ширина малой бороздки (Å) | 7.98 (7-28) | 11.69 (7-38) | 8.68 (11-35) |
Определение параметров тРНК 1O0B и ДНК 1P47 с помощью программ пакета 3DNA
Пакет 3DNA пока работает только со старым форматом PDB, поэтому для начала нужно отформатирвоать PDB файл. 3D-структуру исследуемых тРНК и ДНК вы можете
увидеть на 4 и 5 изображении в апплете Jmol вверху страницы.
Определение торсионных углов нуклеотидов
Рисунок 2. Торсионные углы нуклеиновых кислот и их значения (по презентации)
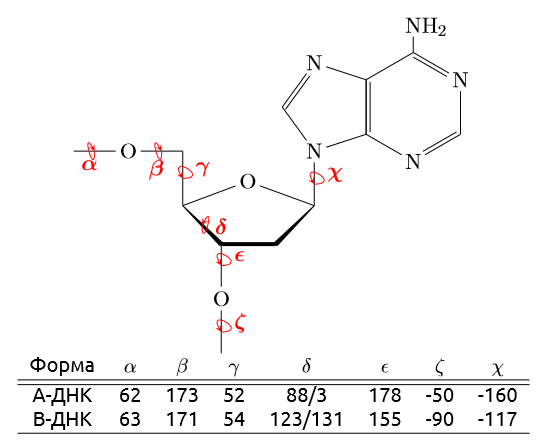
C помощью комманд find_pair, analyze получен выходной файл с описанием водородных связей, значениями всех торсионных углов, шириной малой и большой бороздки.
В Excel определены среднее значение каждого из торсионных углов (краевые нуклеотиды не рассматривались) для тРНК и ДНК.
Таблица 2. Торсионные углы в НК
| α | β | γ | δ | ε | ζ | χ |
| тРНК | -24 | 25 | 32 | 84 | -120 | -66 | -139 |
| А-ДНК | -52 | 175 | 42 | 79 | -148 | -75 | -157 |
| В-ДНК | -38 | 57 | 21 | 134 | -110 | -101 | -120 |
Ссылка на проект Excel с рассчётами
Для вывода о схожести тРНК с какой-либо формой ДНК будем ориентироваться и на таблицу, и на рисунок 2, предоставленный авторитетным источником.
Принимая во внимание большие разночтения и погрешности, я все-таки могу предположить, что тРНК больше похожа на А-форму. Визуально вы можете сравнить их в апплете вверху страницы.
Самое большое отклонение от средних значений в тРНК у урацила 15, а в B-ДНК цитозин 18. Все рассчёты можно увидеть по ссылке выше.
Определение структуры водородных связей
Первый стебель тРНК
|
Strand I Strand II
(0.014) ....>B:.902_:[..G]G-----C[..C]:.971_:B<.... (0.007)
(0.007) ....>B:.903_:[..G]G-----C[..C]:.970_:B<.... (0.007)
(0.005) ....>B:.904_:[..G]G-----C[..C]:.969_:B<.... (0.005)
(0.009) ....>B:.905_:[..G]G-----C[..C]:.968_:B<.... (0.007)
(0.012) ....>B:.906_:[..U]U-----A[..A]:.967_:B<.... (0.010)
(0.012) ....>B:.907_:[..A]A-----U[..U]:.966_:B<.... (0.009)
|
Второй стебель тРНК
|
(0.005) ....>B:.949_:[..C]C-----G[..G]:.965_:B<.... (0.009)
(0.004) ....>B:.950_:[..G]G-----C[..C]:.964_:B<.... (0.003)
(0.003) ....>B:.951_:[..A]A-----U[..U]:.963_:B<.... (0.002)
(0.007) ....>B:.952_:[..G]G-----C[..C]:.962_:B<.... (0.005)
(0.005) ....>B:.953_:[..G]G-----C[..C]:.961_:B<.... (0.006)
(0.002) ....>B:.954_:[..U]U-**--A[..A]:.958_:B<.... (0.010)
|
Третий стебель тРНК
|
(0.009) ....>B:.937_:[..A]A-**--U[..U]:.933_:B<.... (0.006)
(0.004) ....>B:.938_:[..U]U-**--U[..U]:.932_:B<.... (0.005)
(0.004) ....>B:.939_:[..U]U-----A[..A]:.931_:B<.... (0.003)
(0.003) ....>B:.940_:[..C]C-*---G[..G]:.930_:B<.... (0.009)
(0.005) ....>B:.941_:[..C]C-----G[..G]:.929_:B<.... (0.010)
(0.006) ....>B:.942_:[..G]G-----C[..C]:.928_:B<.... (0.005)
(0.009) ....>B:.943_:[..G]G-----C[..C]:.927_:B<.... (0.008)
(0.005) ....>B:.944_:[..C]C-**--A[..A]:.926_:B<.... (0.009)
|
Четвёртый стебель тРНК
|
(0.011) ....>B:.910_:[..G]G-----C[..C]:.925_:B<.... (0.005)
(0.002) ....>B:.911_:[..C]C-----G[..G]:.924_:B<.... (0.009)
(0.002) ....>B:.912_:[..C]C-----G[..G]:.923_:B<.... (0.011)
|
Стабилизирующие структуры
|
(0.004) ....>B:.913_:[..A]A-**+-A[..A]:.945_:B<.... (0.005)
(0.005) ....>B:.914_:[..A]A-**--U[..U]:.908_:B<.... (0.009)
(0.007) ....>B:.915_:[..G]G-**+-C[..C]:.948_:B<.... (0.019)
(0.026) ....>B:.919_:[..G]G-----C[..C]:.956_:B<.... (0.003)
(0.003) ....>B:.955_:[..U]U-**+-G[..G]:.918_:B<.... (0.005)
|
В тРНК есть неканонические взаимодействия азотистых оснований:
U-A (связи O2 - N6 и N3 - N7)
U-G (O2 - N2)
A-U (N7 - O2 и N6 - O2)
U-U (O4 - N3)
C-G (O2 - N2 и N3 - N1)
C-A (O2 - N1 и N3 - N6)
A-A (N6 - N1 и N1 - N6)
G-C (N1 - O2 и N2 - N3)
Стекинг-взаимодействия
В выходном файле также были определены параметры стекинговых взаимодействий - площади перекрывания динуклеотидов.
Пары с максимальной и минимальной площадью перекрывния
|
step i1-i2 i1-j2 j1-i2 j1-j2 sum
GC/GC 4.56( 1.74) 0.00( 0.00) 0.00( 0.00) 6.96( 3.74) 11.52( 5.47)
CG/CA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
|
С помощью команд "ex_str -8 stacking.pdb step8.pdb" и "stack2img -cdolt step8.pdb step8.ps" я получила изображения этих взайимодействий.
Рисунок 3. Перекрывание GC/GC
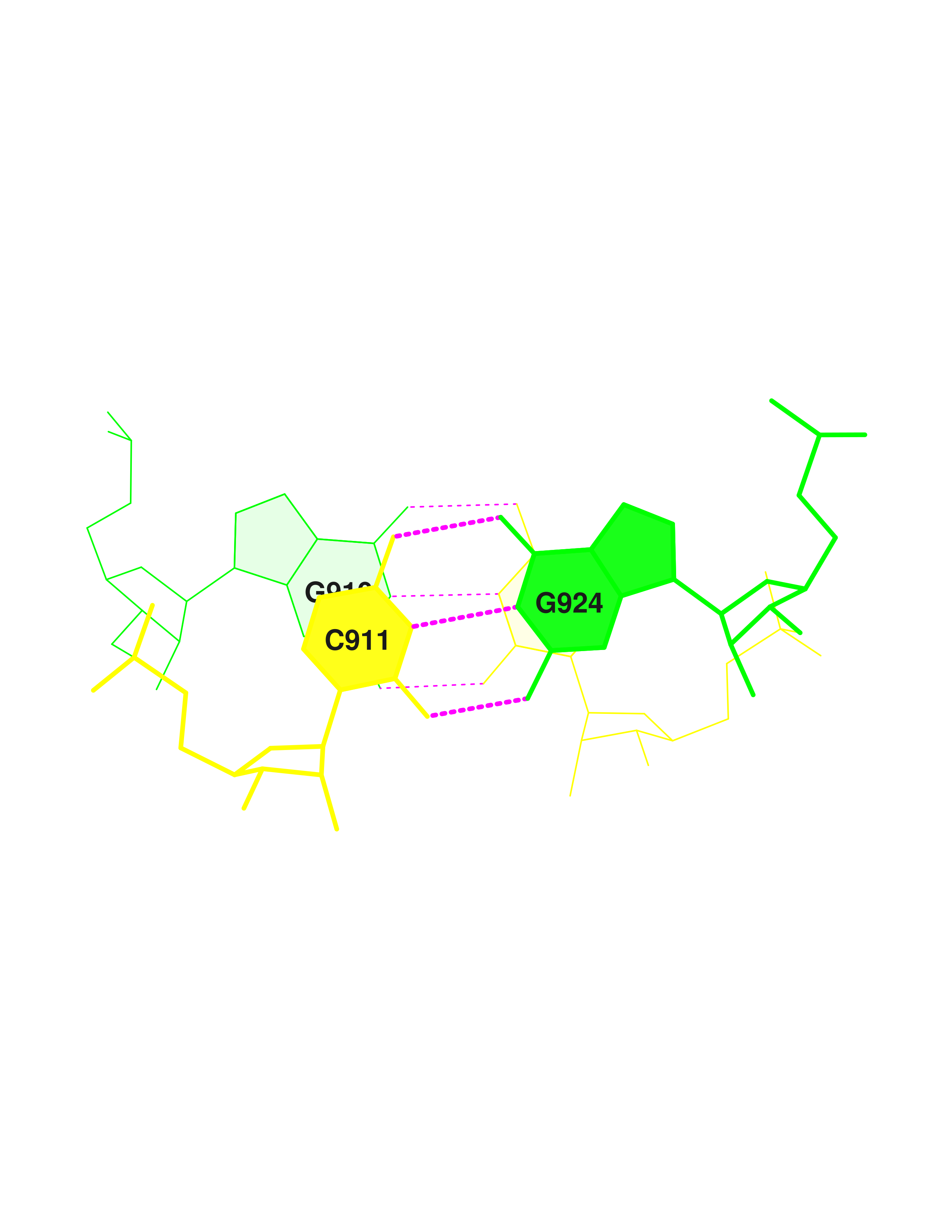
|
| Рисунок 4. Перекрывание CG/CA
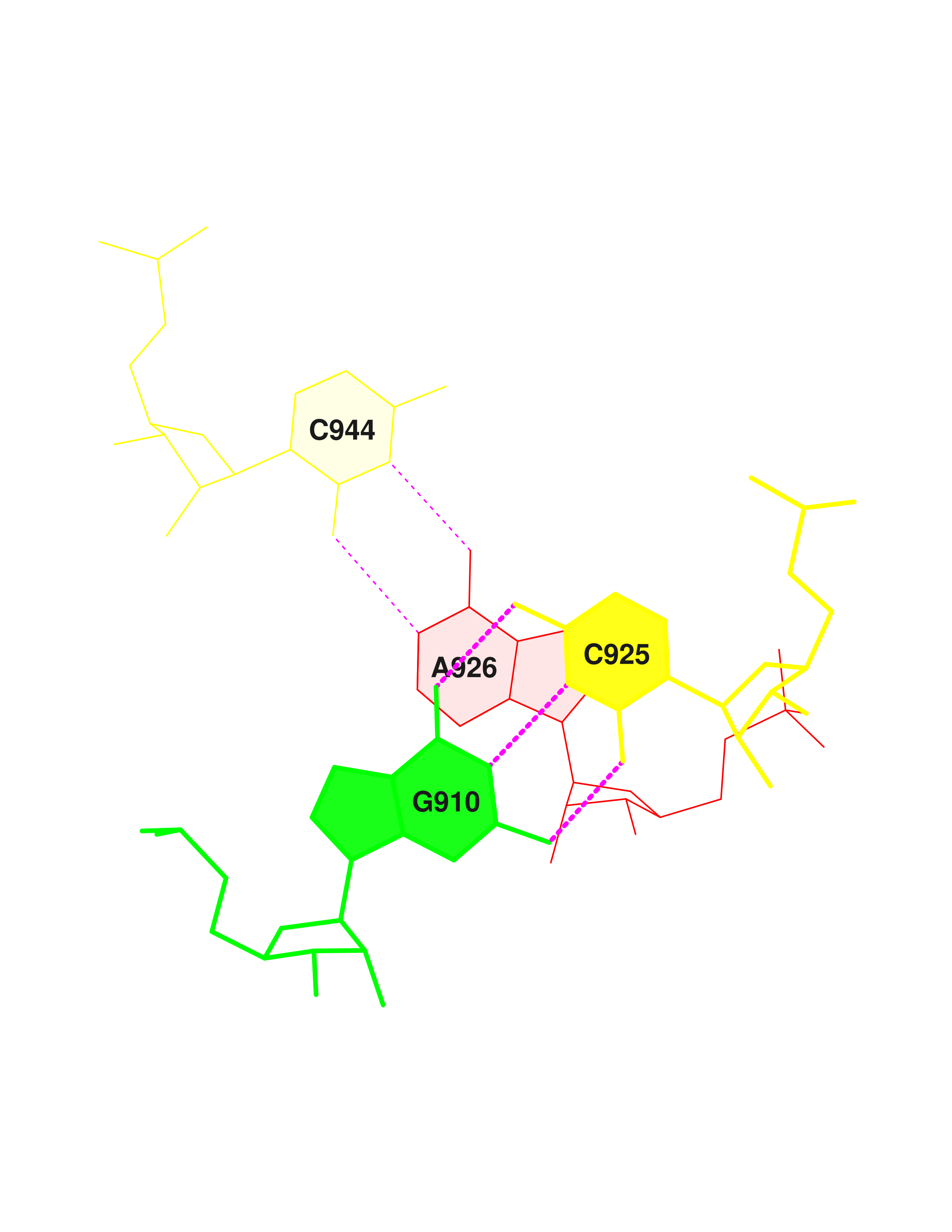
|
|









