



 |  |  |  |
Чтение последовательностей по Сэнгеру |
Вернуться на страницу семестра Прочтение последовательности ДНК на основании данных, полученных из капиллярного секвенатора по СэнгеруКапиллярный секвенатор выдает файлы с хроматограммой и автоматически прочтённой последовательностью в формате .ab1. Дано два файла в формате .ab1, соответствующие прочтению прямой и обратной цепочки секвенируемой ДНК. Для просмотра хроматограмм и редактирования автоматического прочтения используется программа Chromas (Lite). Таблица 1. Границы нечитаемых 5'- и 3'-участков в каждой последовательности (обратная в режиме Reverse)
Проблемный нуклеотид — тот, по которому вы приняли решение, отличное от предложенного программой, или вы согласились с программой, но необходимо было проанализировать хроматограммы и принять решение.
Проблемные нуклеотиды в последовательности выделены в проекте выравнивания строчными буквами.
Таблица 2. Примеры проблемных нуклеотидов или полиморфизмов в последовательности (прямая - вверху, обратная - внизу)
С помощью программы needle я сделала выравнивание двух последовательностей - исправленные мною последовательности хроматограммы (все исправления маленькими буквами). Fasta-файлы с ридом прямой цепи и обратной. С помощью Jalview получила визуализированное выравнивание, приведённое ниже. Выравнивание в Jalview Ссылка на проект выравнивания Итоговая консенсусная последовательность в fasta Пример нечитаемого фрагмента хроматограммыНа рисунке 1 мы можем увидеть пример небольшого нечитаемого участка в середине секвенирования обратной последовательности, которое рассматривали ранее. Это так называемое пятно краски. Но в данном случае, нам повезло, потому что исходную последовательность легко восстановить по прямой. Рисунок 1. Нечитаемый фрагмент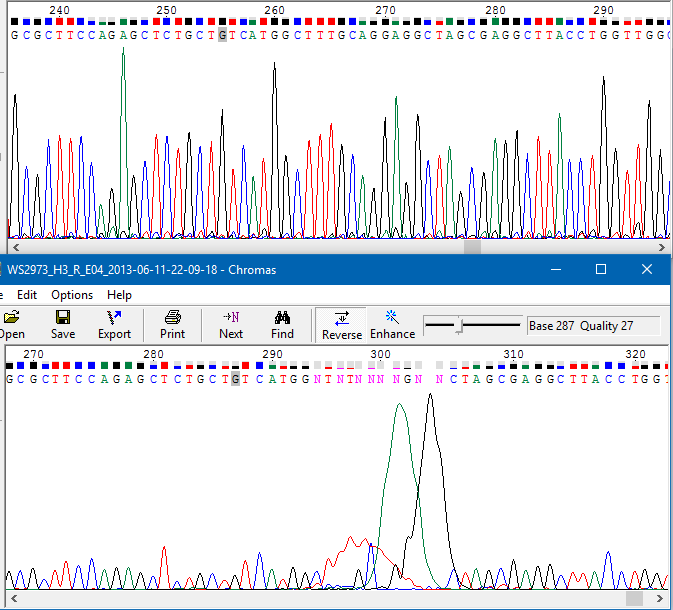
|