



 |  |  |  |
Домены. Pfam. HMM профиль |
Вернуться на страницу семестра Цель: Реконструкция эволюции доменной архитектурыЗадание 1. Построить выравнивание представителей домена Pfam белков с разной доменной архитектуройВыбор доменаДля работы был выбран домен Sec23/Sec24 trunk. Ссылка на страницу в pfam.
В Jalview было открыто полученное выравнивание доменных участков всех белков, содержащих исследуемый домен (раскраска Clustalx). На сайте pfam найдено соответствие структуры Uniprot ID SC23A_HUMAN и PDB 5KYY. Но при добавлении в проект возникает ошибка. Выбор двух архитектур, включающих этот доменБыли выбраны следующие архитектуры:
Первая состоит из доменов PF04810 PF04811. PF04810 это домен Sec23/Sec24 zinc finger(цинковый палец). 
Вторая состоит из доменов PF04810 PF04811 PF08033. Добавился домен PF08033 - Sec23/Sec24 beta-sandwich domain. С помощью скрипта и команды python swisspfam-to-xls.py -i /srv/databases/pfam/swisspfam.gz -z -p PF04811 -o out2.xls
В качестве таксона выбрано Eukaryota. Подтаксоны: Viridiplantae (V), Metazoa (M).
В новом выравнивании удалены пустые столбцы, неправильно выравненные последовательности, N и С концы последовательностей. Для удобства выравнивание разбито на 4 группы по архитектуре и таксономии, окрашено ClustalX. Скачать выравнивание Задание 2: построение филогенетического дерева домена Для наглядности в дереве последовательности переименованы и в начале добавлены идентификаторы.
Обозначения: 1 - двухдоменная архитектура, 2 - трёхдоменная архитектура. Viridiplantae (V), Metazoa (M).
Рисунок 1. Филогенетическое дерево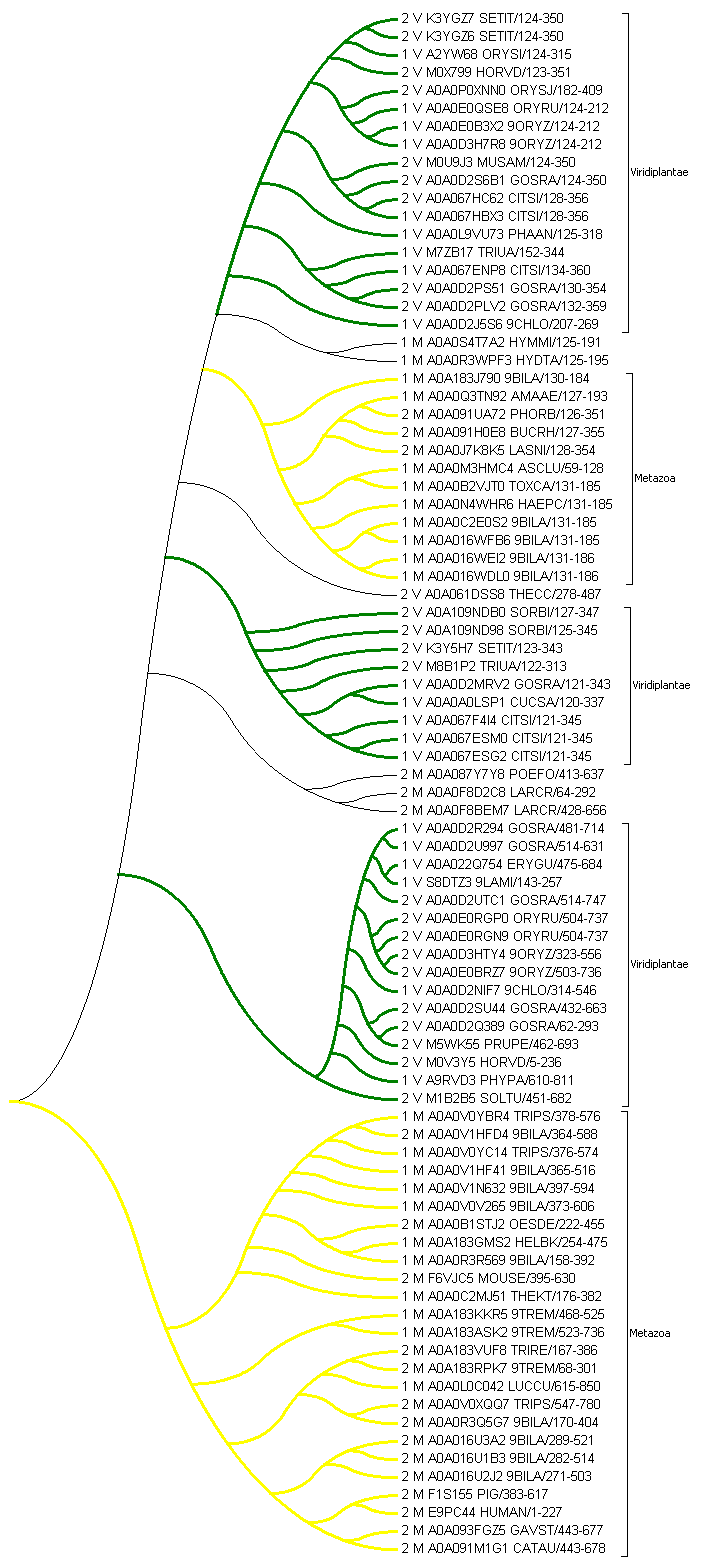
Укоренила дерево так, чтобы разделились самые крупные ветки. Выделила крупные ветви, где встечаются представители только отдного царства. Так как архитектуры по сути различаются лишь добавление дополнительного домена, то эволюция могла происходить с помощью вставок и делеций этого домена PF08033. При этом вставка домена ведёт к превращению архитектуры 1 в 2, а делеция из 2 в 1. Попробуем отследить эти эволюционные изменения и их частоту в дереве. Начну с верхней ветви Viridiplantae. Эволюцию верхних 4 белков может представлять собой разделение 1 V и 2 V делецией от общего предка. Эволюция следующих 4 представителей достаточно реалистичная - сразу отделяется представитель 2 V, а потом идёт эволюция внутри 1 V. То же самое можно сказать о следующих 4 белках, где в самом конце произошла делеция с образованием 1 V. Хорошо прослеживается это также во второй выделенной мною ветке зелёных растений. Делеция домена PF08033 привела к дивергенции, ветки разделены как нужно. Также хочу рассмотреть ветки Metazoa на примере менее крупной из двух отмеченных мною ветвей. Если дерево отражает эволюцию правильно, то можно увиеть интересную историю белка 1 M A0A0Q3TN92. В основном эта ветка о двухдоменной архитектуре 1, но происходит вставка домена и появляются представители 2 M. Затем внутри этих белков происходит делеция и снова появляется представитель 1 M. Таким образом, белок 1 M A0A0Q3TN92 возвращается к истокам, ведь его первоначальный предок имел архитектуру 1. О частоте делеций и вставок. В царстве Viridiplantae в отменных мною ветвях наблюдается 7 переходов 2->1 (делеция) и 1 переход 1->2 (вставка). В Metazoa 2 делеции и 4 вставки. Суммарно по 2 царствам делеции происходят чаще. Скобочная формула дерева выглядит так: ((((((((((((((2_V_K3YGZ7_SETIT/124-350,2_V_K3YGZ6_SETIT/124-350),1_V_A2YW68_ORYSI/124-315),2_V_M0X799_HORVD/123-351),(2_V_A0A0P0XNN0_ORYSJ/182-409,(1_V_A0A0E0QSE8_ORYRU/124-212,(1_V_A0A0E0B3X2_9ORYZ/124-212,1_V_A0A0D3H7R8_9ORYZ/124-212)))),(2_V_M0U9J3_MUSAM/124-350,(2_V_A0A0D2S6B1_GOSRA/124-350,(2_V_A0A067HC62_CITSI/128-356,1_V_A0A067HBX3_CITSI/128-356)))),1_V_A0A0L9VU73_PHAAN/125-318),(1_V_M7ZB17_TRIUA/152-344,(1_V_A0A067ENP8_CITSI/134-360,(2_V_A0A0D2PS51_GOSRA/130-354,2_V_A0A0D2PLV2_GOSRA/132-359)))),1_V_A0A0D2J5S6_9CHLO/207-269),(1_M_A0A0S4T7A2_HYMMI/125-191,1_M_A0A0R3WPF3_HYDTA/125-195)),(1_M_A0A183J790_9BILA/130-184,((((1_M_A0A0Q3TN92_AMAAE/127-193,2_M_A0A091UA72_PHORB/126-351),2_M_A0A091H0E8_BUCRH/127-355),2_M_A0A0J7K8K5_LASNI/128-354),((1_M_A0A0M3HMC4_ASCLU/59-128,1_M_A0A0B2VJT0_TOXCA/131-185),(1_M_A0A0N4WHR6_HAEPC/131-185,((1_M_A0A0C2E0S2_9BILA/131-185,1_M_A0A016WFB6_9BILA/131-185),(1_M_A0A016WEI2_9BILA/131-186,1_M_A0A016WDL0_9BILA/131-186))))))),2_V_A0A061DSS8_THECC/278-487),(2_V_A0A109NDB0_SORBI/127-347,(2_V_A0A109ND98_SORBI/125-345,(2_V_K3Y5H7_SETIT/123-343,(2_V_M8B1P2_TRIUA/122-313,((1_V_A0A0D2MRV2_GOSRA/121-343,1_V_A0A0A0LSP1_CUCSA/120-337),(1_V_A0A067F4I4_CITSI/121-345,(1_V_A0A067ESM0_CITSI/121-345,1_V_A0A067ESG2_CITSI/121-345)))))))),(2_M_A0A087Y7Y8_POEFO/413-637,(2_M_A0A0F8D2C8_LARCR/64-292,2_M_A0A0F8BEM7_LARCR/428-656))),(((((((((1_V_A0A0D2R294_GOSRA/481-714,1_V_A0A0D2U997_GOSRA/514-631),(1_V_A0A022Q754_ERYGU/475-684,1_V_S8DTZ3_9LAMI/143-257)),2_V_A0A0D2UTC1_GOSRA/514-747),(2_V_A0A0E0RGP0_ORYRU/504-737,(2_V_A0A0E0RGN9_ORYRU/504-737,(2_V_A0A0D3HTY4_9ORYZ/323-556,2_V_A0A0E0BRZ7_9ORYZ/503-736)))),1_V_A0A0D2NIF7_9CHLO/314-546),(2_V_A0A0D2SU44_GOSRA/432-663,(2_V_A0A0D2Q389_GOSRA/62-293,2_V_M5WK55_PRUPE/462-693))),2_V_M0V3Y5_HORVD/5-236),1_V_A9RVD3_PHYPA/610-811),2_V_M1B2B5_SOLTU/451-682),(((((((((1_M_A0A0V0YBR4_TRIPS/378-576,2_M_A0A0V1HFD4_9BILA/364-588),1_M_A0A0V0YC14_TRIPS/376-574),1_M_A0A0V1HF41_9BILA/365-516),1_M_A0A0V1N632_9BILA/397-594),1_M_A0A0V0V265_9BILA/373-606),(2_M_A0A0B1STJ2_OESDE/222-455,(1_M_A0A183GMS2_HELBK/254-475,1_M_A0A0R3R569_9BILA/158-392))),2_M_F6VJC5_MOUSE/395-630),1_M_A0A0C2MJ51_THEKT/176-382),((1_M_A0A183KKR5_9TREM/468-525,1_M_A0A183ASK2_9TREM/523-736),((((2_M_A0A183VUF8_TRIRE/167-386,2_M_A0A183RPK7_9TREM/68-301),1_M_A0A0L0C042_LUCCU/615-850),(2_M_A0A0V0XQQ7_TRIPS/547-780,2_M_A0A0R3Q5G7_9BILA/170-404)),(((2_M_A0A016U3A2_9BILA/289-521,2_M_A0A016U1B3_9BILA/282-514),2_M_A0A016U2J2_9BILA/271-503),((2_M_F1S155_PIG/383-617,2_M_E9PC44_HUMAN/1-227),(2_M_A0A093FGZ5_GAVST/443-677,2_M_A0A091M1G1_CATAU/443-678))))))); Источники |