



 |  |  |  |
Умение: восстановить кристалл из PDB файла |
Вернуться на страницу семестра Описание взаимодействия белка 6OM5 с субъединицами из соседних ячеекЦель работы: белковый кристалл поддерживается взаимодействия субъединиц белков (и др. молекул). Эти взаимодействия могут быть природными — реализующимися in vivo или in vitro — или случайными.
Для различения надо уметь эти контакты визуализировать.
Рисунок 1. Вторичная структура белка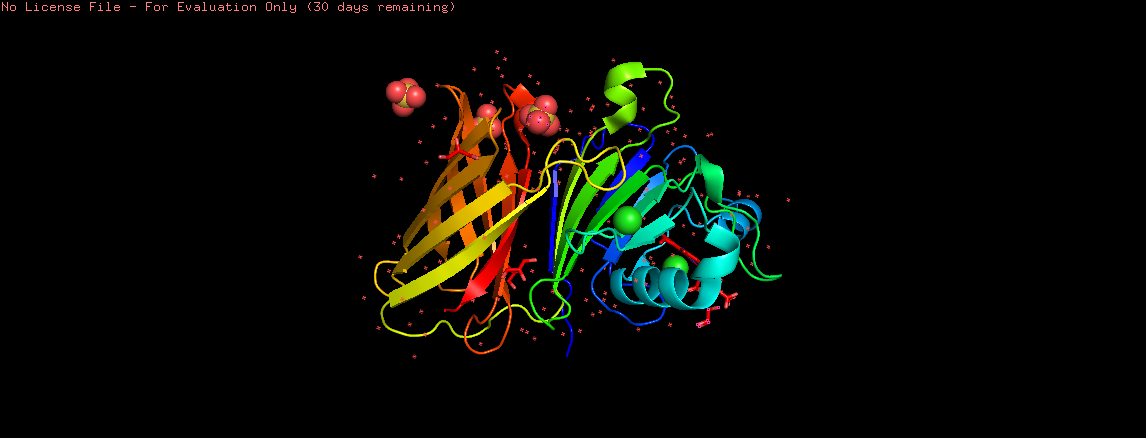
Для визуализации кристалла я использовала следующие команды в PyMOL: PyMOL> fetch 6om5 PyMOL> show cell PyMOL> symexp sym, 6om5, 6om5, 1 (показать соседние молекулы на таком расстоянии) PyMOL> show sticks PyMOL> distance hbonds, all, all, 3.2 (показать связи между молекулами 2 множеств, если они меньше 3.2 ангстрем) Рисунок 2. Положение белка в ячейке. Видно, что 3 направляющих вектора не попарно перпендикулярны
Рисунок 3. Симметрия в ячейке (6 регулярных структур в 1 ячейке)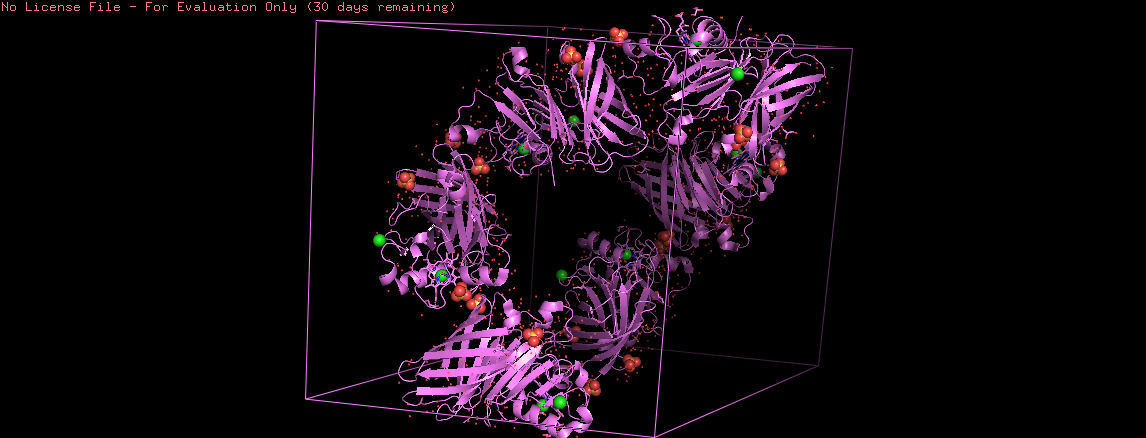
Рисунок 4. Молекулы на расстоянии не больше 1 А
Белок контактирует с 4 соседними молекулами вне ячейки. Я выбрала 1 из молекул. Она находилась ближе всего, я определила это, уменьшая срез. Затем, нашла участки, где молекулы сближены сильнее, чем на 3.2 ангстрем. Интересно получилось, что лиганды SO4 наложились друг на друга со смещением (рис. 5), как раз из-за этого это были самые близкие друг к другу молекулы. Я оставила аминокислоты, которые взаимодействуют с лигандом. Это изолейцины. То, что SO4 наложились со смещением, а также то, что водородных связей очень мало, может указывать на то, что это взаимодействие - артефакт кристаллизации и не играет биологической роли. Я также рассмотрела взаимодействия между другими молекулами из разных ячеек. Водородных связей было найдено мало, а найденные противоречивы (рис. 6): сближение аспартата и глутамата. Это подтверждает предыдущий вывод. Рисунок 5. Взаимодействие с SO4
Рисунок 6. Взаимодействие отрицательно заряженных остатков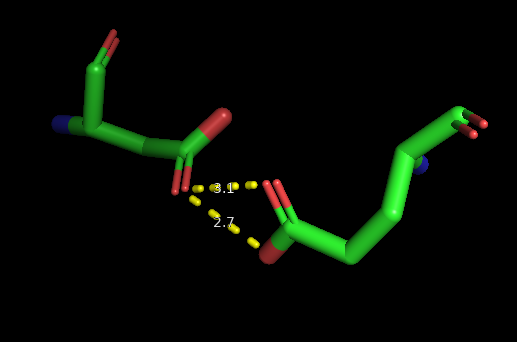
Странные белковые цепи в структуре ДНК-белкового комплекса 3HDDАльфа-спирали находятся "на краю" ДНК, что не может не вызвать подозрений (рис. 7)! Попробуем восстановить соседние ячейки, чтобы разобраться (рис. 8). Получается, что белковая цепь не находится на краю, так как взаимодействует с соседней цепью ДНК. Рисунок 7. 3HDD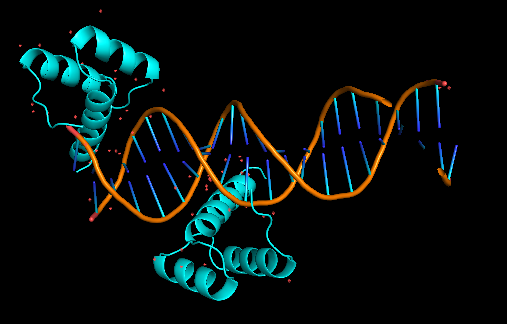
Рисунок 8. 3HDD и структуры из соседних ячеек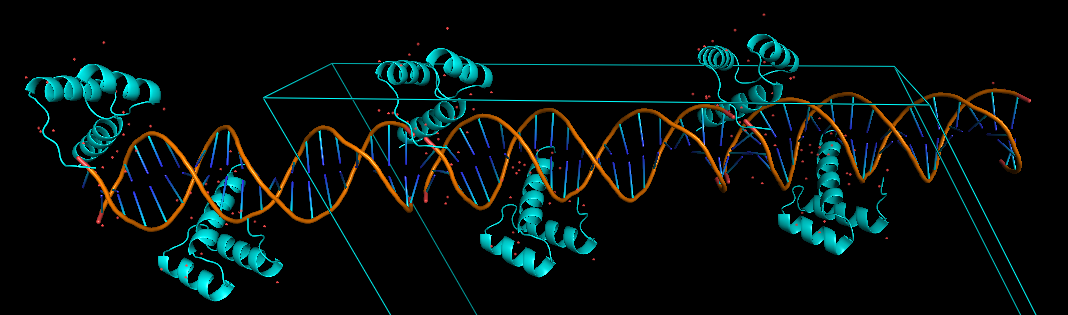
Источники PyMOL |