 |
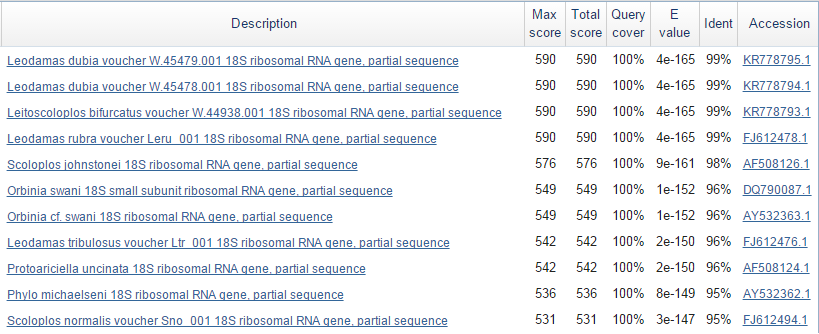
Рис1. Выдача BLAST'а.

| Алгоритм | Число находок | E-value худшей находки | Ident худшей находки | Query cover худшей находки | Уникальные для алгоритма находки |
| megablast | 48 | 5e-42 | 89% | 39% | - |
| discontiguous megablast | 51 | 4e-44 | 89% | 39% | - |
| blastn | 51 | 4e-44 | 89% | 39% | - |
Рис2. Выдача megablast.
Рис3. Выдача discontiguous megablast.
Рис4. Выдача blastn.
| Название | Функция | Число находок | Лучшая находка | Ident лучшей | Query cover лучшей |
| Heat shock cognate 71 kDa protein (HSP7C_HUMAN) | Участие в фолдинге белков | 72 | Sus scrofa heat shock 70kDa protein 8 (HSPA8), mRNA | 94% | 94% |
| Telomerase reverse transcriptase (TERT_HUMAN) | Удлинение теломер | 6 | Sus scrofa telomerase reverse transcriptase (TERT), mRNA | 66% | 100% |
| Citrate synthase, mitochondrial (CISY_HUMAN) | Участие в цикле Кребса, синтез цитрата | 20 | Porcine citrate synthase mRNA, complete cds | 96% | 97% |
| DNA-directed RNA polymerase II subunit RPB1 (RPB1_HUMAN) | Входит в транскрибирующий комплекс | 8 | PREDICTED: Sus scrofa polymerase (RNA) II (DNA directed) polypeptide A, 220kDa (POLR2A), mRNA | 98% | 54% |
| Polyadenylate-binding protein 2 (PABP2_HUMAN) | Регуляция стабильности и трансляции мРНК | 75 | Sus scrofa poly(A) binding protein, nuclear 1 (PABPN1), mRNA | 86% | 63% |
Параметры всех находок очень неплохие, что довольно понятно, ведь свинья и человек не так уж далеки друг от друга на древе жизни.
Для выполнения этого задания были выбраны следующие вирусы из рода Begomovirus:
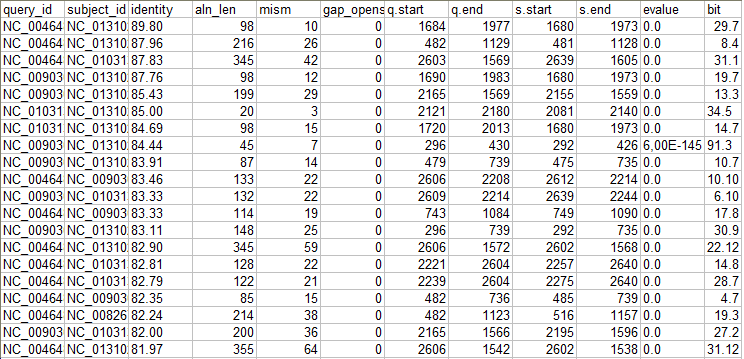 |
Рис5. Таблица результатов, сортированная по identity.
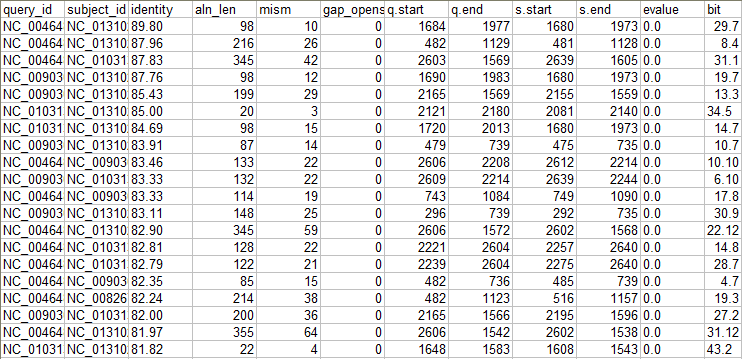 |
Рис6. Таблица результатов, сортированная по evalue.