
Семестр 2, практикум 12
Назад на учебную страницу Птицыной ЕленыBLAST
На этом практикуме мы знакомились с работой сервиса BLAST. Семейство компьютерных программ BLAST, служащих для поиска гомологов белков и нуклеиновых кислот по первичной структуре или её фрагмента, было разработано учёными из системы Национальных институтов здравоохранения США (публикация - 1990 год).
Задание 1
Чтобы найти белки, гомологичные бета-субъединице НАДФ-зависимой трансгидрогеназы из генома бактерии Rhodospirillum rubrum ATCC 11170, был использован сервис BLAST Protein. Кроме алгоритма blastp (поиск аминокислотных последовательностей), который был применен, у BLAST существуют другие алгоритмы: blastn (поиск по нуклеотидным последовательностям), blastx (поиск в аминокислотных базах данных по нуклеотидному запросу), и другие.
Что можно ввести при поиске?
Таблица 1. Описание окошек на странице BLAST| Название | Объяснение | Пример заполнения окошка |
| Enter accession number(s), gi(s), or FASTA sequence(s) | Надо ввести в интерактивном режиме или AC исследуемого белка, или его GI (универсальный идентификатор последовательностей в NCBI), или его последовательность в FASTA-формате. Можно исследовать сразу несколько белков. | ABC22981.1 |
| Or, upload file | Или загрузить файл с аналогичным содержимым | |
| Query subrange (From To) | Можно искать лишь по фрагменту последовательности, заданному координатами. | Оставляем пустыми |
| Align two or more sequences | С помощью алгоритма можно выровнять несколько последовательностей. | Не ставим галочку |
| Database | Можно выбрать базу, по которой будет проводиться поиск. Например, Refseq, SwissProt, PDB. | UniprotKB/Swiss-Prot(swissprot) |
| Organism | Можно ограничить поиск, задав организм, которому будут принадлежать белки. Чтобы добавить несколько организмов, нужно нажать крестик. Чтобы исключить указанный организм, нужно нажать exclude. | Оставляем пустым |
| Exclude: Models (XM/XP) / Non-redundant RefSeq proteins (WP) / Uncultured/environmental sample sequences | Можно исключить модели, основанные на анализе геномной ДНК / неизбыточные последовательности иp RefSeq / образцы, выделенные из окружающей среды и не имеющие четкой таксономической принадлежности (в основном это касается нуклеотидов). | Не ставим галочки |
| Algorithm | Выбор алгоритма | blastp (protein-protein BLAST) |
| Max target sequences | Максимальное количество найденных последовательностей, которое будет выведено. Мы установили 20000, чтобы не упустить ни одного белка. | 20000 |
| Short queries | BLAST может автоматически установить параметры для обработки коротких последовательностей. | Оставляем пустым, т.к. наша последовательность не короткая. |
| Expect threshold | Задание верхнего порога E-value: чем меньше E-value, тем правдоподобнее выравнивание (более статистически значимо). Удобно пользоваться следующим объяснением значения E-value. E-value - это математическое ожидание числа находок BLAST с данным или большим весом в случайном банке того же размера и состава АК (если рассматриваем белки). Поясним примером. Пусть Query – это вход, Shuffled – последовательность, полученная в результате случайной перетасовки букв (это можно реализовать командой Shuffleseq, если бы мы делали это вручную), Subject – находка. Для каждой Query №x построим Shuffled №x и посчитаем число Subjects с весом S’, большим или равным S. Тогда среднее от полученных значений для 1-ой….x-ой…..n-ой Query будет равняться E-value. Таким образом, E-value зависит от: 1) веса выравнивания (↑↓) 2) размера банка (↑↑) 3) длины запроса (↑↑) 4) параметров, используемых для вычисления веса (матрицы и штрафов) (продолжение см.ниже). | 10 |
| Word size | Длина индесированных слов - участков определенной длины, на которые делятся последовательности, а также длина добавленных слов, вес выравнивания которых с оригинальными больше или равен T = 13 (подробнее см.ниже). | 6 |
| Max matches in a query range | Можно ограничить число выравниваний с одним участком белка. Этот параметр может пригодиться, чтобы результат работы алгоритма показал не только сильные совпадения с одной частью белка, но и охватил более слабые сходства по другим участкам. | 0 |
| Matrix | Можно выбрать матрицу сходства, которую будет использовать BLAST для вычисления веса выраванивания. | BLOSUM62 |
| Gap Costs | Штраф за открытие инделя, за каждый следующий символ гэпа | Existence: 11 Extension: 1 |
| Compositional adjustments | Борьба с участками малой сложности, то есть участками, сходство которых будет высоко, но не будет интересно с биологической точки зрения, поскольку будет демонстрировать наличие, например, очень часто встречающихся в белках последовательностей (например, полипролиновые регионы). Эти участки называются участки малой сложности. Корректируется веса в матрице замен, часто с помощью введения определенной константы, что позволяет более точно вычислить E-value. | Conditional compositional score matrix adjustment |
| Filter Low complexity regions | BLAST может замаскировать участки малой сложности. | Не ставим галочку |
| Mask Mask for lookup table only | Также маскировка участков малой сложности, но лишь на первом этапе работы BLAST (при составлении таблицы). | Не ставим галочку |
| Mask Mask lower case letters | Маскировка строчных букв во введенной последовательности. | Не ставим галочку |
Посмотрим,
как работает BLAST?
Пусть у нас есть входная последовательность (Query) QLGVKAGW, заданная пользователем длина слова равна 3.
1) BLAST ищет в Query все слова заданной длины: QLG LGV GVK VKA KAG AGW
2) BLAST добавляет похожие на них слова, вес выравнивания слов которых с оригинальными больше или равен T = 13.
3) BLAST по хэш-таблице устанавливает соответствие: слово – последовательности в базе данных, имеющие точное вхождение данного слова. Сортировка происходит по AC и номеру позиции слова.
4) Для отбора последовательности необходимы два слова на расстоянии меньше или равно A ( A = 20), причем на одной диагонали.
5) Затем выравнивание расширяется направо и налево от обнаруженных «затравок», при этом используется алгоритм динамического программирования (по определению, днамическое программирование в теории управления и теории вычислительных систем — способ решения сложных задач путём разбиения их на более простые подзадачи, термин появился в 1940-е годы в США). Заметим, что сначала выравнивание продолжается без гэпов, а затем полученные выравнивания объединяются с гэпами. Когда расширение прекращается BLAST? Оно прекращается, если падение суммарного веса выравнивания от точки последнего максимума достигнет заранее установленного порога X.
Продолжение объяснения для E-value. Для каждой найденной последовательности необходимо вычислить, насколько она сходна с введенной последовательностью QLGVKAGW, и насколько это сходство статистически значимо. Поскольку при вычислении весов выравниваний последовательностей используются разные матрицы замен и разные штрафы, показатель сходства должен быть преобразован к единому для всех способов виду: B [биты]= (λS – lnK)/ln2, где S – вес выравнивания, K и λ – две константы, зависящие как раз от параметров вычисления веса (матрицы и штрафов). Получается, что B - это как бы нормализованный вес в битах. Сходство ↑↑ число битов. Далее BLAST вычисляет достоверность данного выравнивания: E-value=mn·2^(-B) , где m – длина исходной последовательности, n – размер базы данных. Эти формулы немножко упрощены: они верны, если банк состоит из одной последовательности длины n, a BLAST еще учитывает длины последовательностей банка. Как было указано выше, чем меньше E-value, тем правдоподобнее выравнивание (более статистически значимо).
Примечание. Что такое вес выравнивания и матрица замен, мы изучали на предыдущем занятии. Для подсчета веса выравнивания составляется таблица, у которой по верхней горизонтали, направленной вправо, написаны буквы первой последовательности, по левой вертикали, направленной вниз, написаны буквы второй последовательности, гэпы пропускаются. Потом по матрице прокладывается путь: если одной букве соответствует другая, по ячейке проводется диагональная черта, если в первой последовательности гэп, проводится вертикальная черта вниз, если во второй последовательности гэп, проводится горизонтальная черта направо. Вес выравнивания равен сумме весов замен в ячейках, по которым проведены диагонали. Веса замен извлекаются из матриц весов замен, составленных на основе частотах замен аминокислотных остатков (в случае белков) в блоках гомологичных белков (например, матрицы BLOSUM, PAM).
Итак,
BLAST - сервис, который производит парное сравнение последовательностей и возвращает оценку выравнивания, E-value и собственно выровненные последовательности. Это происходит очень быстро, ведь BLAST сначала отбирает те последовательности и места (номер остатка) в них, с которых имеет смысл начать строить выравнивание. BLAST использует определенный эвристический алгоритм на этапе поиске пар (на следующем этапе уже используется динамическое программирование - исчерпывающие алгоритмы). Гипотетически, может быть входная последовательность и база данных, в которой наилучшее соответствие для входной последовательностей не содержит последовательностей k-длины, общих с последовательностью базы данных. А тогда BLAST не сможет найти это совпадение! Заметим, что если мы найдем подходящее совпадение, то мы можем сказать, что это k-мер идентичных последовательностей в паре. Но это не гарантированно, поэтому BLAST считают эвристическим алгоритмом. Мы будем помнить, что иногда более точные (не эвристические), но не такие быстрые методы более эффективны - это подходы, базирующиеся на глобальном выравнивании, глобальное и локальное выравнивание и даже точечные диаграммы (см. Dot Matrix View в предыдущем практикуме).Скачанные результаты поиска представлены в файле.
На самой странице видна таблица находок с параметрами, представленными в Таблице 2.
Таблица 2.Описание окошек таблицы находок| Description | Max score | Total score | E-value | Query cover | Per.ident | Accession |
| Полное название белка и название организма, из которого исследователи получили белок | Максимальный вес выравнивания участка найденной последовательности и исходной. | Общий вес выравнивания. Он равен максимальному весу выравнивания участка найденной последовательности и исходной, если был лишь один участок сходства, по которому BLAST делал выравнивание. | Математическое ожидание числа находок BLAST с данным или большим весом в случайном банке того же размера и состава АК (если рассматриваем белки). | Доля длины гомологичного участка в найденной последовательности от ее общей длины в процентах. | Доля совпавших аминокислот в процентах. | Идентификатор белковой последовательности в базе данных, которую мы выбрали. |
Графическое изображение результатов BLAST представлено на Рисунке 1.

Рисунок 1. Графическое изображение результатов
Толстой линией сверху изображена query – входная последовательность. Положение находок соответствует их координатам во входной последовательности, цвет – нормализованному весу (bit score). Степень гомологии исследуемых белков высока (красный цвет по полной длине), а три из них укорочены по сравнению с остальными как с N, так и с C-конца.
Так как было найдено всего 6 белков, для выравнивания были оставлены все белки. Последовательности этих белков были скачаны в fasta-формате, с помощью программы JalView было построено выравнивание.Все белки можно признать гомологичными, так как они удовлетворяют указанным критериям. Например, можно выделить участок из 12 аминокислот, полностью идентичных у найденных белков (Рисунок 2).
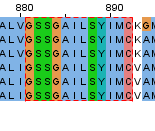
Рисунок 2. Подтверждение гомологичности белков
Задание 2
Карта локального сходства (Dot Matrix View) показывает участки сходства последовательностей на основании работы BLAST. Была построена карта локального сходства для белков A0A067N9N0_PLEOS и A0A061M0C9_9MICO (Риуснок 2). BLAST построил 3 основных линии выравнивания. В основном, это делеции во второй последовательности. Также есть 2 небольших делеции в первой последовательности. Кроме того, скорее всего, есть дупликация и транслокация небольшого кусочка второй последовательности в первой: в первой последовательности эти копии находятся в 40-70 и 160-183 местах. Во второй последовательности тоже есть дупликация и транслокация относительно первой: 235-250 и 385-400.
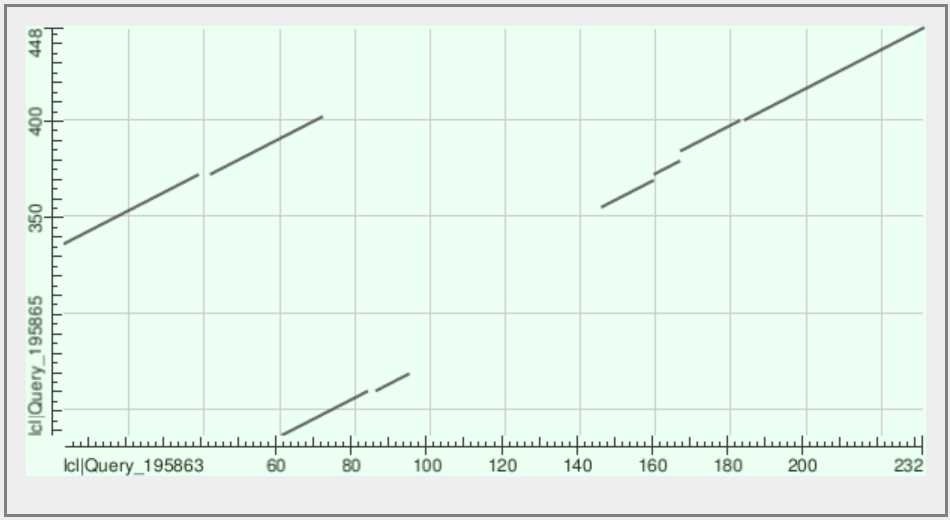
Рисунок 2. Карта локального сходства 2 белков
Задание 3
Игры с последовательностью, которая не кодирует белок
Фрагмент стихотворения А.Блока "На поле Куликовом", переведенный Яндекс-переводчиком на английский язык, лишенный знаков пунктуации, кроме одного восклицательного знака, и пробелов, со всеми прописными буквами был использован для игр с BLAST (AGAINOVERTHEKULIKOVFIELDMISTROSEANDSPREADANDLIKEACLOUDOFHARSHTHECO MINGDAYCLOUDEDOVERFORTHESILENCEOFTHEIMPENETRABLEBEHINDTHESPREADINGM ISTNOTHARTHETHUNDEROFTHEBATTLEWEIRDDONOTSEEMYMOLNIICOMBATBUTRECOGNI ZEYOUTHEBEGINNINGHIGHANDREBELLIOUSDAYS!OVERTHEENEMYCAMPASITHAPPENED ANDTHESPLASHANDTHETRUMPETSOFSWANS).
Изменялись параметры: Word size, Database, Matrix. Скачать: Игры с последовательностью, которая не кодирует белок
Игры с последовательностью нашего белка
Последовательность бета-субъединицы НАДФ-зависимой трансгидрогеназы из генома бактерии Rhodospirillum rubrum ATCC 11170 была использована для игр с BLAST.
Изменялись параметры: Organism, Expect threshold, Gap Costs. Скачать: Игры с последовательностью ABC22981.1
BLAST - удобная программа. Вы можете перейти на страницу сервиса BLAST Protein по ссылке.