Семестр 3, практикум 12
Назад на учебную страницу Птицыной ЕленыСборка de novo.
Команды
| $ cd /nfs/srv/databases/ngs/elena-pt | Переход в рабочую папку |
| wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/000/SRR4240360/SRR4240360.fastq.gz | Скачивание данных |
| $ gunzip SRR4240360.fastq.gz | Распаковка. Итог - 872768784 байт. |
| $ fastqc SRR4240360.fastq | Контроль качества 1 |
| $ cat /P/y18/term3/block3/adapters/*.fa > adapters.fa | Создание файла с адаптерами |
| $ java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR4240360.fastq SRR4240360.trim.fastq ILLUMINACLIP:illumina_adapters.fasta:2:7:7 | Удаление адаптеров. Итог - 868013364 байт. Результат: Input Reads: 8254632 Surviving: 8212774 (99,49%) Dropped: 41858 (0,51%) |
| $ fastqc SRR4240360.trim.fastq | Контроль качества 2 |
| $ java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR4240360.trim.fastq SRR4240360.trim2.fastq TRAILING:20 | Отбор ридов качеством выше 20. Итог - 849067124. Результат: Input Reads: 8212774 Surviving: 8140976 (99,13%) Dropped: 71798 (0,87%). |
| $ fastqc SRR4240360.trim2.fastq | Контроль качества 3 |
| $ java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR4240360.trim2.fastq SRR4240360.trim3.fastq MINLEN:32 | Отбор последовательностей длины более 32. Итог - 834783814. Результат: Input Reads: 8140976 Surviving: 7915474 (97,23%) Dropped: 225502 (2,77%). |
| $ fastqc SRR4240360.trim3.fastq | Контроль качества 4 |
| $ velveth 13_velveth 31 -short -fastq SRR4240360.trim3.fastq | Запуск velveth. Итог - 272. |
| $ velvetg 13_velveth | Запуск velvetg. Результат: Final graph has 596 nodes and n50 of 43070 , max 113474, total 678127, using 0/7915474 reads |
Команда для вывода размера файла в байтах: $ stat file -c %s .
Контроль качества с помощью fastq
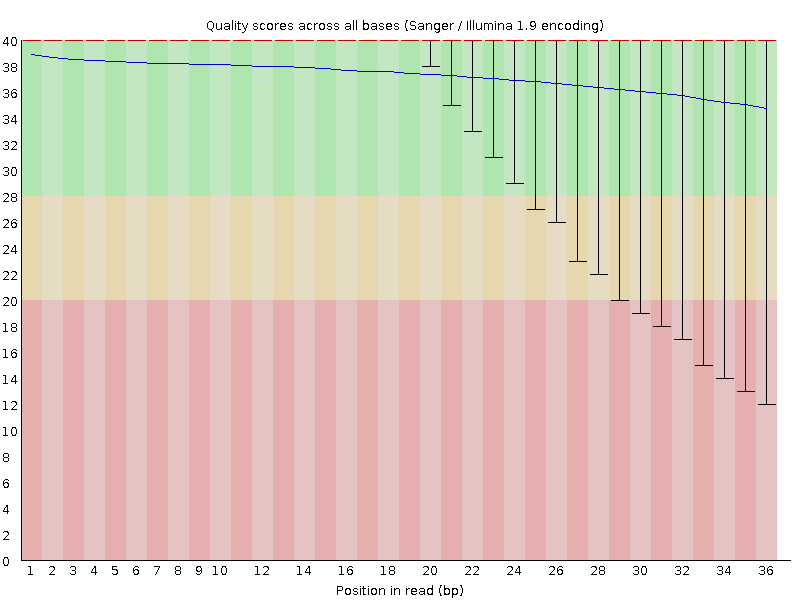
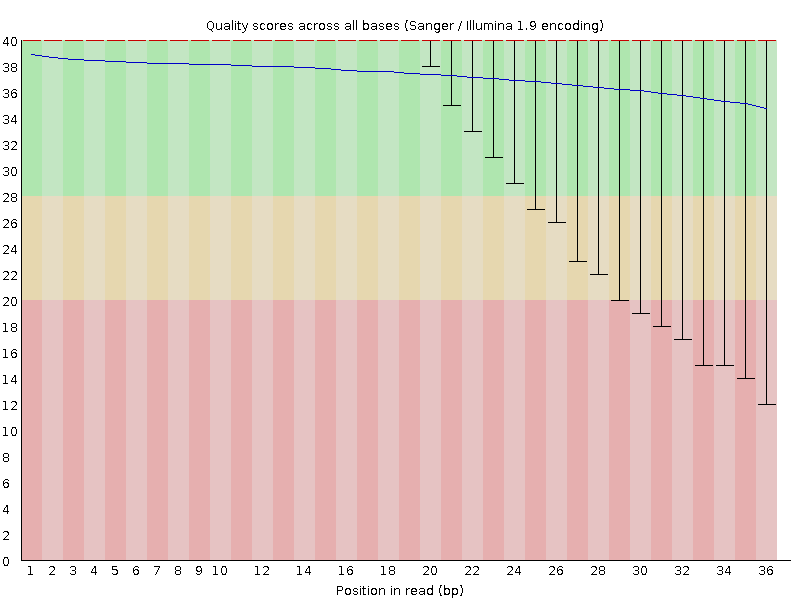
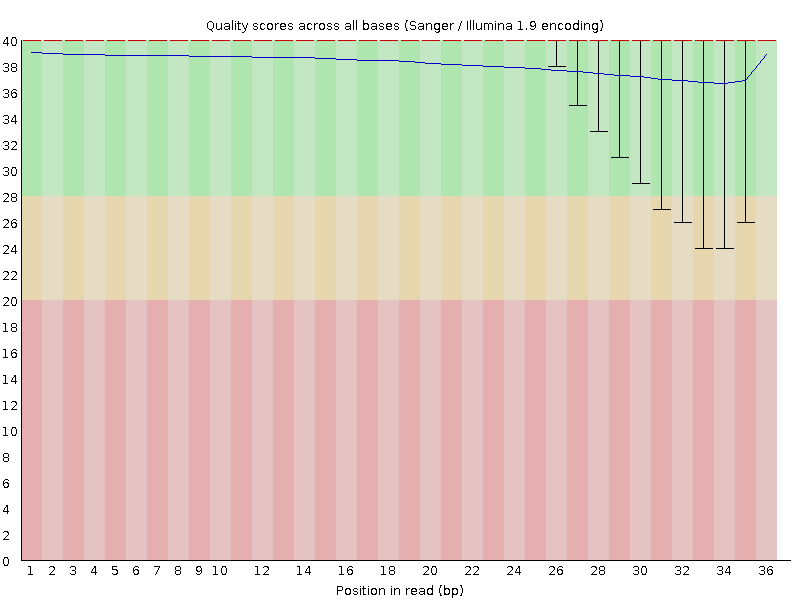
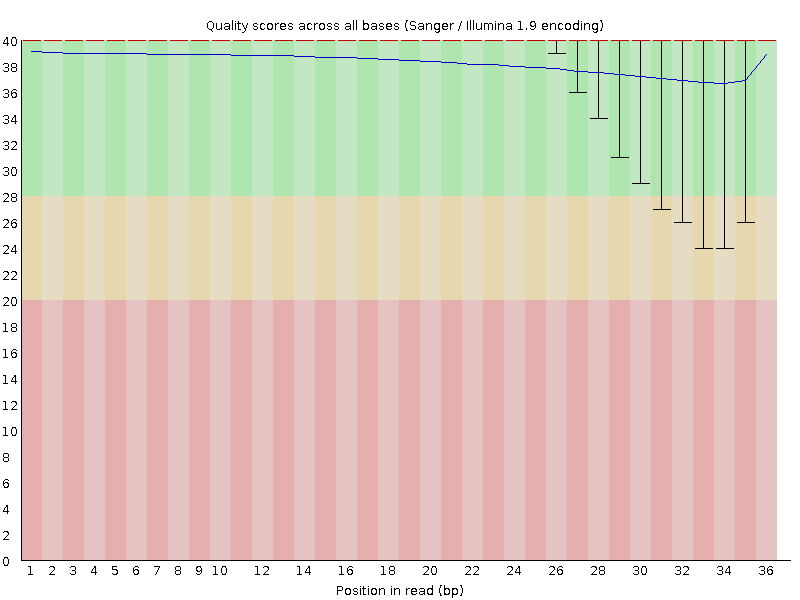
Контиги: общая характеристика
Контиги можно найти в папке 13_velveth в файле contigs.fa. 3 самых длинных контига:
>NODE 1 length 113474 cov 33.525459 >NODE 5 length 83603 cov 33.646065 >NODE 4 length 64155 cov 35.847324
Аномальный контиг с очень маленьким покрытием:
>NODE 422 length 31 cov 1,612903
Аномальные контиги с очень большим покрытием:
>NODE 363 length 40 cov 99,599998 >NODE 40 length 69 cov 109,391304
Выравнивание трех самых длинных контигов с хромосомой Buchnera aphidicola (GenBank/EMBL AC — CP009253)
Buchnera aphidicola - протеобактерия, являющаяся основным симбионтом тли.
Выравнивание трех самых длинных контигов с её хромосомой было проведено с помощью blastn (поставлена галочка "Align two or more sequences", после этого в первое окно введена последовательность контига, во второе - AC хромосомы). Ориентация всегда была plus/plus, что видно по наклону прямой Dot plot и по характеристикам выравниваний (это указано в поле Strand).
Контиг NODE 1.
Лёг на большую часть генома c query cover = 76%, e-value = 0, per.ident = 81,43%, получено выравнивание с 15 участками. Особенно хорошо легла последняя четверть контига.
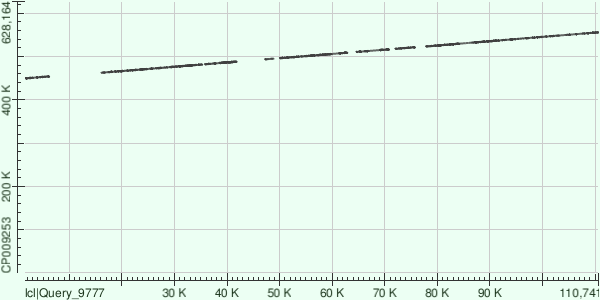
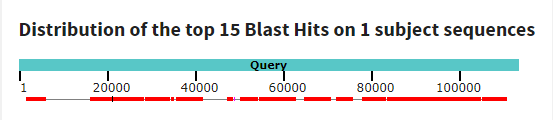
Подробнее о выравниваниях:
Score Expect Identities Gaps Coordinates 17265 bits(9349)0.0 17688/21721(81%)545/21721(2%) 528794 to 550219 4331 bits(2345) 0.0 4573/5655(81%) 127/5655(2%) 550361 to 555905 4047 bits(2191) 0.0 5691/7389(77%) 208/7389(2%) 467412 to 474667 3949 bits(2138) 0.0 6513/8614(76%) 345/8614(4%) 500370 to 508806 3895 bits(2109) 0.0 4894/6238(78%) 194/6238(3%) 510438 to 516539 3029 bits(1640) 0.0 4373/5687(77%) 210/5687(3%) 523105 to 528679 2724 bits(1475) 0.0 3862/5015(77%) 162/5015(3%) 462496 to 467421 2278 bits(1233) 0.0 4621/6238(74%) 308/6238(4%) 481997 to 488106 2237 bits(1211) 0.0 4426/5971(74%) 250/5971(4%) 474844 to 480660 2167 bits(1173) 0.0 3571/4732(75%) 152/4732(3%) 449411 to 454069 2128 bits(1152) 0.0 2922/3782(77%) 99/3782(2%) 517766 to 521500 1914 bits(1036) 0.0 3253/4323(75%) 153/4323(3%) 496111 to 500325 1014 bits(549) 0.0 1108/1384(80%) 13/1384(0%) 493487 to 494864 573 bits(310) 2e-162 564/686(82%) 20/686(2%) 480874 to 481545 145 bits(78) 1e-33 107/120(89%) 5/120(4%) 495033 to 495148
Контиг NODE 5.
Лёг на большую часть генома c query cover = 58%, e-value = 0, per.ident = 74,95%, получено выравнивание с 8 участками.
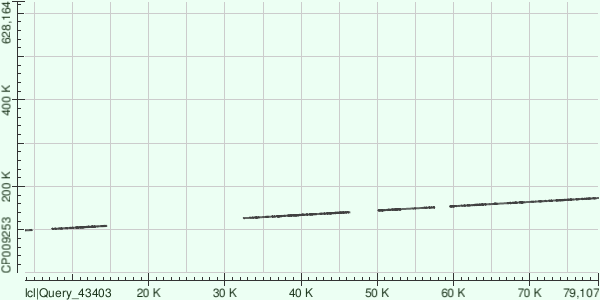

Подробнее о выравниваниях:
Score Expect Identities Gaps Coordinates 5465 bits(2959) 0.0 9751/13010(75%) 548/13010(4%) 127825 to 140555 4796 bits(2597) 0.0 6355/8168(78%) 264/8168(3%) 153752 to 161738 4401 bits(2383) 0.0 5859/7536(78%) 243/7536(3%) 144368 to 151796 3777 bits(2045) 0.0 5567/7274(77%) 215/7274(2%) 101712 to 108876 3415 bits(1849) 0.0 3911/4914(80%) 112/4914(2%) 161898 to 166752 3301 bits(1787) 0.0 4967/6517(76%) 159/6517(2%) 166750 to 173180 1123 bits(608) 0.0 1004/1199(84%) 11/1199(0%) 126623 to 127815 713 bits(386) 0.0 731/901(81%) 9/901(0%) 98408 to 99303
Контиг NODE 4.
Лёг на большую часть генома c query cover = 70%, e-value = 0, per.ident = 78,38%, получено выравнивание с 12 участками.
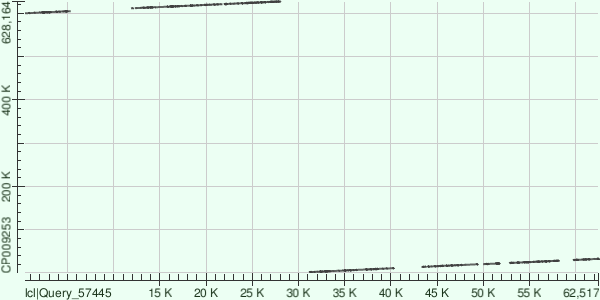

Подробнее о выравниваниях:
Score Expect Identities Gaps Coordinates 5749 bits(3113) 0.0 7229/9223(78%) 256/9223(2%) 2004 to 11103 4959 bits(2685) 0.0 5845/7379(79%) 184/7379(2%) 613658 to 620926 3068 bits(1661) 0.0 3946/5046(78%) 170/5046(3%) 599832 to 604795 2889 bits(1564) 0.0 4678/6173(76%) 248/6173(4%) 621055 to 627104 2772 bits(1501) 0.0 4159/5433(77%) 219/5433(4%) 23067 to 28363 2270 bits(1229) 0.0 1902/2231(85%) 30/2231(1%) 17962 to 20182 1583 bits(857) 0.0 2451/3226(76%) 88/3226(2%) 14727 to 17919 1578 bits(854) 0.0 2150/2777(77%) 84/2777(3%) 30013 to 32745 1476 bits(799) 0.0 1509/1851(82%) 51/1851(2%) 20358 to 22183 1238 bits(670) 0.0 1625/2086(78%) 66/2086(3%) 611633 to 613671 403 bits(218) 1e-111 393/478(82%) 9/478(1%) 13994 to 14465 209 bits(113) 3e-53 236/297(79%) 2/297(0%) 611229 to 611524