Семестр 3, практикум 3
Назад на учебную страницу Птицыной ЕленыКомплексы ДНК-белок
Задание 1. Предсказание вторичной структуры заданной тРНК
Упражнение 1.
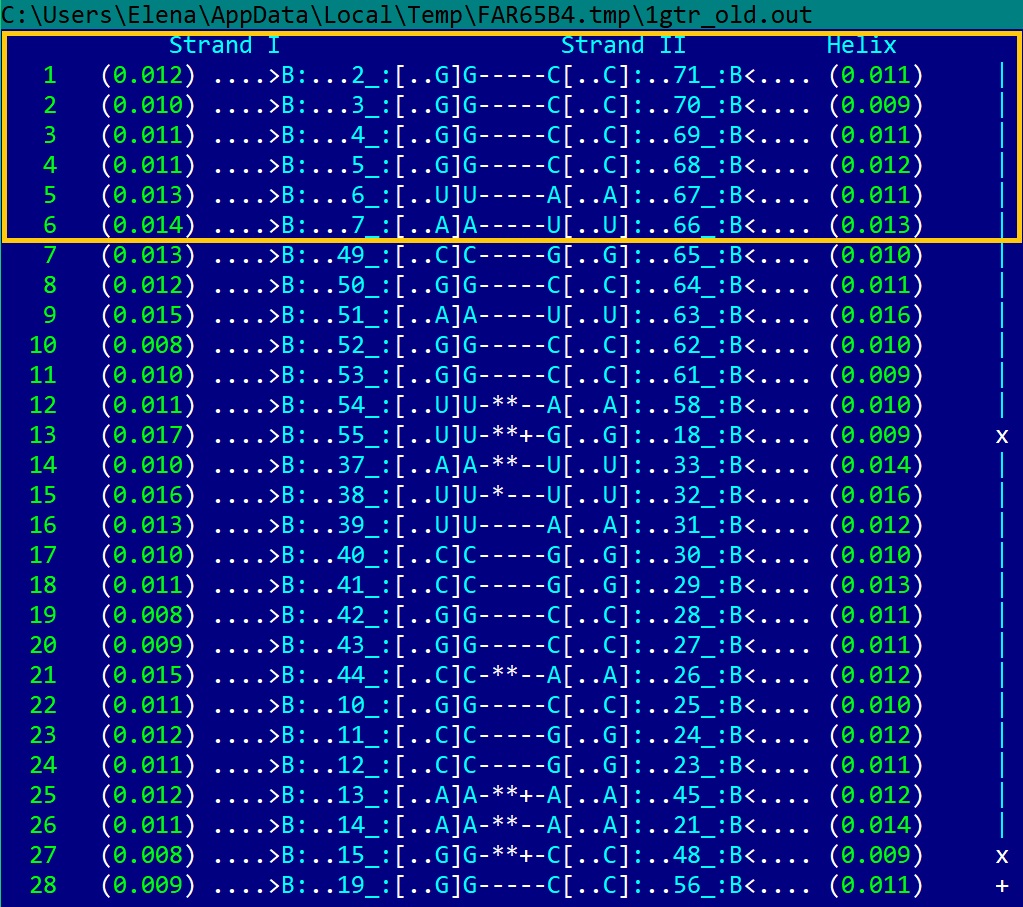
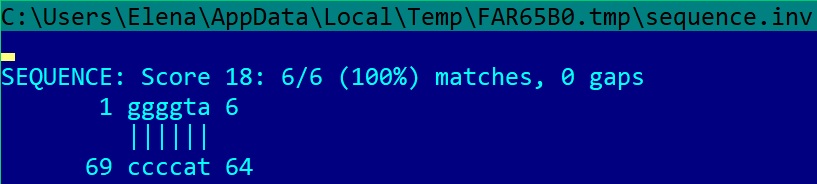
С помощью пакета EMBOSS, работающего под операционной системой Linux, мы проанализировали вторичную структуру 1gtrRNA на наличие инвертированных повторов. Для этого из PDB был скачан файл в fasta-формате, вырезана часть с нуклеотидами, перенесена в новый файл 1GTR.fasta. Далее выполнена команда einverted 1GTR.fasta -threshold 1 (мы перебрали несколько значений threshold) Выходной файл: sequence.inv.
| find_pair | einverted |
|
|
Видно, что команда einverted нашла меньше пар, чем find_pair. Нумерация оснований сдвинута.
Упражнение 2.
Далее была использована web-версия программы RNAfold
. Удачной является структура, полученная в первый раз (Рис.1):
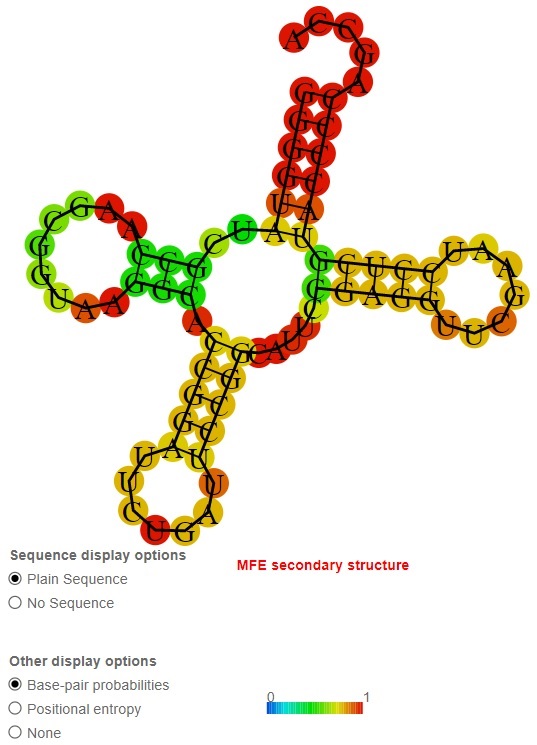
Далее было проведено сравнение результатов работы команд из предыдущего и из этого практикумов (Табл. 2).
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'-2-7-3' 5'-66-71-3' всего 6 пар |
Предсказано 6 пар из реальных 6 | Предсказано 6 пар из реальных 6 |
| D-стебель | 5'-10-12-3' 5'-23-25-3' всего 3 пары |
Предсказано 0 пар из реальных 3 | Предсказано 3 пары из реальных 3 |
| T-стебель | 5'-49-53-3' 5'-61-65-3' всего 5 пар |
Предсказано 0 пар из реальных 5 | Предсказано 5 пар из реальных 5 |
| Антикодоновый стебель | 5'-37-44-3' 5'-26-33-3' всего 8 пар |
Предсказано 0 пар из реальных 8 | Предсказано 5 пар из реальных 8 |
| Общее число канонических пар нуклеотидов | 22 | 6 | 19 |
Примечание. Результат работы не web-версии RNAfold (команды export PATH=${PATH}:/home/preps/golovin/progs/bin, далее cat my.fasta | RNAfold --MEA представлен ниже (Рис.2). Обозначения: точки - нуклеотиды, не образующие водородные связи, круглые скобки - нуклеотиды, образующие водородные связи, квадратные и фигурные скобки - взаимодействия, образующие псевдоузлы.
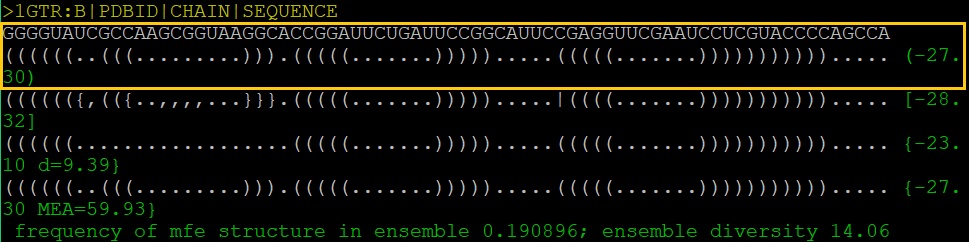
Задание 2. Поиск ДНК-белковых контактов в заданной структуре
Упражнение 1.
Далее был написан скрипт для Jmol, в котором последовательно проходят изображения всего белкового комплекса 1rh6, только ДНК комплекса, затем красным выделяется множество атомов кислорода 2'-дезоксирибозы, желтым - множество атомов кислорода в остатке фосфорной кислоты, синим - множество атомов азота в азотистых основаниях.
СкриптУпражнение 2.
Будем считать полярными атомы кислорода и азота, а неполярными - атомы углерода, фосфора и серы. Назовем полярным контактом ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5Å. Аналогично, неполярным контактом будем считать пару неполярных атомов на расстоянии меньше 4.5Å. Опишем ДНК-белковые контакты в 1rh6 учитывая лишь цепь B белка. Мы выполнили скрипт (последовательность действий в Jmol), определяя количество связей как наименьшее количество выделенных атомов на каждом шаге (Табл. 3).
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 4 | 19 | 23 |
| остатками фосфорной кислоты | 6 | 4 | 10 |
| остатками азотистых оснований со стороны большой бороздки | 3 | 3 | 6 |
| остатками азотистых оснований со стороны малой бороздки | 1 | 0 | 1 |
Видно, что цепь белка удерживается в основном неполярными связями с сахарофосфатным остовом, а его взаимодействие с атомами малой бороздки практически сводится к нулю.
Скрипт с визуализацией атомов.
Упражнение 3.
С помощью команд remediator --old ''1rh6.pdb'' > ''1rh6_old.pdb'' (для перевода pdb-файла в старый формат) и nucplot 1rh6_old.pdb было получен файл nucplot.ps.
Затем была скачана программа GSview, в ней открыт файл nucpol.ps. Сохраненная в jpg-формате популярная схема ДНК-белковых контактов
представлена ниже (Рис. 3, 4).
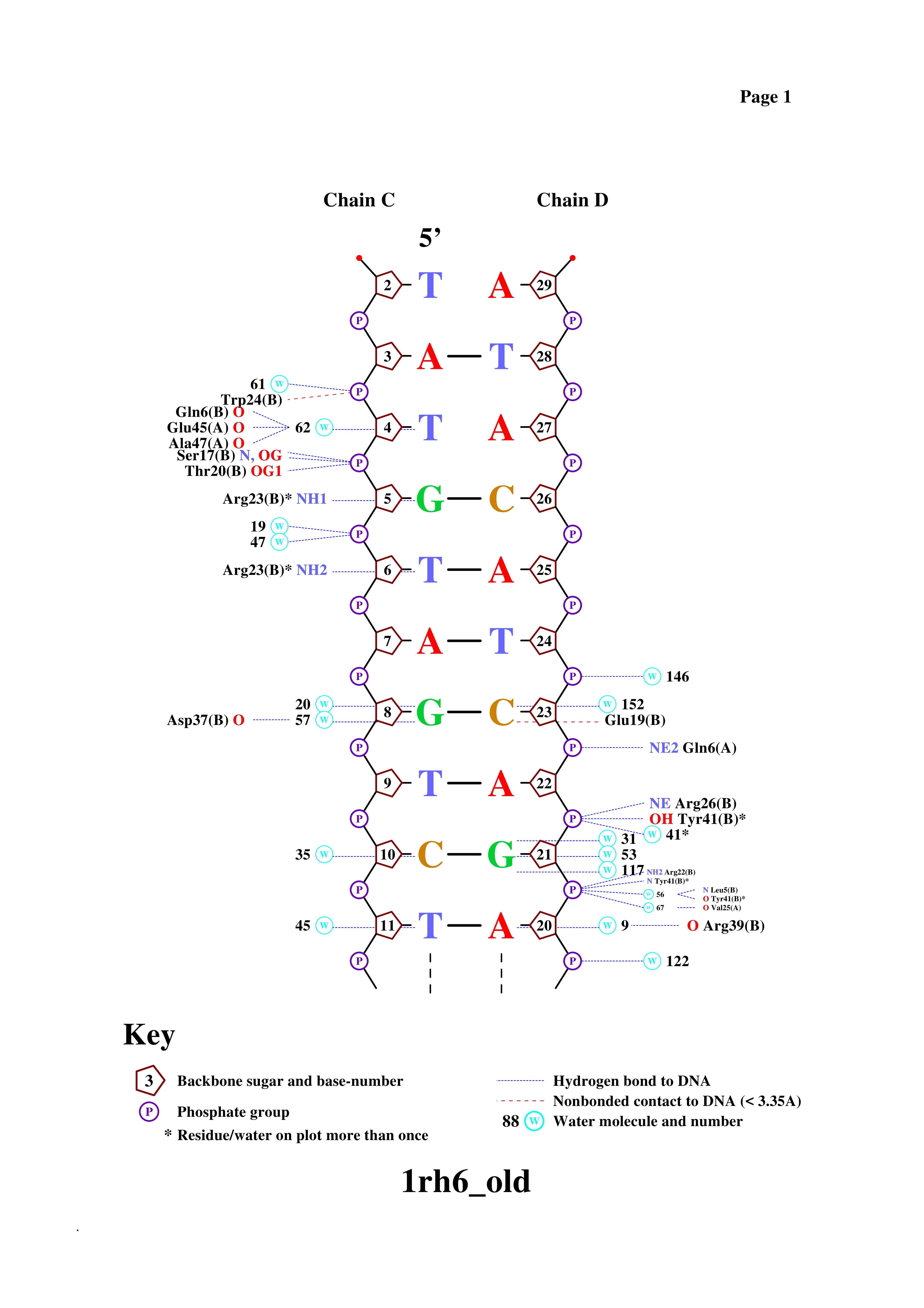
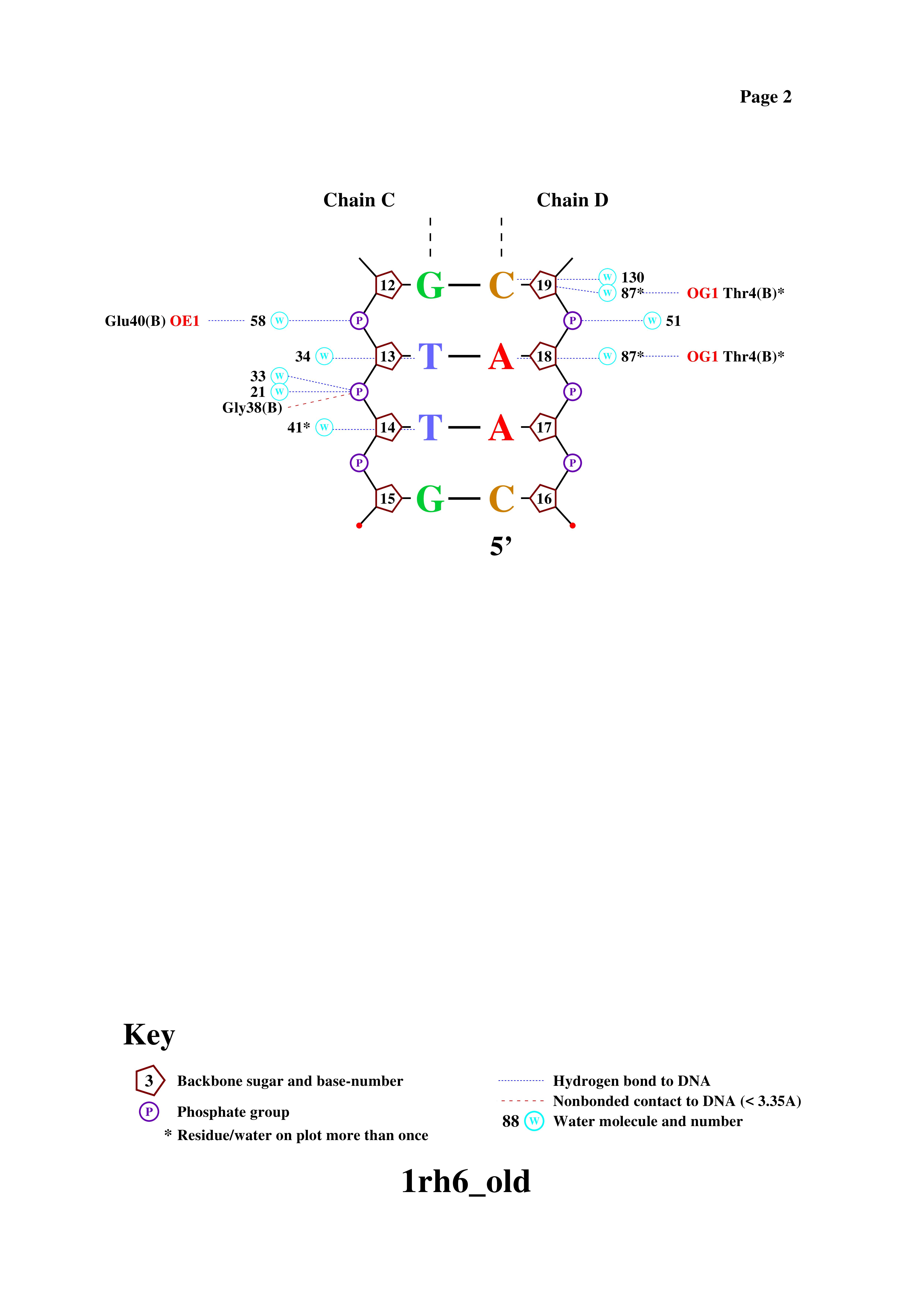
Чтобы понять, какие аминокислотные остатки образуют больше всего связей, мы перенесли информацию из файла 1rh6_old.bond (один из файлов, полученных по команде nucplot) в Excel: см. файл с расчётом. Получилось, что наибольшее количество связей (2) образуют остатки Ser17, Arg23, Tyr41. Их можно найти и на рисунках выше.
Упражнение 4.
Наиболее важными для распознавания последовательности ДНК, наверное, являются остатки,
удоволетворящие критериям:
1) максимальное число связей -> Ser17, Arg23, Tyr41
2) связи с нуклеотидами, а не с сахарофосфатным остовом -> Ser17 и Tyr41 образуют связи с атомами кислорода остатков фосфорной кислоты, то есть с сахарофосфатным остовом,
а Arg23 - c атомами N7 и O4, относящимся к 2 нуклеотидам -> Arg23 самый важный для распознавания.
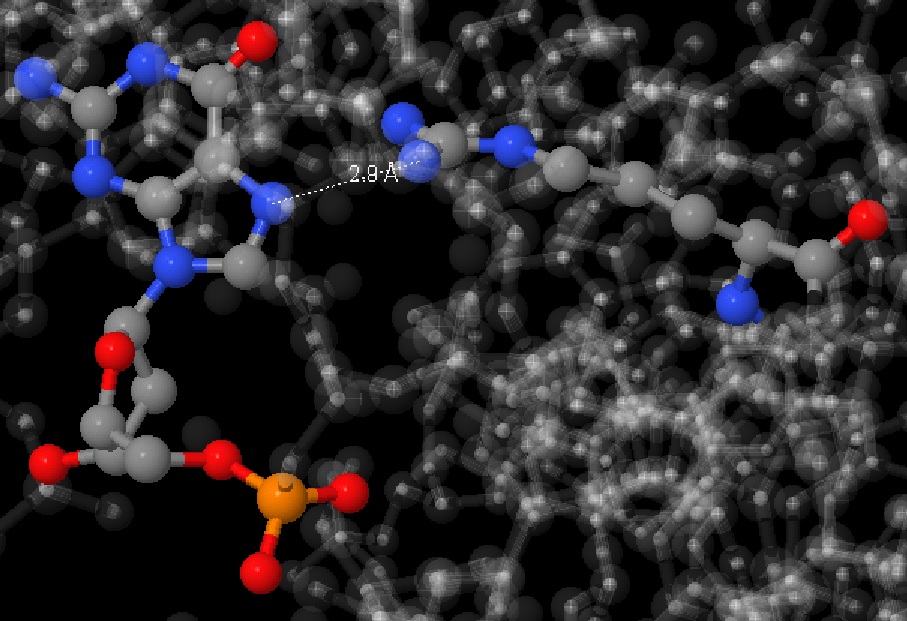
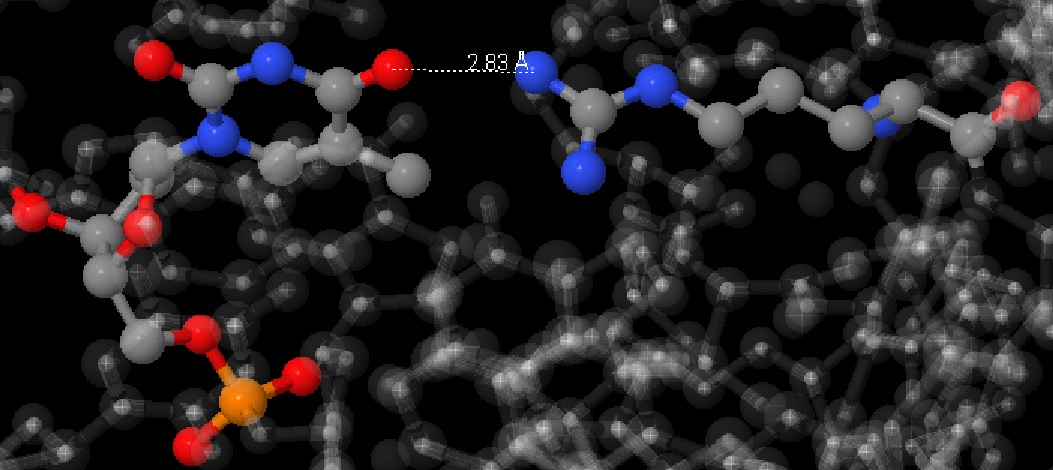
На рисунке 5 представлены взаимодействия [ARG]23:B.NH1 - [DG]5:C.N7 (Рис. 5), на рисунке 6 [DT]6:C.O4 - [ARG]23:B.NH2 (Рис. 6).