Семестр 3, практикум 11
Назад на учебную страницу Птицыной ЕленыРесеквенирование. Поиск полиморфизмов у человека.
Задача этого практикума - найти и описать полиморфизмы у пациента (в хромосоме 16 в нашем случае).
Команды
Исходное количество чтений3965 (см. вывод Trimmomatic: input reads: 3965 surviving: 3798 (95,79%) dropped: 167 (4,21%)) До и после очисткиПрограмма trimmomatic отбросила 167 (4,21%) последовательностей. Графики качества чтения оснований были сгненерированы fastqc (рис.1) На этих графиках красной линий обозначается медиана, желтый блок означает межквартильный диапазон, верхние и нижние усы - 10 и 90%, синяя линия - среднее значение. По оси y отложены quality scores. До очистки качество чтения было хорошее, но наблюдалось и выпадение в область плохих quality scores. После очистки ни один нуклеотид не имеет плохого качества прочтения, значит, очистка была оправдана. 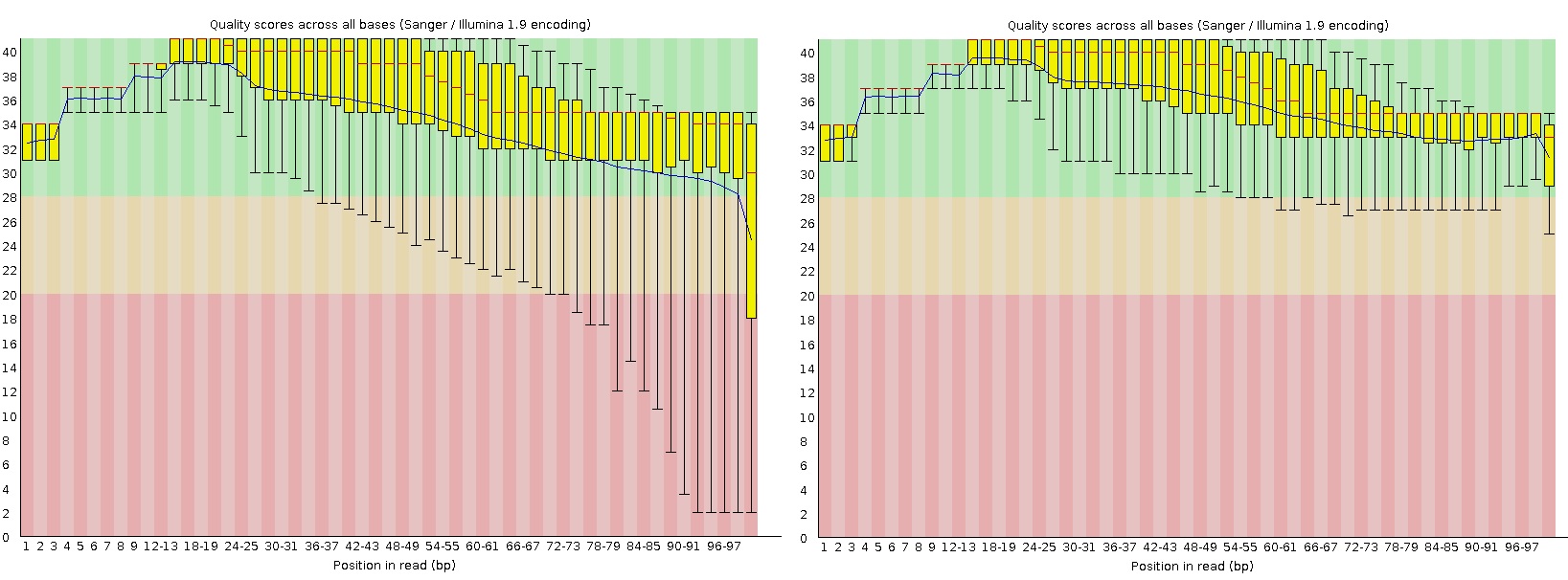
К сожалению, очистка trimmomatic не позволяет исправить gc content. Распределение в обычной случайной библиотеке должно быть нормальным, в нашем случае это не совсем так, много острых пиков. Это может свидетельствовать о загрязнении библиотеки. 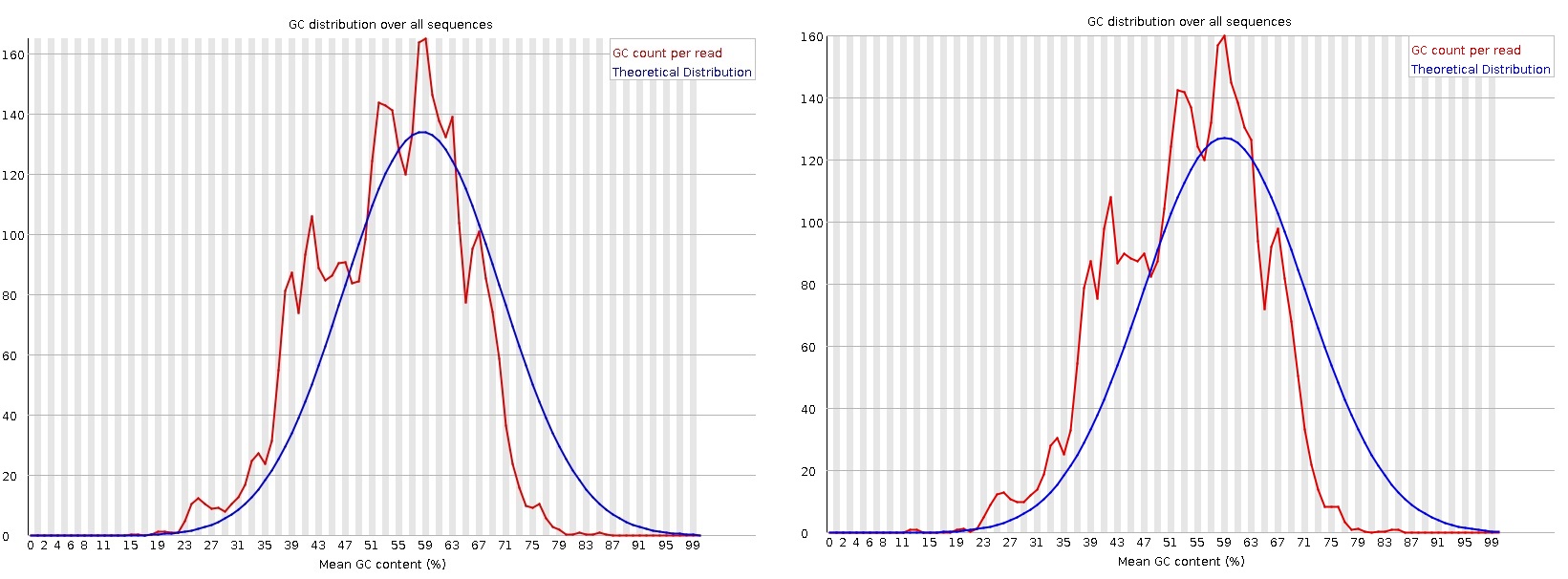
Картирование3763 последовательностей aligned exactly 1 time. Можно сказать, что качество хорошее, так как это составляет 99.08% от всех последовательностей. Кроме того, только 3 последовательности были выровнены более 1 раза. Cм. вывод hisat2-build: 3798 reads; of these: 3798 (100.00%) were unpaired; of these: 32 (0.84%) aligned 0 times 3763 (99.08%) aligned exactly 1 time 3 (0.08%) aligned >1 times 99.16% overall alignment rate Описание полиморфизмов из .vcfВсего в хромосоме 16 пациента 65 полиморфизмов, из них 64 замены и 1 вставка, делеций не было. Примеры. В первой колонке таблицы ниже указано название хромосомы, далее позиция полиморфизма, его суть, качество чтения и глубина покрытия. chr16 11348273 T A 4.12853 DP=10 chr16 11361895 C G 225.009 DP=50 chr16 11444454 gaaaaaaaaaaa gaaaaaaaaaa,gaaaaaaaaa 9.03477 DP=55 Для качества чтений медиана 19,8099, среднее арифметическое 75,826144, для глубины покрытия медиана 3,00, среднее арифметическое 17,56. Категории snp по база данных refseq в annovarЭту информацию можно получить из файла ann_chr16_refgene.variant_function: downstream (3), exonic (9), intergenic (17), intronic (24), ncRNA_intronic (1), upstream (5), UTR3 (3), UTR5 (3). Гены с snp, нуклеотидные и аминокислотные заменыЭту информацию можно получить из того же файла. Интересно посмотреть на уточнённый файл, охватывающий экзонные мутации - ann_chr16_refgene.exonic_variant_function: TNR2, PRM3, PRM2, PRM1, RMI2, PRSS53, HERPUD1. В ann_chr16_refgene.exonic_variant_function можно увидеть обозначение мутации в нуклеотидах и в аминокислотах (например, R131W означает замену 131-ого аргинина на триптофан. nonsynonymous SNV TNP2:NM_005425:exon1:c.C391T:p.R131W, G A stopgain PRM3:NM_021247:exon1:c.C310T:p.R104X, G A nonsynonymous SNV PRM3:NM_021247:exon1:c.G299A:p.R100Q, C T nonsynonymous SNV PRM2:NM_001286359:exon1:c.C290T:p.P97L, G A nonsynonymous SNV PRM2:NM_001286359:exon1:c.C281T:p.A94V, G A synonymous SNV PRM1:NM_002761:exon2:c.C139A:p.R47R, G T synonymous SNV RMI2:NM_152308:exon2:c.A369C:p.T123T, A C nonsynonymous SNV PRSS53:NM_001039503:exon8:c.C1216G:p.P406A, G C nonsynonymous SNV HERPUD1:NM_001010989:exon2:c.G149A:p.R50H, HERPUD1:NM_001272103:exon2:c.G149A:p.R50H, HERPUD1:NM_014685:exon2:c.G149A:p.R50H, G A snp c rsЭту информацию можно получить из файла ann_chr16_dbsnp.hg19_snp138_dropped: 58 snp. Средняя частота snpЭту информацию можно получить из файла ann_chr16_1000g.hg19_ALL.sites.2014_10_dropped: медиана 0,4715455, среднее арифметическое 0,464222799 . Клиническая аннотация snpЭту информацию можно получить из ann_chr16_gwas.hg19_gwasCatalog: 3 SNP имеют клиническое значение. Они связаны с Obesity-related traits (экзонная синонимичная мутация гена PRM1), Parkinson's disease (интронная мутация PRSS53), Metabolic syndrome chr16 (экзонная несинонимичная мутация гена HERPUD1). Аннотация clinvarЭту информацию можно получить из ann_chr16_clinvar.hg19_clinvar_20150629_dropped. Ни один из изучаемых SNP не аннотирован. |