Семестр 3, практикум 6
Назад на учебную страницу Птицыной ЕленыСеквенирование по Сэнгеру
В этом практикуме мы анализировали хроматограммы, полученные при секвенировании по Сэнгеру.
Задание 1.
Исходные файлы: 10_F.ab1, 10_R.ab1,
Хроматограммы были открыты в программе Chromas (Lite).
Нечитаемые 5' и 3' участки. Программа выделила жёлтым начальные и концевые участки, являющиеся, по её мнению, нечитаемыми. Мы немного поправили решение программы. Если мы хотим поправить выделение на левом конце хроматограммы, надо поставить галочку Options -> Continuous Edit, потом зажать клавишу Shift и правой кнопкой мыши менять выделение, щёлкая по нуклеотидам. Если мы хотим поправить выделение на правом конце, надо перевернуть хроматограмму с помощью Reverse и выполнить те же действия. Дальше вернуть c помощью Reverse в исходное положение. Номера нуклеотидов, соответствующих нечитамым 5' и 3' концам, представлены в Таблице 1 (табл. 1). Нумерация нуклеотидов для обратной цепи дана такой же, какой она получается при загрузке изначального файла 10_R.ab1 в программу.
Таблица 1. Нечитаемые участки. Без скобок дано решение программы, в скобках - скорректированное решение.| 5’ | 3’ | |
| Прямая | 1-163 (1-158) | 677-717 (676-717) |
| Обратная | 1-279 (1-248) | нет (1-39) |
Удалены были 5' и 3' нечитаемые концы уже после сравнения хроматограмм, поэтому нумерация нуклеотидов на прямой и обратных последовательностях в примерах ниже дана такая же, как в необрезанных файлах.
Сравнение хроматограмм. Далее обратная последовательность была переведена в комплементарную (Edit > Reverse+Complement), затем с помощью поиска (Find) подслов проведено ручное выравнивание хроматограмм в одинаковом масштабе. Далее были рассмотрены проблемные места. Примеры проблемных мест приведены ниже, исправления сделаны заменой прописных букв на строчные.
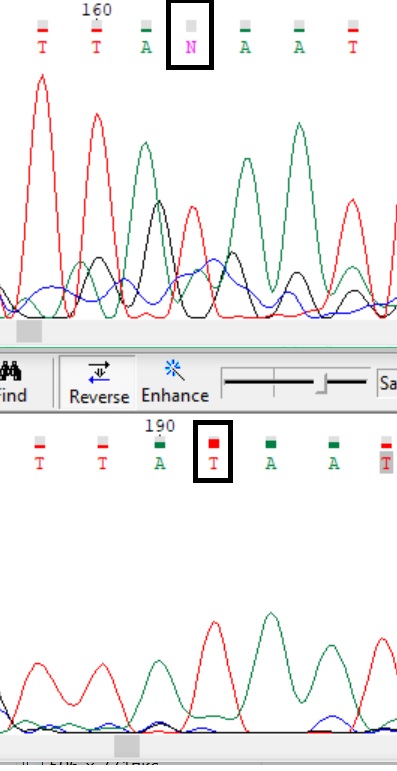
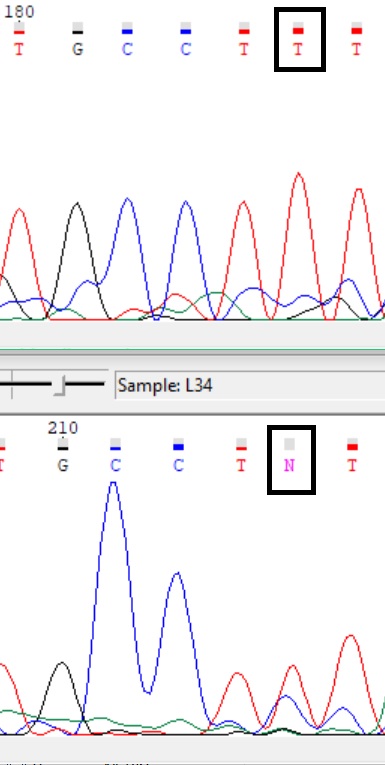
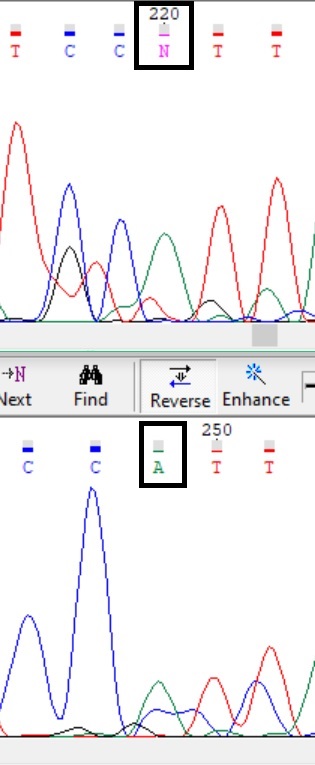
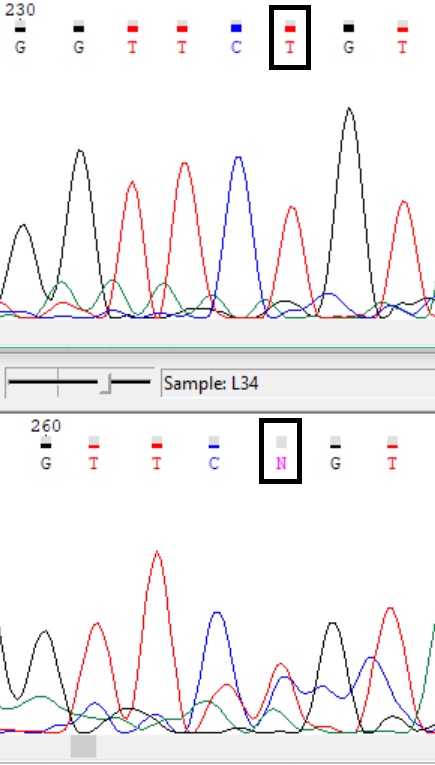
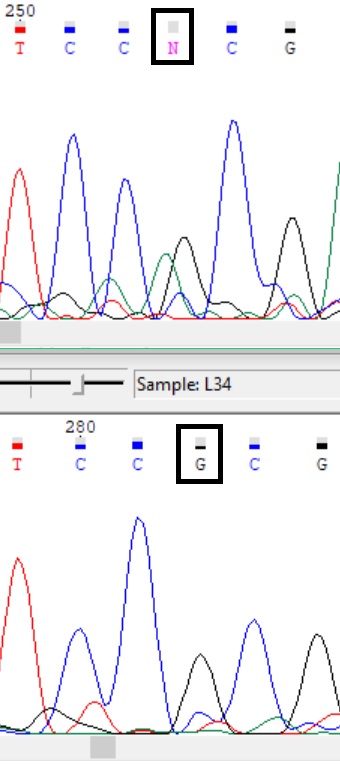
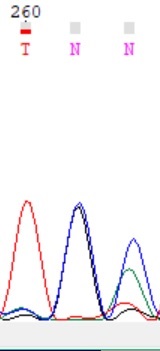
Иногда по хроматограмме одной цепи можно сделать выводы о замене N на значащую букву, поскольку пики интерпретируются при визуальном просмотре:
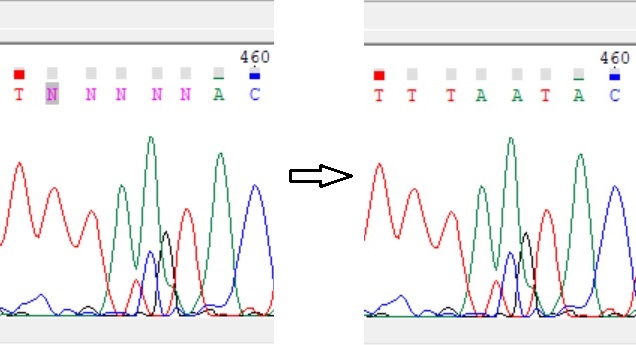
Были и другие исправления. К сожалению, в консенсунсной последовательности остались 2 N - эти нуклеотиды никак нельзя было определить.
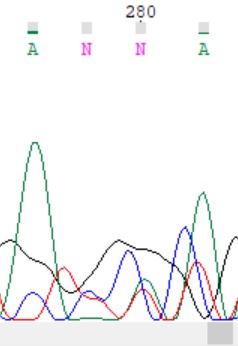
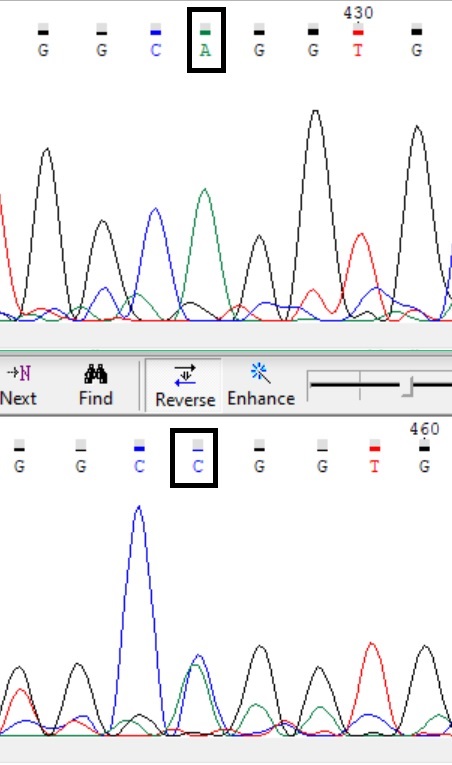
Кроме того, нашёлся полиморфизм (рис. 6).
Дальше были удалены нечитаемые 5' и 3' участки, полученные файлы сохранены в формате ab1: 10_F_red.ab1, 10_R_red.ab1, и fasta: 10_F_red.fasta, 10_R_red.fasta.
Bыравнивание полученных последовательностей. Все строчные буквы, которыми мы заменили проблемные нуклеотиды, перевели в прописные, потому что в противном случае не работала команда needle, сохранили изменения в файлах: 10_F_red_bez.fasta, 10_R_red_bez.fasta. Далее с помощью команды needle 10_F_red_bez.fasta 10_R_red_bez.fasta -aformat3 fasta -out align.fasta получили оптимальное полное выравнивание align.fasta.
Потом выравнивание было открыто в Jalview. Из align.jvp мы скопировали консенсунсную последовательность cons.fasta и посмотрели, что по ней находит blastn. Оказалось, что больше всего это похоже на Modiolus modiolus voucher MT04964 cytochrome oxidase subunit 1 (COI) gene, partial cds; mitochondrial из двухстворчатого моллюска.

Примечание. Таким же образом полученное для исходных (необрезанных и неисправленных хроматограмм, переведённых в fasta) файлов выравнивание доступно здесь (на него, открытого в Jalview, можно подсматривать при сравнении хроматограмм, чтобы ничего не пропустить).
Задание 2
Примеры нечитаемых фрагментов хроматограмм из файлов kamp3_18SIII_F_F03_WSBS-Seq-1-08-15.ab1 WSWS2950_H3_F_G09_2013-06-11-22-39-58 приведены ниже (рис. 8, 9 соответственно).