Семестр 3, практикум 12
Назад на учебную страницу Птицыной ЕленыАнализ транскриптомов.
Задача этого практикума - картировать чтения, полученные в результате секвенирования транскриптома.
Команды
| $ cp /nfs/srv/databases/ngs/Human/rnaseq_reads/chr16.2.fastq /nfs/srv/databases/ngs/elena-pt/pr12/ | Копирование данных |
| $ cd /nfs/srv/databases/ngs/elena-pt/pr12 | Переход в рабочую папку |
| $ fastqc chr16.2.fastq | Анализ качества |
| $ java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr16.2.fastq cleaned.fastq TRAILING:20 | Отбор ридов качеством выше 20. Результат: Input Reads: 9517 Surviving: 9517 (100,00%) Dropped: 0 (0,00%) |
| $ java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 cleaned.fastq cleaned.45.fastq MINLEN:45 | Отбор последовательностей длины более 45. Результат: Input Reads: 9517 Surviving: 9512 (99,95%) Dropped: 5 (0,05%) |
| $ fastqc cleaned.45.fastq | Анализ качества после триммирования |
| $ hisat2-build ../chr16.fasta indexed | Индексирование файла, скачанного в практикуме 11 |
| $ hisat2 -x indexed -U cleaned.45.fastq -S samchr16.2.sam --no-softclip | Картирование чтений на проиндексированную рефернсную последовательность без параметра --no-spliced-alignment (так как мРНК уже созрели и в них нет интронов, а выравниваем на ДНК-последовательность с интронами). Результат: 9512 reads; of these: 9512 (100.00%) were unpaired; of these: 152 (1.60%) aligned 0 times, 9360 (98.40%) aligned exactly 1 time, 0 (0.00%) aligned >1 times, 98.40% overall alignment rate. |
| $ samtools view samchr16.2.sam -b >> bam.bam | Перевод из sam в bam (бинарный формат) |
| $ samtools sort bam.bam sortedbam | Сортировка выравнивания по координате в референсе |
| $ samtools index sortedbam.bam | Индексирование отсортированного файла |
| $ htseq-count -i gene_id -s no -m union -f bam sortedbam.bam /nfs/srv/databases/ngs/Human/rnaseq_reads/gencode.v19.chr_patch_hapl_scaff.annotation.gtf -o count.sam >> count.txt | Подсчёт ридов с параметрами: -i (задание атрибута gff, который будет использоваться как feature ID), -s (yes или no – надо учитывать 1 или 2 направления считывания), -m (режим обработки ридов, входящих в несколько features – см. иллюстрацию работы режимов), -f (указывает на формат входного файла bam/sam).Результат: Идентификаторы генов с числами (в основном 0) и __no_feature 3834__ambiguous 0 __too_low_aQual 0 __not_aligned 152 __alignment_not_unique 0 |
Исходное количество чтений
9517 (см. вывод Trimmomatic: после отбора по качеству Input Reads: 9517 Surviving: 9517 (100,00%) Dropped: 0 (0,00%), после отбора по длине Input Reads: 9517 Surviving: 9512 (99,95%) Dropped: 5 (0,05%))
До и после очистки
Программа trimmomatic отбросила 5 (0,05%) последовательностей.
Графики качества чтения оснований были сгненерированы fastqc (рис.1) На этих графиках красной линий обозначается медиана, желтый блок означает межквартильный диапазон, верхние и нижние усы - 10 и 90%, синяя линия - среднее значение. По оси y отложены quality scores. До очистки качество чтения было и так хорошее, график совсем не изменился, ведь trimmomatic отбросила 0 последовательностей при отборе по длине.
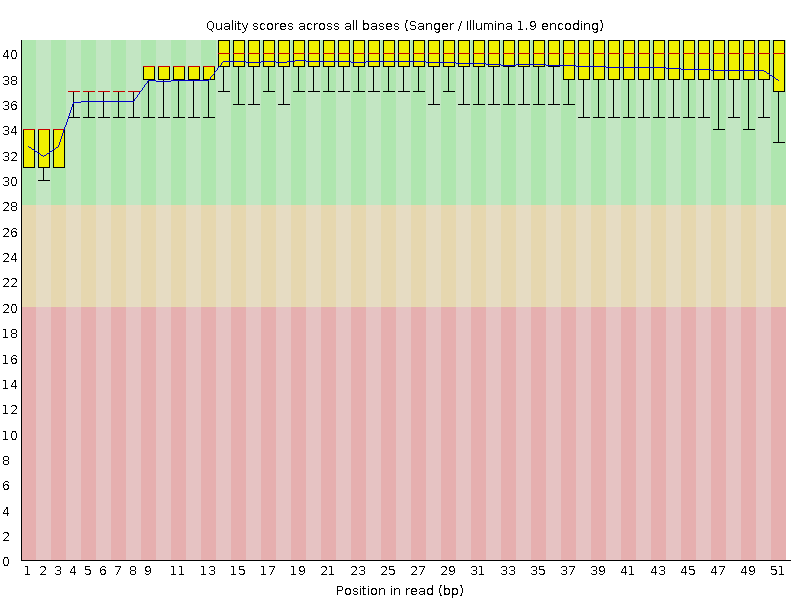
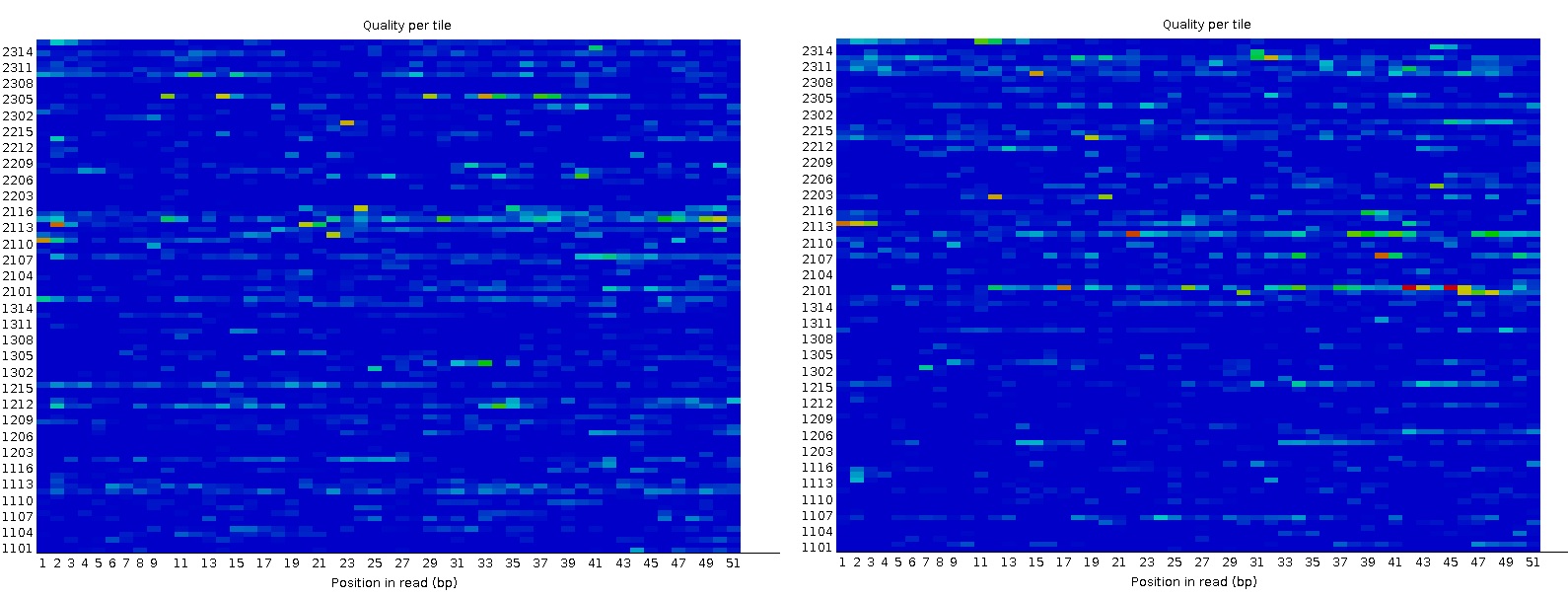
К сожалению, очистка trimmomatic привела к ухудшению проблемам с Per tile sequence quality (рис.2), поэтому, возможно, эту операцию не стоило проводить.
Картирование
9360 последовательностей aligned exactly 1 time. Можно сказать, что качество хорошее, так как это составляет 98.40% от всех последовательностей. Кроме того, 0 последовательностей было выровнено более 1 раза. Cм. вывод hisat2-build:
9512 reads; of these: 9512 (100.00%) were unpaired; of these: 152 (1.60%) aligned 0 times 9360 (98.40%) aligned exactly 1 time 0 (0.00%) aligned >1 times 98.40% overall alignment rate
Подсчёт чтений
Программа htseq-count вывела список Ensembl идентификаторов генов с соответствующими количествами чтений и показала, что:
__no_feature 3834 __ambiguous 0 __too_low_aQual 0 __not_aligned 152 __alignment_not_unique 0
Всего 5526 чтений легло на ENSG00000166501.8. ENSG00000166501 кодирует protein kinase C beta. Протеинкиназы C - семейство протеинкиназ, активируемых кальцием и вторичным мессенджером диацилглицеролом и вовлеченных во множество сигнальных путей в клетке. Эта киназа известна способностью к активации B-клеток, индукции апоптоза и пролиферации клеток эндотелия.