Семестр 4, практикум
Назад на учебную страницу Птицыной ЕленыПрактикум 9. Домены и профили.
Доменная архитектура
Доменная архитектура HSDR_N, SWI2_SNF2 (Рис.1) состоит из 2 доменов PF04313 (HSDR_N) и PF18766 (SWI2_SNF2). Она есть у 2057 последовательностей ("There are 2057 sequences with the following architecture").
Первый домен PF04313 (Type I restriction enzyme R protein N terminus), число последовательностей среди бактерий 5721.
Второй домен PF18766 (SWI2/SNF2 ATPase), число последовательностей среди бактерий 4913.
 Рисунок 1.
Доменная архитектура HSDR_N, SWI2_SNF2
Рисунок 1.
Доменная архитектура HSDR_N, SWI2_SNF2
Бактериальные белки с этой доменной архитектурой
Поиск по базе Uniprot был выполнен командой database:(type:pfam pf04313) database:(type:pfam pf18766) taxonomy:"Bacteria [2]" . Найдено 20674 последовательности.
Скачать файл c результатом поиска Далее найденные последовательности были сортированы
по возрастанию длины (График 1). Мода (=МОДА(B2:B20675)) 1032,
25-й персентиль (=КВАРТИЛЬ(B2:B20675;1)) 999,
75-й персентиль (=КВАРТИЛЬ(B2:B20675;3)) 1061.
То есть характерные длины составляют 999-1061(см.лист 2 файла pr9.xlsx).
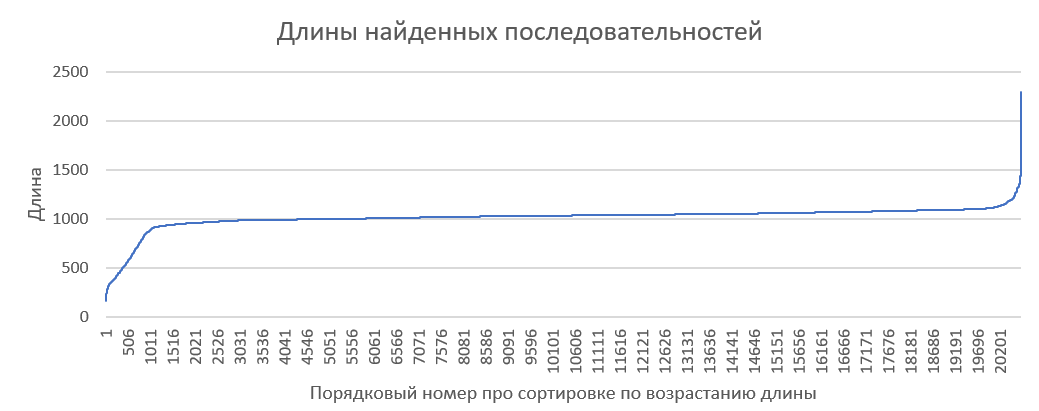 График 1.
Распределение длин последовательностей.
График 1.
Распределение длин последовательностей.
Чтобы выбрать 40-60 характерных последовательностей, были выполнены действия: 1) Оставлены только последовательности с 2 рассматриваемыми доменами. 2) С помощью числового фильтра от 999 до 1061 включительно выбраны последовательности характерной длины. 3) Таблица скопирована и отсортирована по phyllum в алфавитном порядке (сначала мы попробовали брать по 2 представителя из семейства, но их оказалось так много, что при последовательном переборе оказывались не затронутыми некоторые отделы). Из каждого phylum была взята первая находка. После этого получилось меньше, чем 40 последовательностей (установленный в задании минимум), поэтому дальше из каждого отдела было выбрано еще по 1, иногда 2 последовательности из наиболее представленных семейств (разных).
Получилось 55 последовательностей (см. 3-5 листы файла pr9.xlsx).
HMM профиль
1) Чтобы найти бактериальные белки с одним доменом, а не со всей доменной архитектурой, поиск по базе Uniprot был выполнен командой database:(type:pfam pf04313) taxonomy:"Bacteria [2]" . Найдено 26508 последовательностей (на 5834 больше, чем при поиске доменной архитектуры из 2 доменов.
Скачать файл c результатом поискаПотом были скачаны все их последовательности в fasta-формате в файл one_domain.fasta (здесь не приводится, так как большой по размеру).
2) Дальше была взята выборка из 55 последовательностей из предыдущего задания. Чтобы получить её выравнивание, была запущена программа Jalview, там выбрано File - Fetch sequences - Uniprot. Текст запроса был сформирован в Excel (лист 6 файла pr9.xlsx) и вставлен в строку поиска. Через некоторое время все последовательности были найдены. Они были вручную выделены синим выделением. После этого нажата кнопка ОК, потом проведено выравнивание с помощью Muscle (Web Service - Muscle) (скачать файл c выравниванием).
N-конец: Началом N-консервативного блока можно считать позицию 246 (Рис. 2).
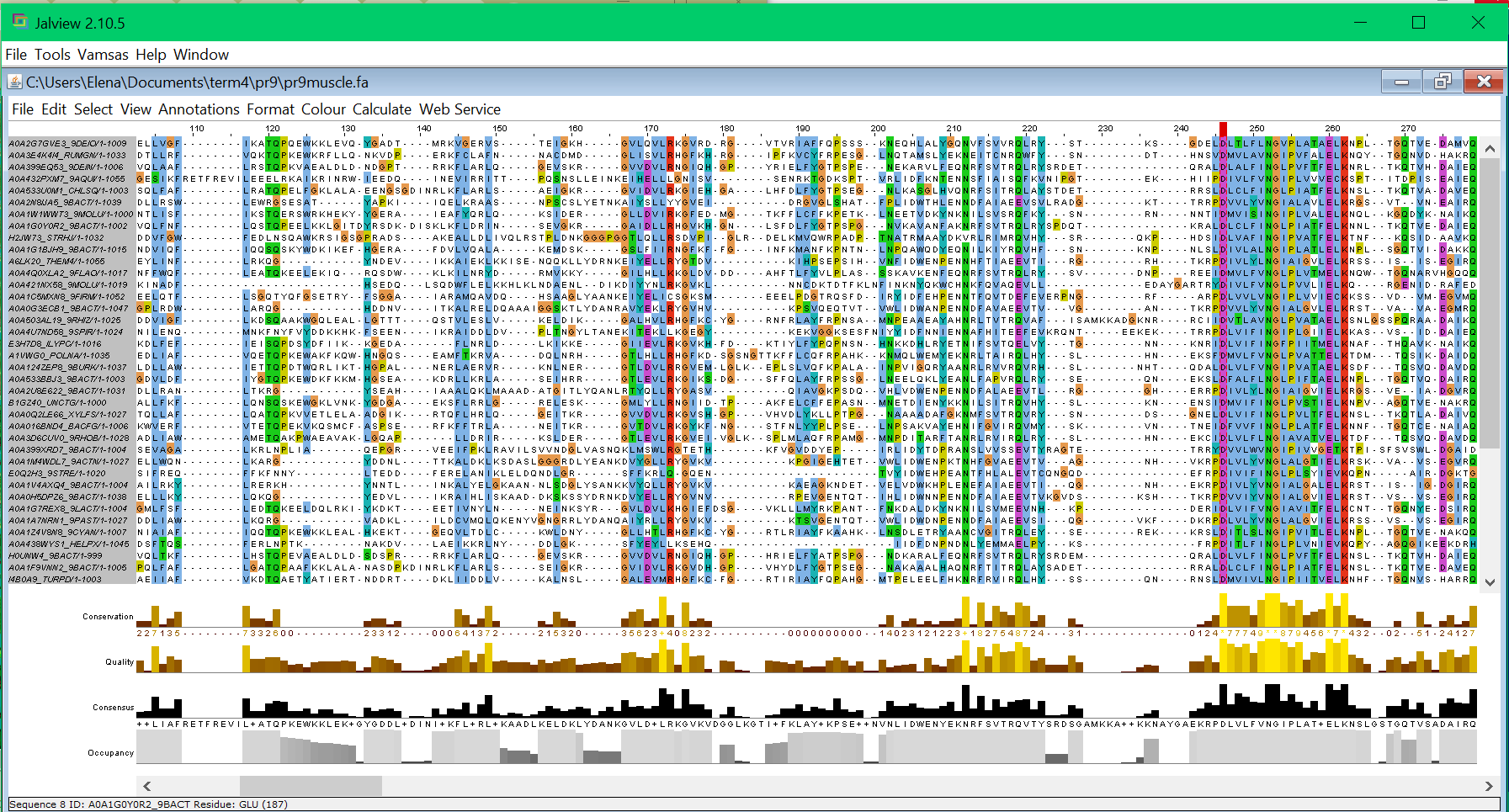
С-конец: Концом C-консервативного блока можно считать позицию 1132 (нумерация до удаления N-конца) (Рис. 4).
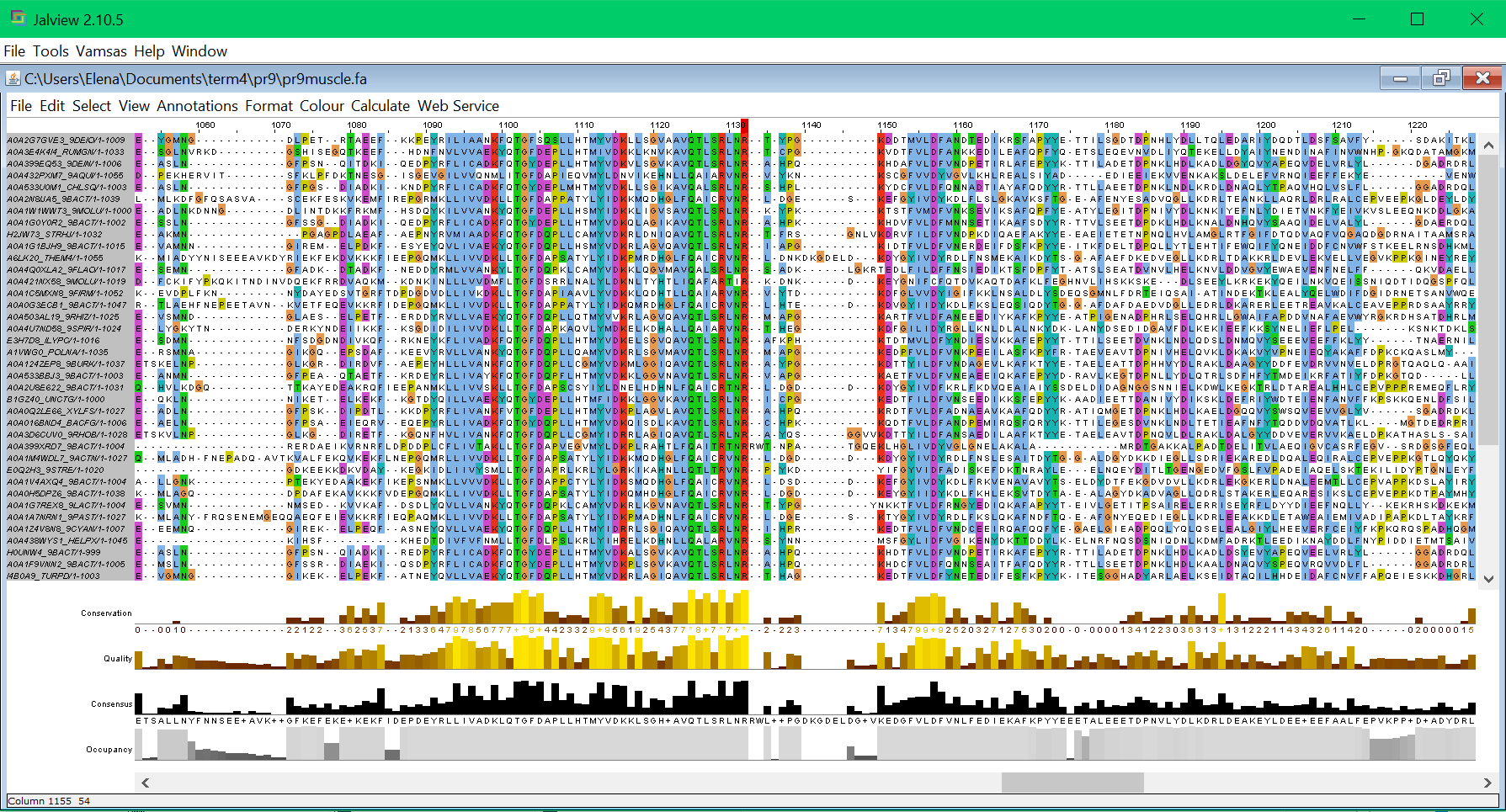
Все колонки до начала N-консервативного блока и после конца C-консервативного были удалены. Все последовательности оставлены (итоговое выравнивание с обрезанными концами).
3) Далее по полученному обрезанному выравниванию был построен HMM-профиль: $ hmm2build -g pr9.hmm pr9muscle-delete.fa (опция -g для глобального выравнивания).
4) Потом профиль был откалиброван: hmm2calibrate pr9.hmm (скачать pr9.hmm)
5) Для выполнения этого действия требуется и откалиброванный профиль, построенный по исходя из выравнивания 55 отобранных двухдоменных последовательностей с обрезанными концами, и fasta однодоменных последовательностей one_domain.fasta (пункт 1)): hmm2search -E 0.1 -T 0 pr9.hmm one_domain.fasta > poisk.txt . Здесь проводится выравнивание белковых последовательностей с одним доменом, которые нашёл uniprot, c hmm-профилем, с вычислением веса выравнивания (score) и E-value, при этом в итоговый файл выводятся лишь находки c порогом E-value 0,1 и положительными score. Полученный файл poisk.txt выглядит так (Рис. 4):
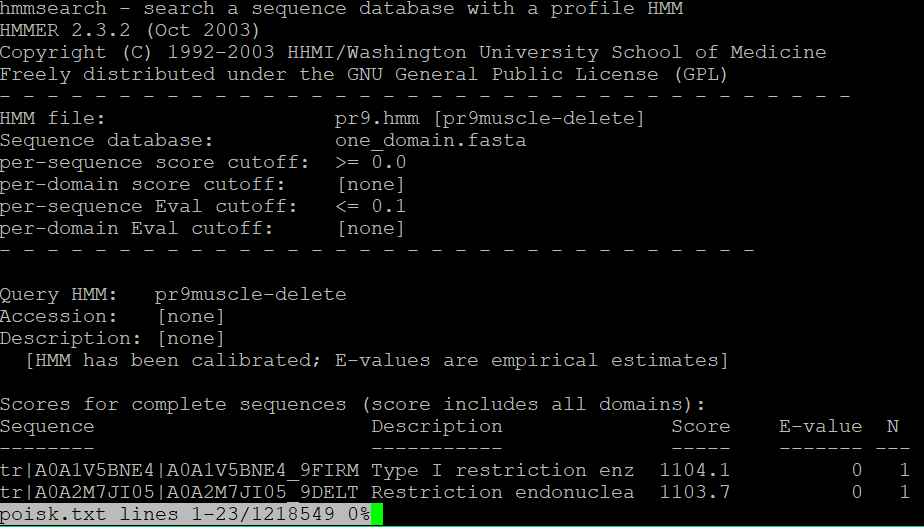
-----Дальнейшие действия находятся в файле pr9final.xlsx В светло-зелёных ячейках лежат находки из файла poisk.txt, то есть последовательности, которые получены hmm при работе с hmm-профилем, построенном на отобранных двухдоменных находках Uniprot, и однодоменными находками (лист 'hmm from 2dom sample&1dom' файла pr9final.xlsx). Во светло-жёлтых ячейках лежат все двухдоменные находки Uniprot (лист '2dom all' pr9final.xlsx). -----
В списке находок по профилю (файл poisk.txt) были найдены те, которые имелись в изначальном списке двухдоменных белков (поиск с помощью функции ВПР в листе 'Сравнение' файла pr9final.xlsx), рядом с каждой находкой по профилю в столбце I указано либо "is_in_2dom_spisok", либо "#H/Д"). Их 19964 штуки из 20583 штук всех находок по профилю, то есть 96,99%.
6) Попробуем подобрать порог веса для предсказания, имеет ли находка требуемую доменную архитектуру.
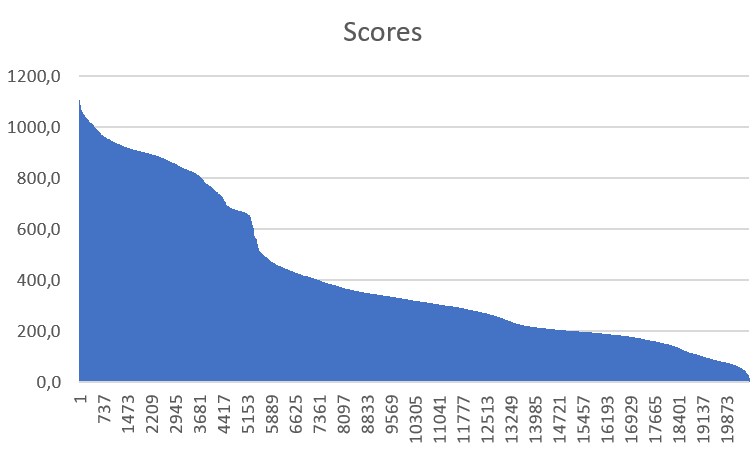
а) Отсортируем находки hmm из файла poisk.txt по убыванию веса и построим график весов находок (График 2) (лист 'График со scores' файла pr9final.xlsx) .
'
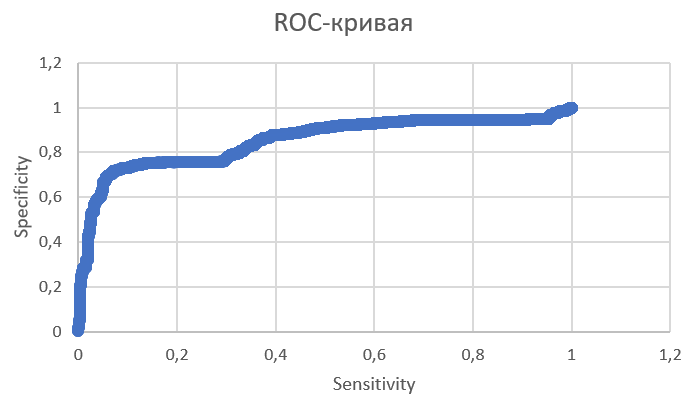
б) Далее были рассчитаны параметры TN, TP, FN, FP, Sensitivity, Specificity. На ROC-кривой (График 3) отображена зависимость специфичность-чувствительность. Расчёты находятся в листе 'Основные расчёты" файла pr9final.xlsx, сама кривая - в листе 'ROC-кривая'.
Там вычислены: TN (True Negatives) - истинно отрицательные случаи. Это число последовательностей ниже порога без искомого домена. TP (True Positives) - истинно положительные случаи. Это число последовательностей выше порога с искомым доменом. FN (False Negatives) - ложно отрицательные случаи. Это число последовательностей ниже порога с искомым доменом. FP (False Positives) - ложно положительные случае. Это число последовательностей выше порога без искомого домена.
Далее вычислены: SE (Sensitivity) - чувствительность = TP/(TP+FN). Чувствительность в нашем случае - это отношение количества предсказанных белков с доменом к количеству последовательностей, которые правда содержат домен. SP (Specificity) - специфичность = TN/(TN+FP). Специфичность в нашем случае - это отношение количества предсказанных белков без домена к количеству последовательностей, которые правда не содержат домен. Precision - точность = TP/(TP+FP) F1-Score = (2*SE*P)/(SE+P)
7) Далее были посчитаны F1-Scores и выбрано максимальное значение. Оно считается пороговым. В нашем случае это 0,984758052582. Удивительно, но это последняя строчка таблицы (20584-ая). Исходя из этого в листе 'Основные расчёты' посчитана матрица ошибок (Рис. 5).

Таким образом, пользуясь нашим предсказанием, то есть основываясь на HMM-профиле, мы почти никогда не будем получать отрицательные результаты, и довольно вероятно, что получим истинно положительный результат.