Семестр 4, практикум 11
Назад на учебную страницу Птицыной ЕленыПрактикум 11: Gene Ontology
Часть 1 - GO Enrichment Analysis
1) В практикуме 11 был рассмотрен набор 32 ID как имён генов или РНК или белков в зависимости от изучаемой базы данных:
list27.txt ADH5 ANPEP ESD GCLC GCLM GGACT GGCT GGT1 GGT2 GGT5 GGT6 GGT7 GGTLC1 GGTLC2 GGTLC3 GPX2 GPX3 GPX4 GPX5 GPX6 GPX7 GPX8 GSR GSS LAP3 NPEPPS OPLAH PRDX1 PRDX2 PRDX3 PRDX5 PRDX6
2) Был использован сервис http://geneontology.org/ с настройками "biological process", "Homo sapiens". Список ID был вставлен в окно поиска. Произошло перенаправление на сайт http://pantherdb.org/. В блоке Results было указано, что 32 из 33 ID было отобрано программой для анализа: это связано с тем, что для гена GPX6 нашлось несколько вариантов, что может быть связано с несоотвествиями ID между различными базами данных:
Single Uploaded id mapped to multiple genes Count PANTHER ids
GPX6 3 HUMAN|HGNC=4559|UniProtKB=Q96SL4,
HUMAN|HGNC=4558|UniProtKB=P59796,
HUMAN|Ensembl=ENSG00000281185|UniProtKB=A3KN74
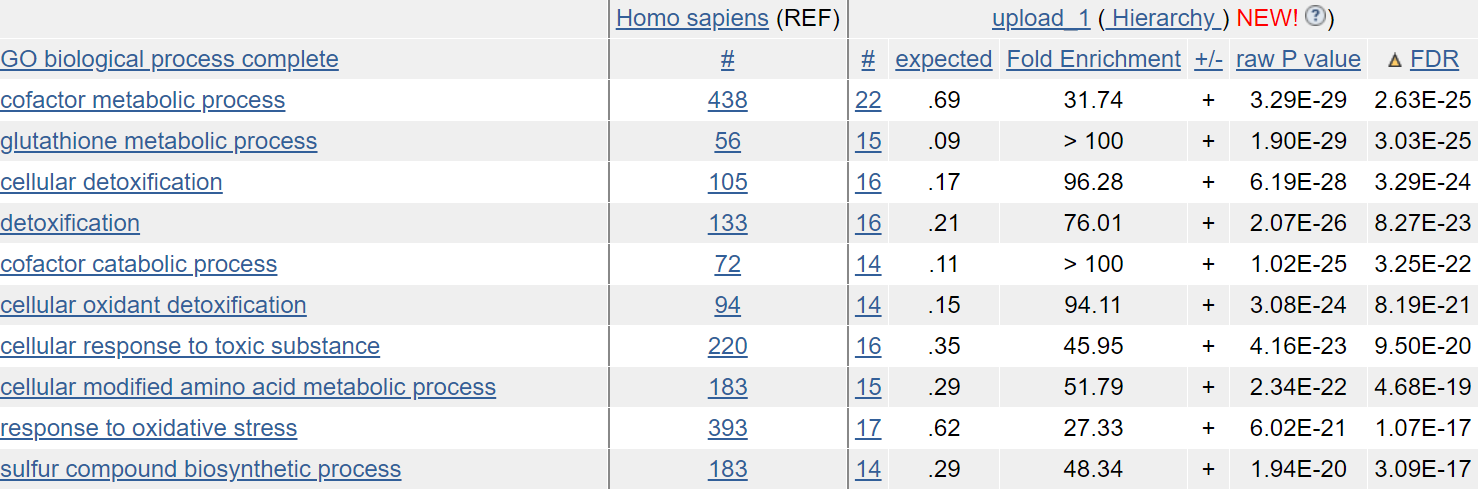
3) В выдаче было 101 GO terms, соответствующих FDR ( False discovery rate) P < 0.05.
4) Чтобы посмотреть на самые значимые GO terms, их список был отсортирован по возрастанию FDR. 10 самых значимых GO-terms: GO:0051186, GO:0006749, GO:1990748, GO:0098754, GO:0051187, GO:0098869, GO:0097237, GO:0006575, GO:0006979, GO:0044272 (Рис. 1).
5) После этого с помощью сервиса https://www.ebi.ac.uk/QuickGO/ отношения между пятью самыми значимыми GO-terms (это GO:0006749, GO:0098754, GO:0051187, GO:0051186, GO:1990748) были визуализированы. Была выбрана вкладка Basket, там через запятую с пробелом введены GO-terms. После этого нажата иконка с изображением дерева. Полученный граф приведен ниже (Рис. 2), жёлытм цветом на нём обозначены блоки, соответствующие введённым GO-terms.
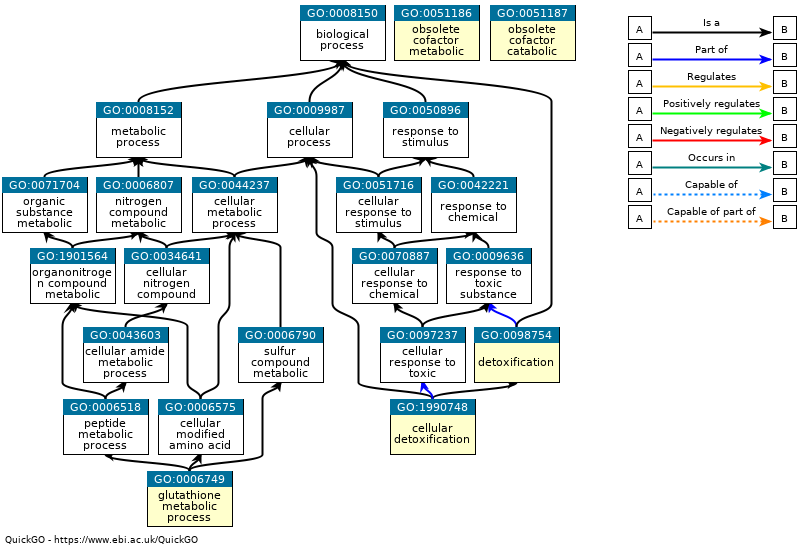
6) Два GO-terms, введённые в QuickGO, оторваны от графа. Это cofactor metabolic process (GO:0051186) и cofactor catabolic process (GO:0051187).
Другие три GO-terms соединены рёбрами с некоторыми узлами. В данном случае наблюдалось два вида рёбер, то есть два вида отношений. "is a"- основное отношение, формирующее структуру GO. "A is a B" означает, что узел A является подтипом узла B (например, митохондрия - is a - органелла). "is part of" применяется для представления отношения часть-целое. "A is a part of B" означает, что B обязательно является частью A (например, митохондрия - is part of - цитоплазма) [1].
Граф распадается на две части. К первой левой части принадлежит glutathione metabolic process (GO:0006749), а ко второй правой - cellular detoxification (GO:1990748) и detoxification (GO:0098754). Части объединяются на уровне принадлежности к подтипам cellular processess и biological processes. Метаболические пути левой части графа, связанные с глутатионом, защищают клетку от окислительно-восстанавительного стресса, а метаболические пути правой части - от детоксикации химическими реагентами, что, впрочем, можно считать разделом окислительно-восстановительной защиты. Кроме того, в печени тот же глутатион может присоединяться к токсичным веществам в процессе их выведения в составе желчи, он участвует в детоксикации метилглиоксаля. Поэтому можно сказать, что данный набор генов имеет отношение к защите от стресса - скорее всего, связанного с токсинами (пришедшими извне и как метаболиты самой клетки).7) Судя по найдённым GO-terms, все ID нашего списка объединяет принадлежность к защитным и утилизационным путям организма. Среди 10 значимых GO-terms несколько раз встречаются детоксикационные модули, ниже встречаются и повторы глутатионных GO-terms. Поскольку глутатион содержит серу и азот, часто попадаются GO-terms, связанные с метаболизмом этих элементов.
Часть 2 - String
Далее список ID был вставлен в окно сервиса https://string-db.org/ на вкладке Multiple proteins с выбором Homo sapiens как организма. Для некоторых ID String предложил несколько альтернативных названий, но правильно выбрал именно те ID, которые были введены.
1) Построенный граф представлен ниже (Рис. 3).

Textmining Experiments Databases Coexpression Neighborhood Gene Fusion Cooccurrence
2) Имеется ли 3D-структура у белка узла, можно понять по его содержимому. Если узел пустой, то 3D-структуры нет, если в нём изображена маленькая молекула - есть. В нашем случае 3D-структуры получены для всех белков, кроме одного - GPX6.
3) Типы взаимодействий, которыми связаны узлы графа, подразделяются на три подгруппы: Known Interactions, Predicted Interactions, Others (Рис. 4).
4) Далее после перехода на вкладку Viewers была нажата кнопка Cooccurrence для изучения представленности генов у разных организмов ("Gene families whose occurrence patterns across genomes show similarities") (Рис. 5).

На выведенной схеме по оси x расположены ID, а по оси y - организмы. Цвет ячеек варьирует от белого до чёрного через красный (Рис. 6):

Если ячейка закрашена в два цвета, это означает, что в кладе есть две группы геномов - с высокой similarity (ярко-красный) и с низкой (розовый).
В нашем случае все гены набора присутствуют у Opisthokonta. Если развернуть кладу несколько раз, выбирая таксоны с наибольшей представленностью, можно обнаружить более мелкую таксономическую единицу, в которой хорошо представлены все введённые гены. В нашем случае это Eutheria из Opisthokonta (Рис. 7). Далее продолжать развёртывание, наверное, не очень целесообразно.

Также интересно, что ген GCLM ( Glutamate-cysteine ligase modifier subunit) представлен у очень малого числа таксонов, кроме Opisthokonta.
5) Далее снова была выбрана вкладка Viewers и выбрана кнопка Coexpression
("Proteins whose genes are observed to be correlated in expression, across a large
number of experiments"). Появившаяся схема, состоящая из 2 частей (для человека и для других организмов)
представлена ниже (Рис. 8).
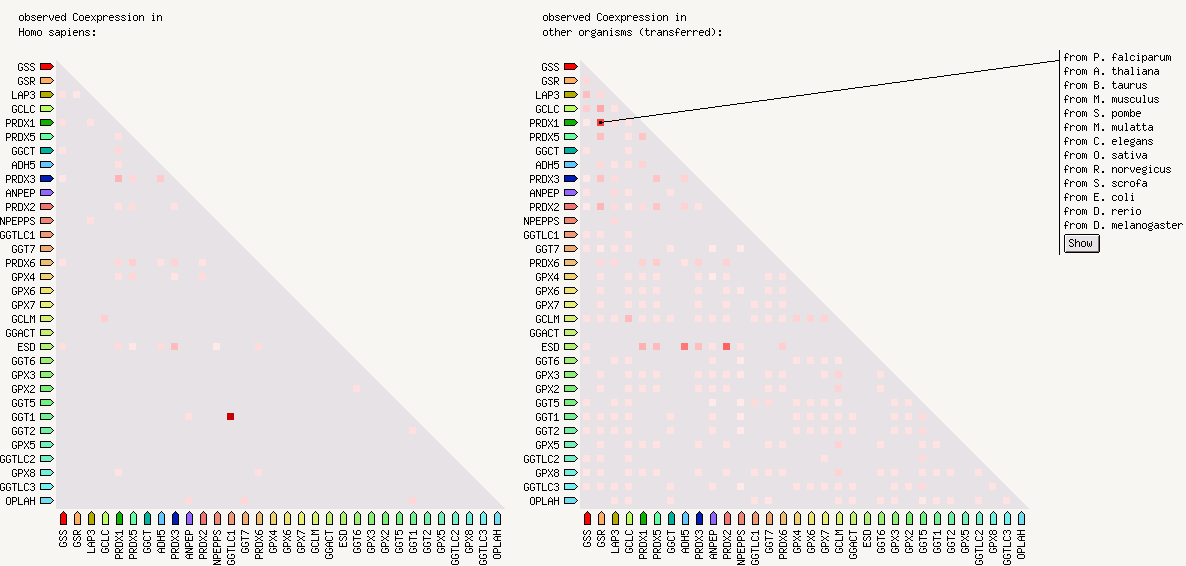
Интенсивность закрашивания ячейки от белого к чёрному через красный означает достоверность выявления коэкспрессии.
В нашем случае у человека более-менее достоверно коэкспрессируются гены GGT1 (Glutathione hydrolase 1 proenzyme) и GGTLC1 (Gamma-glutamyltransferase light chain 1), RNA coexpression score 0.674.
Этой особенности нет у других организмов. Зато у некоторых из них в некоторой степени коэкспрессируются гены PRDX1 (Peroxiredoxin-1) и GSR (Glutathione reductase, mitochondrial). Co score = 0,309 они, предположительно, могут соединяться друг с другом. У меньшего количества организмов String находит коэкспрессию ESD (S-formylglutathione hydrolase) и PRDX2 (Peroxiredoxin-2).
Часть 3 - Human Protein Atlas
1) Далее один из ID (GCLM - Glutamate-cysteine ligase modifier subunit) был вставлен в окно сервиса https://www.proteinatlas.org/.
2) Во вкладке Summary (сверху) есть раздел Gene summary (Entrez), где указано, что название лигазы, субъединицу которой кодирует ген GCLM - glutamate-cysteine ligase = gamma-glutamylcysteine synthetase, и что это первый лимитирующий фермент синтеза глутатиона. Он состоит из двух субъединиц - тяжёлой каталитической и лёгкой регуляторной, и именно регуляторную кодирует ген GCLM. Имеет место альтернативный сплайсинг, приводящий к появлению нескольких изоформ. У данной субъединицы есть два разных транскрипта. Дефицит белка glutamate-cysteine ligase имеет отношение к некоторым формам гемолитической анемии.
3) В разделе Brain Atlas вкладки Summary написано, что ни в мозге человека, ни в мозге свинки, ни в мозге мыши нет специфичных мест локализации GCLM (low region specifity). Эту информацию Human Protein Atlas получает исходя из уровней экспресии мРНК в 10 главных регионах мозга из баз GTEX и FANTOM5.
4) Во вкладке Cell (сверху) можно увидеть
специфичную субклеточную локализацию белка. Он локализуется в основном в нуклеоплазме и цитозоле,
а также был детектирован и в плазматической мембране.

5) Во вкладке Tissue (сверху) есть информация о том, различается ли ID по экспрессии РНК и белка. В Cerebral cortex белка чуть больше, чем РНК. В eye нет экспрессии белка. В endocrine tissues РНК и белок экспрессируются примерно одинаково. В lung чуть больше экспрессии белка, и т.д (Рис. 10). Можно заметить, что особенно активно экспрессируется ID в печени и желчном пузыре, что соответствует тому, что GCLM участвует в синтезе глутатиона.
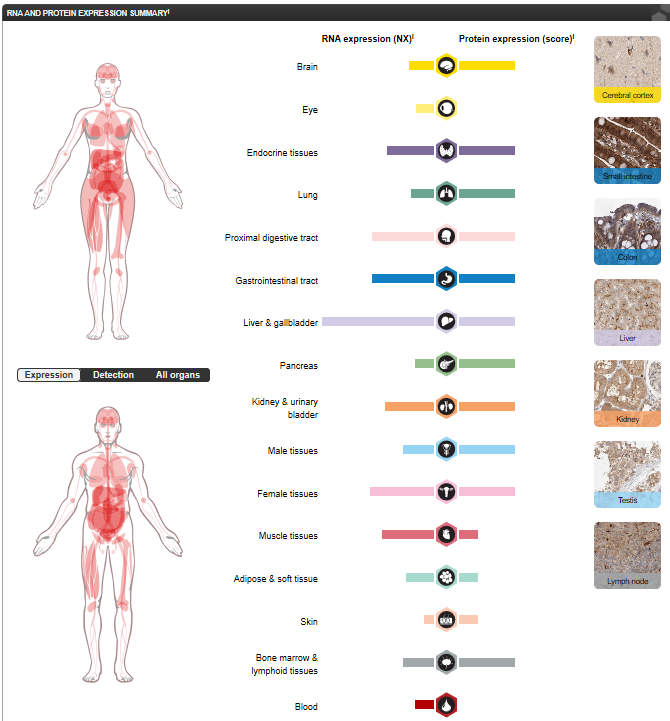
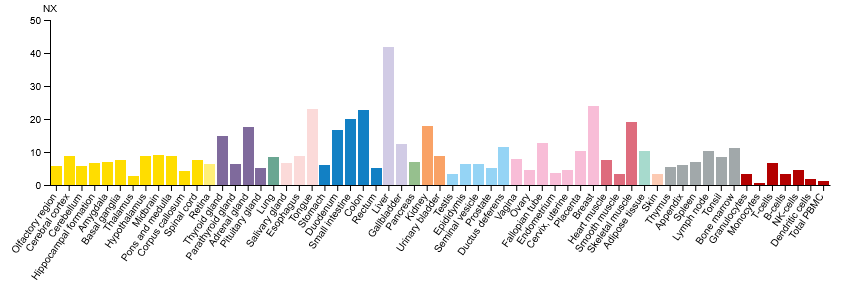
6) Далее после возврата на вкладку Summary (сверху) была нажата кнопка RNA DATA (серый блок слева). Там можно найти информацию о том, в каких тканях экспрессируется ID, если рассматривать его как РНК (Рис. 11). Данные приведены для 55 тканей и 6 типов клеток крови по базам HPA, GTEx и FANTOM5. Больше всего, опять же, РНК экспрессируется в liver, чуть меньше - в breast и tongue (языке). Меньше всего - в моноцитах.
Примечание. Во вкладке Pathology (сверху) можно посмотреть, с какими болезнями
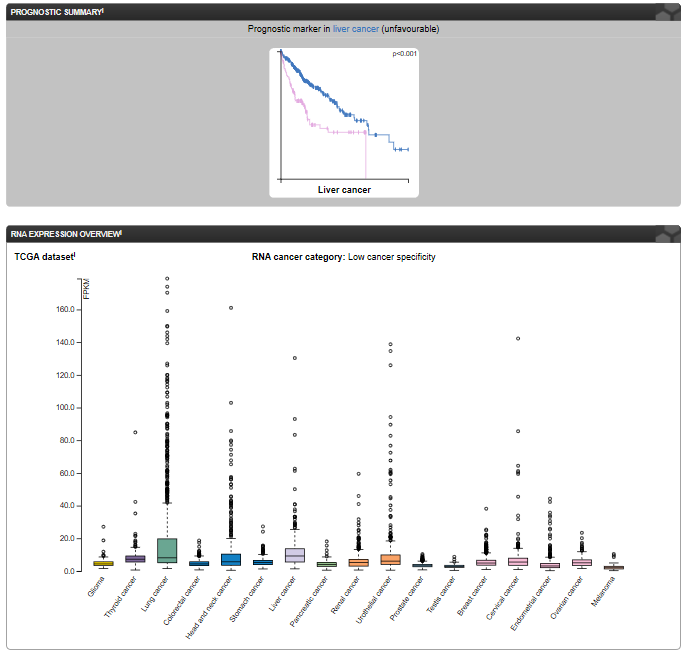
ассоциирован ID - в первую очередь злокачественными (их Gene summary мы помним, что еще полная лигаза, субъединицей которой является наш белок, связана с гемолитической анемией.
В принципе, он может рассматриваться как неблагоприятный прогностический маркер рака печени
(Рис. 12), но имеет низкую специфичность по разным видам злокачественных опухолей (Рис. 12).
Источник:
[1]Relations in the Gene Ontology