Семестр 4, практикум 4
Назад на учебную страницу Птицыной ЕленыПаралоги, визуализация
В 4 практикуме был выполнен поиск гомологов белка CLPX_ECOLI в некоторых других бактериях. CLPX_ECOLI - это компонент протеазы Clp. Он направляет протеазу к специфичным субстратам, используя в некоторых случаях адаптерные белки (например, SspB).
Бактерии
| Название | Мнемоника |
|---|---|
| Streptococcus pyogenes serotype M1 | STREQ |
| Streptococcus pneumoniae serotype 4 | STRPN |
| Lactococcus lactis | LACLA |
| Lactobacillus acidophilus | LACAC |
| Lactobacillus delbrueckii subsp. bulgaricus | LACDA |
| Enterococcus faecalis | ENTFA |
| Staphylococcus aureus | STAA8 |
| Staphylococcus epidermidis | STAEQ |
По сравнению с предыдущим практикумом сделаны замены: STRP1 = STREQ, LACLM = LACLA, STAAR = STAA8, STAES = STAEQ.
Предварительные действия
1) Скопирована папка Proteomes:
elena-pt@kodomo:~/term4/pr3$ cp -r /P/y18/term4/Proteomes .
2) C помощью cat склеены fasta файлы (содержимое первого файла добавляется к содержимому второго):
elena-pt@kodomo:~/term4/pr3/Proteomes$ cat STREQ.fasta >> STRPN.fasta elena-pt@kodomo:~/term4/pr3/Proteomes$ cat STRPN.fasta >> LACLA.fasta elena-pt@kodomo:~/term4/pr3/Proteomes$ cat LACLA.fasta >> LACAC.fasta elena-pt@kodomo:~/term4/pr3/Proteomes$ cat LACAC.fasta >> LACDA.fasta elena-pt@kodomo:~/term4/pr3/Proteomes$ cat LACDA.fasta >> ENTFA.fasta elena-pt@kodomo:~/term4/pr3/Proteomes$ cat ENTFA.fasta >> STAA8.fasta elena-pt@kodomo:~/term4/pr3/Proteomes$ cat STAA8.fasta >> STAEQ.fasta
3) Полученный суммарный файл скопирован на уровень выше и переименован в bact.fasta:
elena-pt@kodomo:~/term4/pr3/Proteomes$ cp STAEQ.fasta .. elena-pt@kodomo:~/term4/pr3$ mv STAEQ.fasta bact.fasta
4) Индексирование последовательностей:
makeblastdb -in bact.fasta -dbtype prot -out bactdb.fasta Building a new DB, current time: 03/06/2020 02:51:54 New DB name: bactdb.fasta New DB title: bact.fasta Sequence type: Protein Keep Linkouts: T Keep MBits: T Maximum file size: 1000000000B Adding sequences from FASTA; added 18242 sequences in 1.16651 seconds.
5) Загружена аминокислотная последовательность P0A6H1, чьи гомологи надо найти:
elena-pt@kodomo:~/term4/pr3$ wget https://www.uniprot.org/uniprot/P0A6H1.fasta
6) Проведен поиск гомологов, список находок - blastp.fasta:
blastp -query P0A6H1.fasta -evalue 0.001 -db bactdb.fasta -outfmt 6 -out blastp.fasta
7) Из файла с протеомами бактерий bact.fasta в search.fasta скопирована последовательности найденных blastp белков, перечисленных в blastp.fasta.
8) Файл search.fasta загружен в программу MEGA, методом Muscle получено выравнивание (в окне с открывшимися последовательностями Align -> Align By Muscle). Далее в том же окне Data -> Phylogenetic analysis. Выравнивание сохранено в файл align_par.fas.
Дерево
9) Дерево построено в MEGA методом Maximum Likelihood method (Рис.1), сохранено в .nwk формате в Newick Export.nwk. Его скобочная формула с указанием длин: ((((((CLPX_LACLA:0,07175365,CLPX_STRPN:0,11995846):0,07430756,CLPX_ENTFA:0,09719450):0,05216251,(CLPX_STAEQ:0,03911702,CLPX_STAA8:0,00000020):0,19613194):0,11005405,(Q1GAP8_LACDA:0,04724551,Q5FKR6_LACAC:0,05720961):0,06760805):0,48274292,((HSLU_LACDA:0,11847268,HSLU_LACAC:0,12627263):0,12014057,(HSLU_ENTFA:0,14113818,(HSLU_STAEQ:0,04804726,HSLU_STAA8:0,06017697):0,28430339):0,07318007):0,86503565):0,43072226,((Y979_STAEQ:0,13721750,Y1413_STAA8:0,05431039):0,94195723,(((Q1G869_LACDA:0,07027711,M4YWY5_STREQ:0,12726455):0,11737295,CLPL_STAA8:0,12253827):0,12997265,(Q5FHW6_LACAC:0,27961635,((Q1GB74_LACDA:0,10636115,CLPE_LACLA:0,19511972):0,12595903,(A0A0H2USJ7_STRPN:0,08233969,M4YZ72_STREQ:0,14149778):0,36865929):0,12780520):0,10509779):0,49791543):0,21828668,(Q5HRP3_STAEQ:0,25994055,(FTSH_LACLA:0,23595425,((FTSH_STRPN:0,04589359,M4YVJ8_STREQ:0,11191946):0,08962051,(Q839B1_ENTFA:0,12999559,(Q1GBN8_LACDA:0,02160178,Q5FMA3_LACAC:0,09845369):0,12449006):0,04216309):0,06964355):0,15004778):1,44408397);
Без указания длин: ((((((CLPX_LACLA,CLPX_STRPN),CLPX_ENTFA),(CLPX_STAEQ,CLPX_STAA8)),(Q1GAP8_LACDA,Q5FKR6_LACAC)),((HSLU_LACDA,HSLU_LACAC),(HSLU_ENTFA,(HSLU_STAEQ,HSLU_STAA8)))),((Y979_STAEQ,Y1413_STAA8),(((Q1G869_LACDA,M4YWY5_STREQ),CLPL_STAA8),(Q5FHW6_LACAC,((Q1GB74_LACDA,CLPE_LACLA),(A0A0H2USJ7_STRPN,M4YZ72_STREQ))))),(Q5HRP3_STAEQ,(FTSH_LACLA,((FTSH_STRPN,M4YVJ8_STREQ),(Q839B1_ENTFA,(Q1GBN8_LACDA,Q5FMA3_LACAC))))));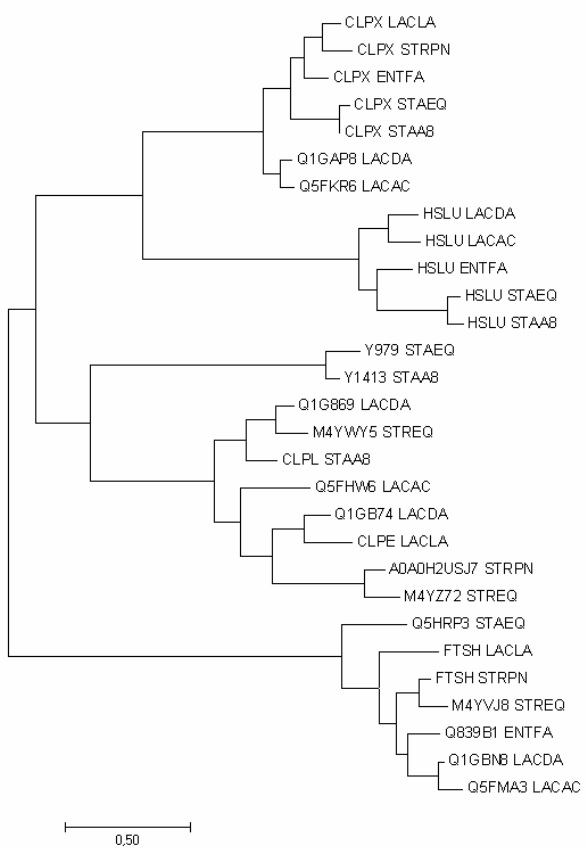
Гомологичные белки - белки, имеющие общее происхождение. Два гомологичных белка будем называть ортологами, если они: а) из разных организмов; б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования. Примеры ортологов: CLPX LACLA и CLPX STRPN, CLPX STAEQ и CLPX STAA8, HSLU LACDA и HSLU LACAC. Два гомологичных белка из одного организма будем называть паралогами. Примеры паралогов: CLPX ENTFA и HSLU ENTFA, CLPX STAA8 и HSLU STAA8, Q1G869 LACDA и Q1GB74 LACDA.
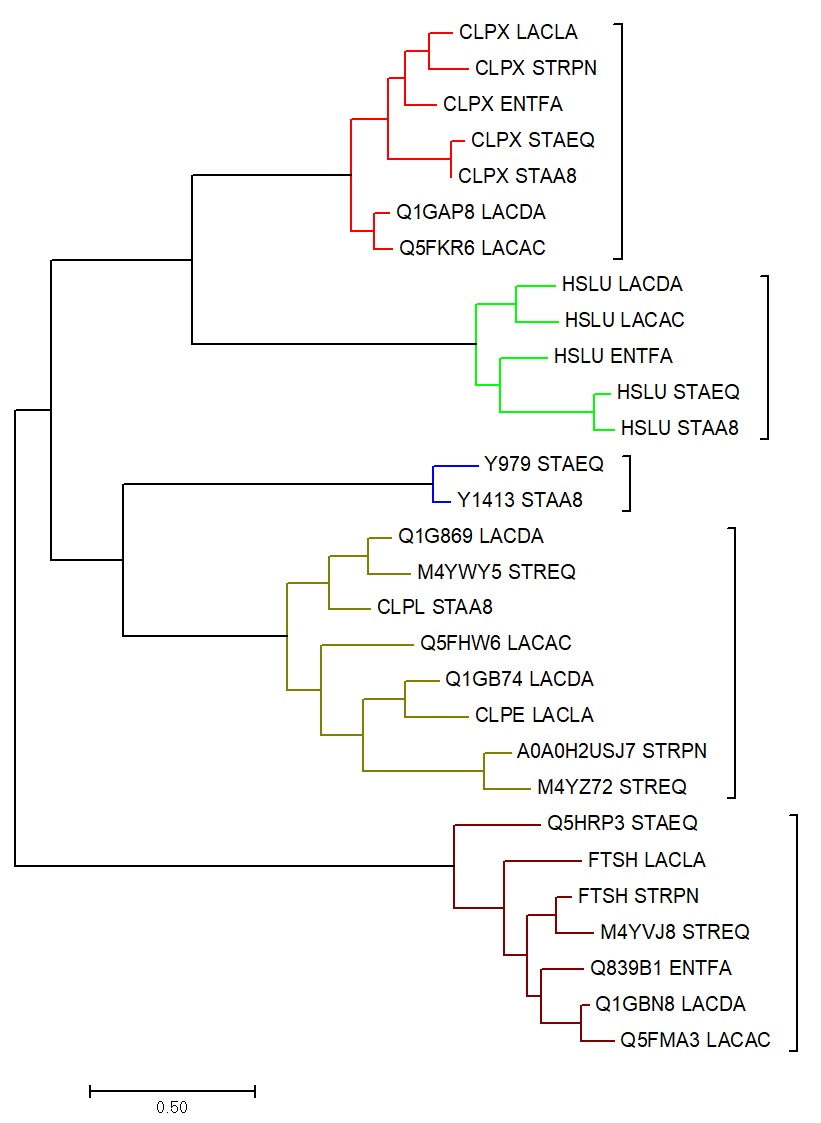
Группы ортологов были покрашены в разные цвета (Рис. 2) и далее схлопнуты (Рис. 3). Чтобы покрасить поддерево, нужно выделить его нижний узел, далее нажать Subtree -> Draw Options. Чтобы схлопнуть поддерево, нужно выделить узел и выбрать опцию схлапывания, а потом получившимся инструментом нажимать на узлы остальных поддеревьев.
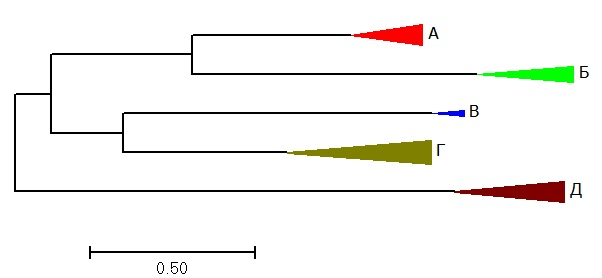
Вспомним филогенетическое дерево, построенное в предыдущем практикуме (по техническим причинам STRP1 = STREQ, LACLM = LACLA, STAAR = STAA8, STAES = STAEQ).
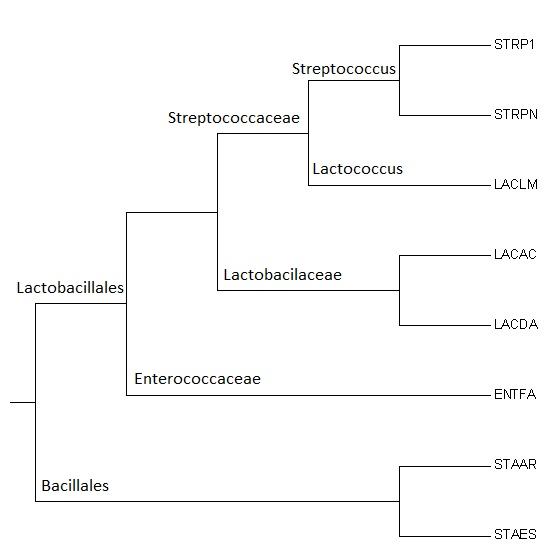
В группе А представлены белки CPLX (5 штук) и отделившаяся от них ветвь Q1GAP8-Q5FKR6. ATP-dependent Clp protease ATP-binding subunit ClpX - это шаперон, служащий субстрат-специфичным адаптером для сериновой протеазы ClpP, как мы уже говорили в начале практикума, в комплексах протеаз ClpXP и ClpAXP. Q1GAP8 и Q5FKR6 - те же CLPX для других бактерий. Как видно, разделение Q1GAP8 и Q5FKR6 полностью повторяет филогенетическое разделение LACDA и LACAС. Так же правильно разделились CLPX для бактерий STAEQ и STAA8. Расположение CLPX ENTFA немножко выбивается.
В поддереве Б сгруппировались белки HSLU - ATPase component of the HslVU protease. Структура поддерева очень напоминает нижнюю часть филогенетического дерева из предыдущего практикума, разве что ENTFA в филогенетическом дереве ближе к LACDA и LACAC, чем к STAEQ и STAA8, как здесь.
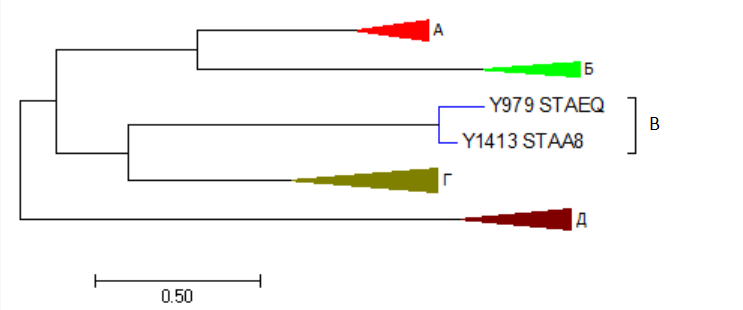
Группа B очень маленькая, по указаниям в задании её можно не выделять отдельным цветом. В ней всего два белка - Y979 из STAEQ и Y1413 из STAA8, это отражает видообразование STAA8 и STAEQ, воспроизведённое на дереве из предудыщего практикума.
В группе Г Д 8 белков. Q1G869 - это ATP-dependent Clp protease ATP-binding subunit. CLPL - это ATP-dependent Clp protease ATP-binding subunit ClpL. Q5FHW6 - ATP-dependent Clp protease ATP-binding subunit, и т.д. Филогения не совсем повторена. Например, в филогенетическом дереве LACAC и LACDA образуют родственную пару, отдельную от LACLA, здесь же родственную пару образуют LACDA и LACLA, LACAС находится в соседней ветви. Однако некоторые отношения точно воспроизведены.
В последнем поддереве Д встречаются белки FTSH - ATP-dependent zinc metalloprotease FtsH (2 штуки), и другие, так или иначе связанные с металлами. Например, Q5HRP3 - ATP-dependent metallopeptidase FtsH/Yme1/Tma family protein. Отражено раннее отделение STAEQ, правда, без STAA8. Необычно близко к корню находится LACLA. Хорошо повторены отношения между LACAC, LACDA и ENTFA, правильную пару образуют STRPN и STREQ.