Комплексы ДНК-белок
Задание 1. Предсказание вторичной структуры заданной тРНК
1G59: Score 160: 23/28 ( 82%) matches, 15 gaps
1 g-g-ccccatcgtctagcgg-ttaggacgcggccc-tctcaag 39
| | ||||| | | || | | || | ||| | || |
73 cactggggt--c----ccccttagcttg-g--gggcaaag--c 42
Результат - первая половина последовательности выровнена со второй, такие предсказания вторичной структуры ненадёжны. Однако нуклеотиды с 3 по 7 спаренные, а это значит, что в структуре такой стебель действительно есть.
Изображение тРНК, полученное при помощи алгоритма Зукера
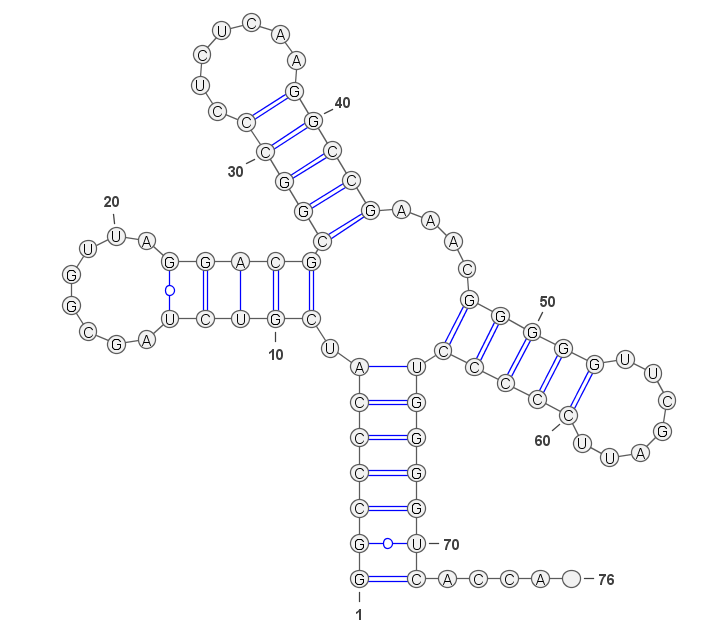
Таблица 1. Сравнения предсказаний структуры тРНК
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5' 01-07 3' 3' 72-66 5' 7 пар | 6 пар из 7 | 7 пар из 7 |
| D-стебель | 5' 10-12 3' 3' 25-23 5' 3 пары | - | 3 пары из 3 + 2 лишних |
| T-стебель | 5' 26-32 3' 3' 44-38 5' 7 пар | - | 5 пар из 7 |
| Антикодоновый стебель | 5' 49-54 3' 3' 65-58 5' 7 пар | - | 5 пар из 7 |
| Общее число канонических пар нуклеотидов | 24 | 23 | 22 |
Задание 2.
Зададим с помощью команды define множества атомов в JMol:
Скрипт, который задаёт эти множества можно найти здесь. Теперь получим скрипт, вызов которого в JMol даст:
1) изображение всей структуры ДНК
2)только ДНК в проволочной модели
3)той же модели, но с выделенными шариками множеством атомов set1, затем set2 и set3.
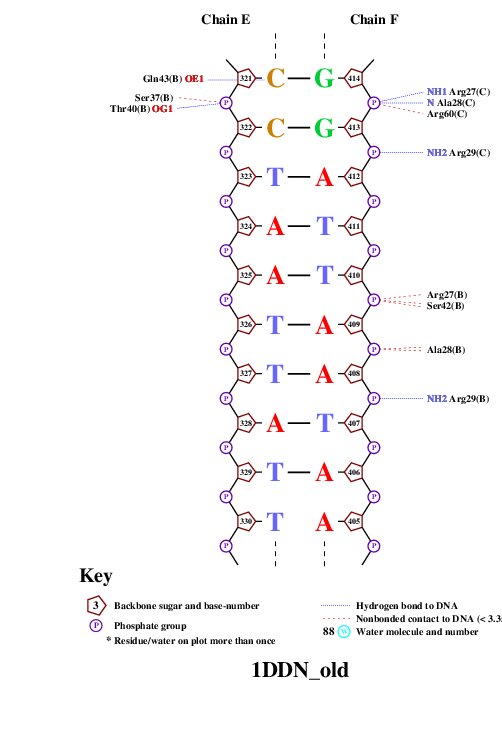
Таблица 2. Число контактов между ДНК и белком в 1DDN
| Контакты атомов белка с различными остатками | Полярные | Неполярные | Всего |
| 2'-дезоксирибозы | 13 | 47 | 60 |
| фосфорной кислоты | 45 | 129 | 174 |
| азотистых оснований со стороны большой бороздки | 9 | 21 | 30 |
| азотистых оснований со стороны малой бороздки | 0 | 3 | 3 |
Больше всего неполярных контактов белка с фосфорной кислотой. Также примечательно, что белок лежит почти исключительно в больших бороздках ДНК (см. Таблицу 1).
| Рисунок 1. | Рисунок 2. | Рисунок 3. | Рисунок 4. |
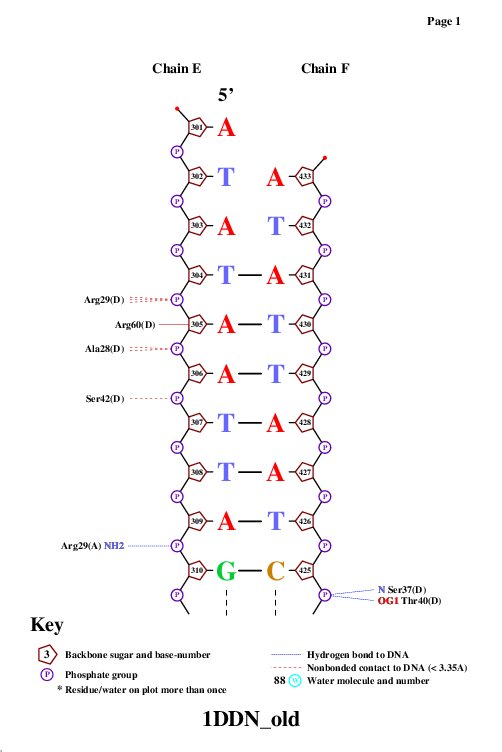
|
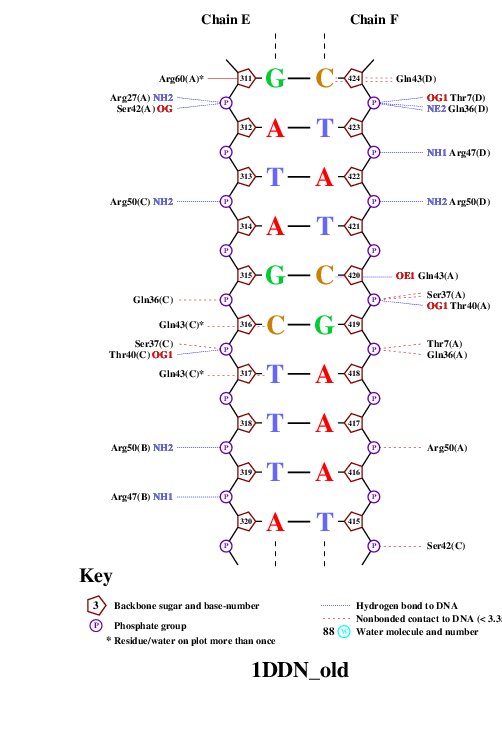
|
|
|
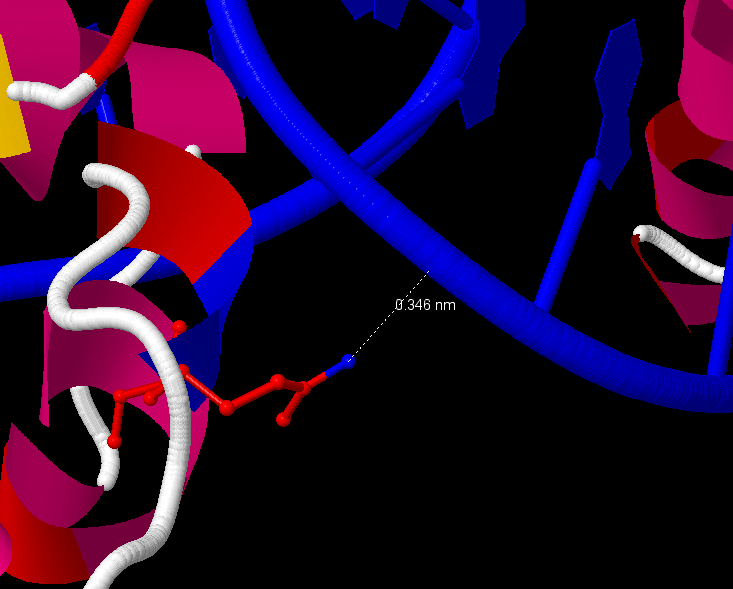
Теперь найдём аминокислотный остаток, образующий больше всего контактов с ДНК. Это Arg29 из цепи D (образует 3 водородных связи)(см. рисунок 1).
Рис.5. Все аргинины в белке выделены красным
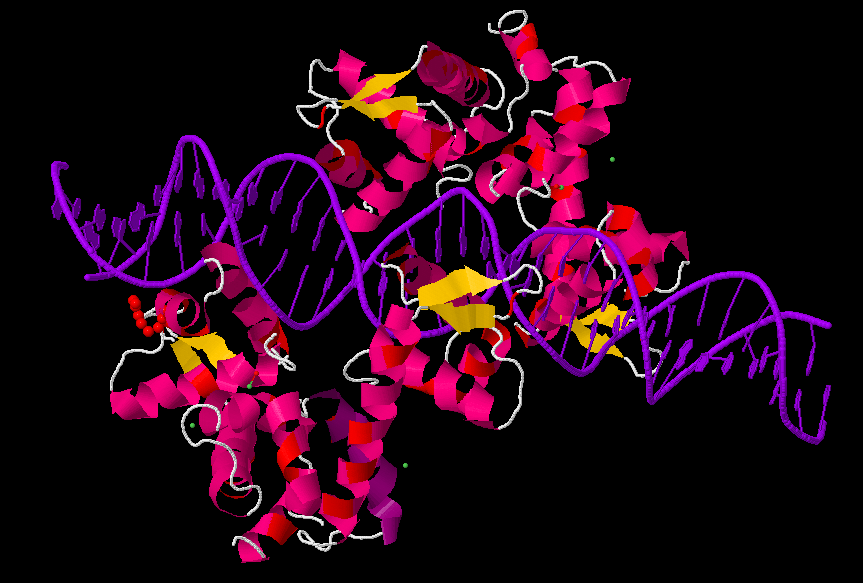
Рис.6. Одна из водородных связей между аргинином (выделен cpk) и ДНК (выделена синим)