Филогенетические деревья
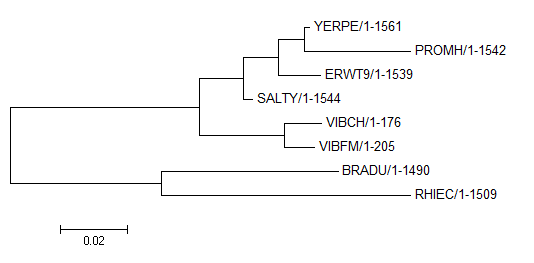
Сравним полученное дерево с деревом, построенным по белковой последовательности (реконструированным тем же методом в MEGA6):
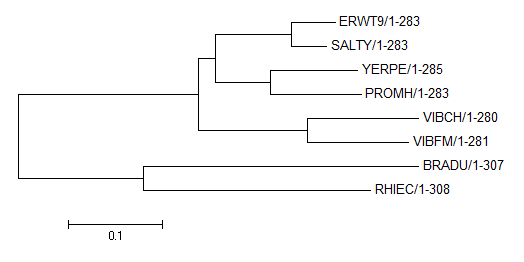
При сравнении двух рисунков видно, что деревья не отличаются расположением ветвей, а только их длинами. Таким образом, точность реконструкции изменилась в лучшую сторону (получена более точная информация о длинах ветвей). На мой взгляд, это произошло потому, что анализируемые нуклеотидные последовательности были длинее белковых в среднем в 1,7 раз (за исключением штаммов VIBCH и VIBFM), и в процессе эволюции появлялось больше мутаций в одной последовательности.
makeblastdb -in allfasta.fasta -dbtype prot -out db.fasta
blastp -query ecoli.fasta -db db.fasta -outfmt 6 -out result.fasta
На выходе я получила файл с информацией о том, какие белки сходны с белком CLPX_ECOLI с E-value = 0.001. Fasta-файл, содержащий последовательности этих белков, я выровняла в Jalview методом Muscle vith defaults, и по полученному выравниванию построила дерево в MEGA6 методом Neighbour-Joining:
Теперь найдём на этом дереве ортологи и паралоги. Напомню, что паралоги - это гомологи из одного организма, а ортологи - гомологи из разных организмов, и разделение их общего предка произошло в результате видообразования.
На рисунке 3 ортологичные группы отмечены зелёной рамкой (в первой ортологической рамке находятся два паралога в красной рамке - очевидно, ортологами являются все белки в данной рамке за исключением этих двух паралогичных белков). Пример паралогов выделен красным, примеры попарных ортологов - фиолетовым.
Я обратила внимание, что в дереве есть две очень короткие ветви - ветвь с паралогами (что говорит о недавней дупликации) и ветвь в оранжевой рамке - практически идентичные белки двух родственных видов.