Практикум 3. Знакомство с Uniprot
В практикуме 3 требовалось ознакомиться с устройством базы данных Uniprot, найти в ней информацию о выданном в 1 семестре белке и научиться проводить сеансы поиска белков.
Получение информации о белке PMEA_DICD3
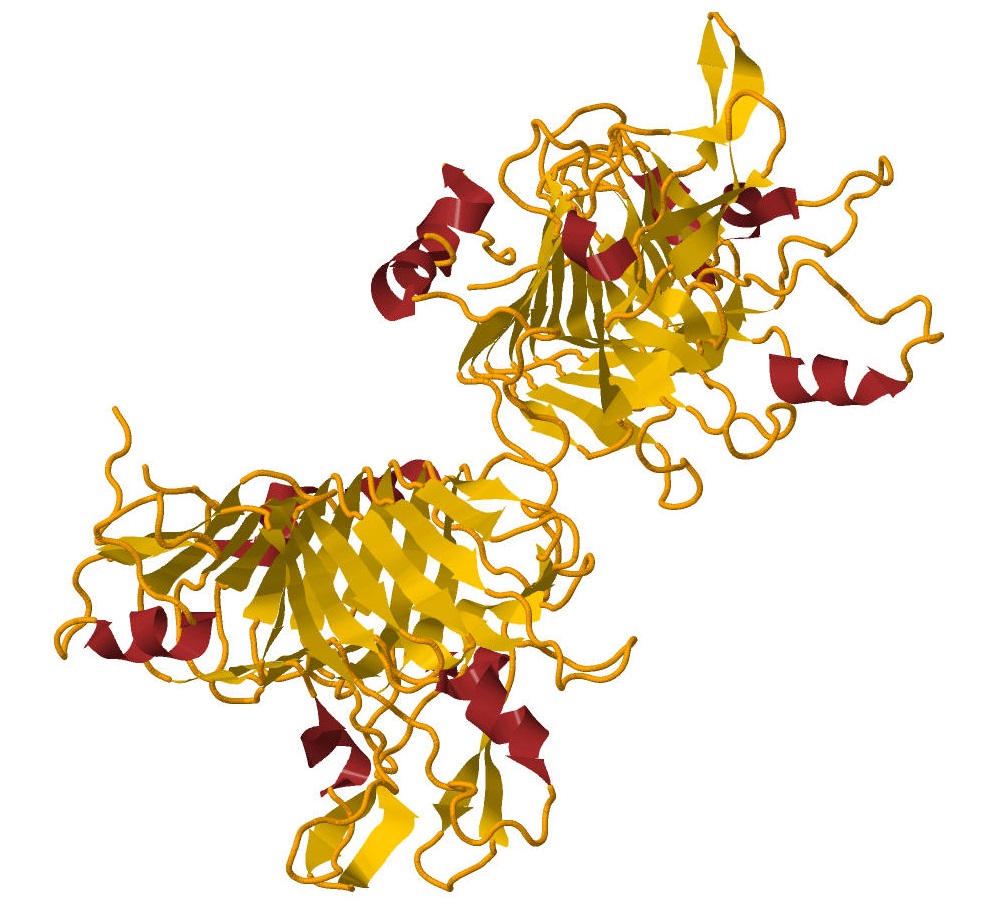
Поиск проводился в форме "Retrieve/ID mapping" по номеру белка (ADM99554.1). Перейдя по ссылке в столбце Entry и изменив формат страницы на .txt, мы получили текстовый файл, из которого взяли некоторую важную информацию. Полученная информация представлена в таблице 1.
| UniProt ID | PMEA_DICD3 |
|---|---|
| UniProt AC | P0C1A9; E0SAZ5; P07863 |
| Название белка | Pectinesterase A (PE A) |
| RefSeq ID | WP_013318985.1 |
| PDB ID | 2NSP; 2NST; 2NT6; 2NT9; 2NTB; 2NTP; 2NTQ |
| Длина белка (а.о.) | 366AA |
| Молекулярная масса (Да) | 39373 MW |
| Рекомендуемое название | Full=Pectinesterase A; Short=PE A; EC=3.1.1.11 |
Белок пектинэстераза состоит из двух субъединиц, и каждая состоит из 366 аминокислотных остатков. Тем не менее, в текстовом варианте страницы UniProt о белке было указано, что первые 24 аминокислотные остатка относятся не цепи (CHAIN), а к сигнальному отрезку (SIGNAL {ECO:0000250}), и они не отображаются в PDB-файлах.
Для белка известно 7 определенных рентгеноструктурным анализом структур разного разрешения.
Поиск белка PMEA_DICD3 в UniRef
Для поиска белковых кластера в строке поиска была выбрана база данных UniRef. Поиск проводился по AC. После отображения таблицы с тремя белковыми кластерами (UniRef90_P0C1A9, UniRef50_P0C1A9, UniRef100_P0C1A9) был просмотрен каждый кластер. Найденная таким образам информация представлена в таблице 2.
| Раздел UniRef | ID кластера | Название кластера | Размер кластера |
| UniRef100 | UniRef100_P0C1A9 | Cluster: Pectinesterase A | 2 |
|---|---|---|---|
| UniRef90 | UniRef90_P0C1A9 | Cluster: Pectinesterase A | 28 |
| UniRef50 | UniRef50_P0C1A9 | Cluster: Pectinesterase A | 204 |
Кластер UniRef100_P0C1A9 содержит всего две последовательности, которые фактически описывают один и тот же белок возможно разных штаммов одного вида бактерий Erwinia chrisanthemi. Кластер UniRef90_P0C1A9 содержит 28 последовательностей, отвечающих нескольким видам одного рода Erwinia. Из этого следует, что белок пектинэстераза A весьма специфичен для этого рода и отвечает конкретным потребностям в приспособлении к паразитизму на высших растениях.
Кластер UniRef50_P0C1A9 составляют, кроме белков бактерий рода Erwinia, еще и белки других патогенных бактерий, вызывающих заболевания в том числе и человека, например, Enterobacter cloacae или Serratia sp.. Не совсем понятно, зачем понадобилась пектинэстераза возбудителю инфекций дыхательных и мочевыводящих путей. Можно предположить, что в автоматически составленной TrEMBL записи допущена ошибка, и белок у Serratia sp. на самом деле выполняет гидролиз не пектина, а другого вещества с сложноэфирной связью (класс 3.1.1 по классификации EC).
Сеансы поиска в UniProt
По заданию требовалось научиться проводить сеансы поиска в UniProt. Для поиска использовался определенный синтаксис: логические операции соединяют выбранные значения определенного типа. Так как в синтаксисе отсутствует понятие "скобок", то в одном из сеансов возникли трудности с поиском белков, удовлетворяющим сложному условию (гистон & (Членистоногие / Инфузории)). Существование определенного синтаксиса, с одной стороны, облегчает поиск, но, с другой стороны, вызывает проблемы с формулировкой условия.
Поиск белка пектинэстеразы A
Поиск по краткому имени
Текст запроса: [name:"pe a"]
Количество находок в Swiss-Prot: 2
Общее количество находок: 53
Поиск по краткому имени и названию организма
Текст запроса: [name:"pe a" AND organism:"Dickeya dadantii (strain 3937) (Erwinia chrysanthemi (strain 3937)) [198628]"]
Количество находок в Swiss-Prot: 1
Общее количество находок: 1
Поиск по краткому имени и названию семейства
Текст запроса: [name:"pe a" AND xxx]
Количество находок в Swiss-Prot: 2
Общее количество находок: 2
Поиск по краткому имени и названию отдела
Текст запроса: [name:"pe a" taxonomy:"Proteobacteria [1224]"]
Количество находок в Swiss-Prot: 2
Общее количество находок: 2
Результаты сеансов показывают, что максимально аккуратным надо быть с поиском по краткому имени, так как могут выдаваться в качестве результата совершенно не отвечающие нашим предпочтениям белки совершенно посторонних организмов (например, никоим образом нас не волнующий организм под названием HUMAN). Поэтому рекомендуется пользоваться полным названием белка. Наиболее информативен поиск по названию белка и таксономическим группам.
Поиск гистонов
Поиск гистонов членистоногих
Текст запроса: [name:histone taxonomy:arthropoda]
Количество находок в Swiss-Prot: 139
Общее количество находок: 32273
Поиск гистонов инфузорий
Текст запроса: [name:histone taxonomy:ciliophora]
Количество находок в Swiss-Prot: 61
Общее количество находок: 491
Из результатов двух запросов можно сделать заключение, что общее число описанных хотя бы компьютером гистонов артропод и цилиофор составляет 32764, из них проверенных кураторами SwissProt - 200. Это возможно потому, что никакие мечехвосты не являются сувойками и никакие парамеции не являются пауками, обратное было бы странно и несомненно привлекло бы научное сообщество.
Поиск трипсинов
Поиск по слову "трипсин"
Текст запроса: [name:trypsin]
Количество находок в Swiss-Prot: 312
Общее количество находок: 22706
Поиск трипсинов, исключая их ингибиторы
Текст запроса: [name:trypsin NOT name:inhibitor]
Количество находок в Swiss-Prot: 101
Общее количество находок: 18270
Результаты запросов показывают, что иногда требуется уточнять условия поиска по названию, так как названия белков бывают очень сложны и длинны. Поиск по слову "трипсин" выдал наряду с непосредственно трипсиноподобными белками сравнимое количество ингибиторов трипсина, например, известный ингибитор трипсина у Arabidopsis, играющий весомую роль в механизме защиты высших растений.