Практикум 10. Геномные браузеры
Task 1
Нужно было найти информацию о каком-то белоккодирующем гене в браузере UCSC. Мы выбрали ген upf1, белок с этого гена входит в сигнальный путь NMD, отвечающий за деградацию мРНК со слишком ранним стоп-кодоном. Информацию нашли, нажав на экзонно-интронный рисунок транскриптов (которых было 2).
| Gene_name, Gencode_ID | upf1, ENSG00000005007.12 |
|---|---|
| Strand | + |
| Chromosome | 19 |
| Band | chr19:p13.11 |
| Location | chr19:18831159-18870673 |
| Alter_products | 2 |
| NM_002911 (Gencode ID ENST00000262803.9) | chr19:18831938-18868236, 24 exons, RENT1_HUMAN 1118 aa |
| NM_001297549 (Gencode ID ENST00000599848.5) | chr19:18832001-18868229, 24 exons, RENT1_HUMAN 1129 aa |
Изображение окружения гена с требуемыми параметрами:
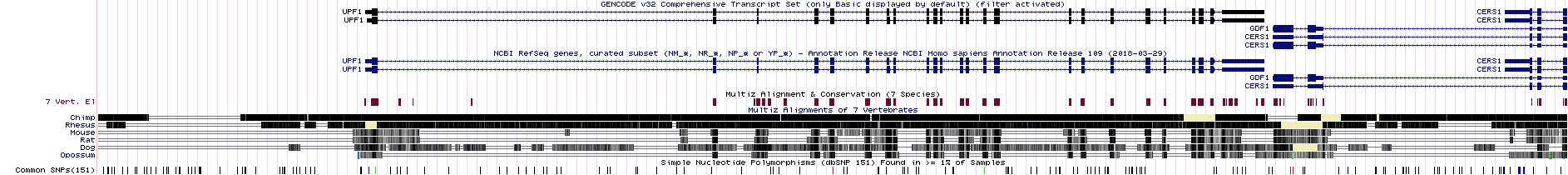
Вполне ожидаемо консервативные среди 7 позвоночных участки находятся преимущественно в экзонных областях.
Task 2
С помощью браузера Ensembl мы получили выравнивание генов upf1 человека и шимпанзе в формате msf. Далее командой "infoalign -sequence Human_Chimpanzee_lastz.msf -outfile Hu_Ch.infoalign -html" была получена информация о выравнивании в виде html-таблицы.
| USA | Name | Sequence Length | Aligned Length | Gaps | Gap Length | Identity | Similarity | Difference | % Change | Weight |
|---|---|---|---|---|---|---|---|---|---|---|
| msf::Human_Chimpanzee_lastz.msf:homo_sapiens/1-36396 | homo_sapiens/1-36396 | 36299 | 36396 | 20 | 97 | 36299 | 0 | 0 | 0.266513 | 1.000000 |
| msf::Human_Chimpanzee_lastz.msf:pan_troglodytes/1-36396 | pan_troglodytes/1-36396 | 36347 | 36396 | 14 | 49 | 35985 | 0 | 362 | 1.129245 | 1.000000 |
Как можно заметить, для последовательности гена у человека в выравнивании примерно 0.27% оснований неконсервативны, для последовательности гена шимпанзе - примерно 1.13% оснований неконсервативны. Общий процент неконсервативности поэтому можно считать находящимся в границах 1.13-1.40% (он не меньше процента неконсервативности любой из последовательностей, и не больше суммы процентов неконсервативности всех последовательностей (т.к. это худший случай, когда позиции неконсервативных оснований не пересекаются для выравненных последовательностей)). Ссылаясь на статью в "Nature", скажем, что в среднем однонуклеотидные замены привели к различию геномов человека и шимпанзе на 1.23%, а индели - к различию на 1.5%. Наша оценка для гена укладывается в эти рамки.








