Практикум 11. Поиск полиморфизмов
Task 1
Работа велась по сборке hg19 по 12 хромосоме. Использованные команды:
fastqc chr12.fastq | Процедура проверки качества ридов |
|---|---|
java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr12.fastq new_chr12.fastq TRAILING:20 MINLEN:50 | Отброс ридов малой длины, обрезка с конца ридов оснований плохого качества |
fastqc new_chr12.fastq | Проверка качества ридов после триммирования |
hisat2-build chr12.fasta chr12 | Индексирование референса |
hisat2 -x chr12 -U new_chr12.fastq --no-spliced-alignment --no-softclip -S chr12.sam | Выравнивание ридов на референс |
samtools view -b -o chr12.bam chr12.sam | Перевод картирования в бинарный формат |
samtools sort chr12.bam chr12_s | Сортировка картирования |
samtools index chr12_s.bam | Индексирование картирования |
samtools flagstat chr12_s.bam > chr12_s_stat.txt | Статистика картирования |
samtools mpileup -uf chr12.fasta chr12_s.bam -o chr12_pol.bcf | Поиск полиморфизмов |
bcftools call -cv chr12_pol.bcf -o chr12_pol.vcf | Перевод файла с полиморфизмами в читаемый формат |
vcftools --vcf chr12_pol.vcf --remove-indels --recode --out chr12_snp.vcf | Оставить только snp |
convert2annovar.pl -format vcf4 chr12_snp.vcf.recode.vcf -outfile chr12_snp.avinput | Перевод в читаемый для annovar формат |
annotate_variation.pl -out chr12_ann_refgene -build hg19 -dbtype refGene chr12_snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Поиск snp по RefGene |
annotate_variation.pl -filter -out chr12_ann_snp -build hg19 -dbtype snp138 chr12_snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Поиск snp по SNP |
annotate_variation.pl -filter -dbtype 1000g2014oct_all -buildver hg19 -out chr12_ann_1000genomes chr12_snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Поиск snp по 1000 Genomes |
annotate_variation.pl -regionanno -build hg19 -out chr12_ann_gwas -dbtype gwasCatalog chr12_snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Поиск snp по GWAS |
annotate_variation.pl -filter -dbtype clinvar_20150629 -buildver hg19 -out chr12_ann_clinvar chr12_snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Поиск snp по ClinVar |
Мы хотели триммировать риды, потому что не все основания в них были хорошего качества. Поэтому мы обрезали эти основания.
Качество ридов до триммирования:
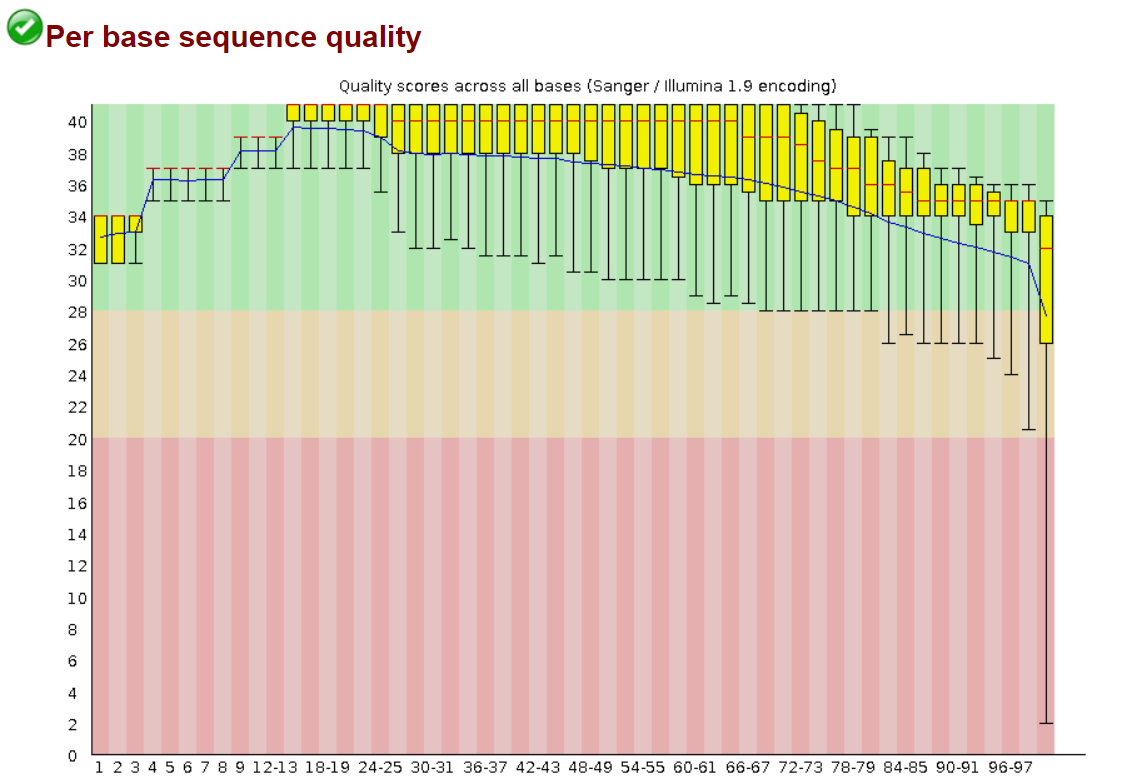
Качество ридов после триммирования:
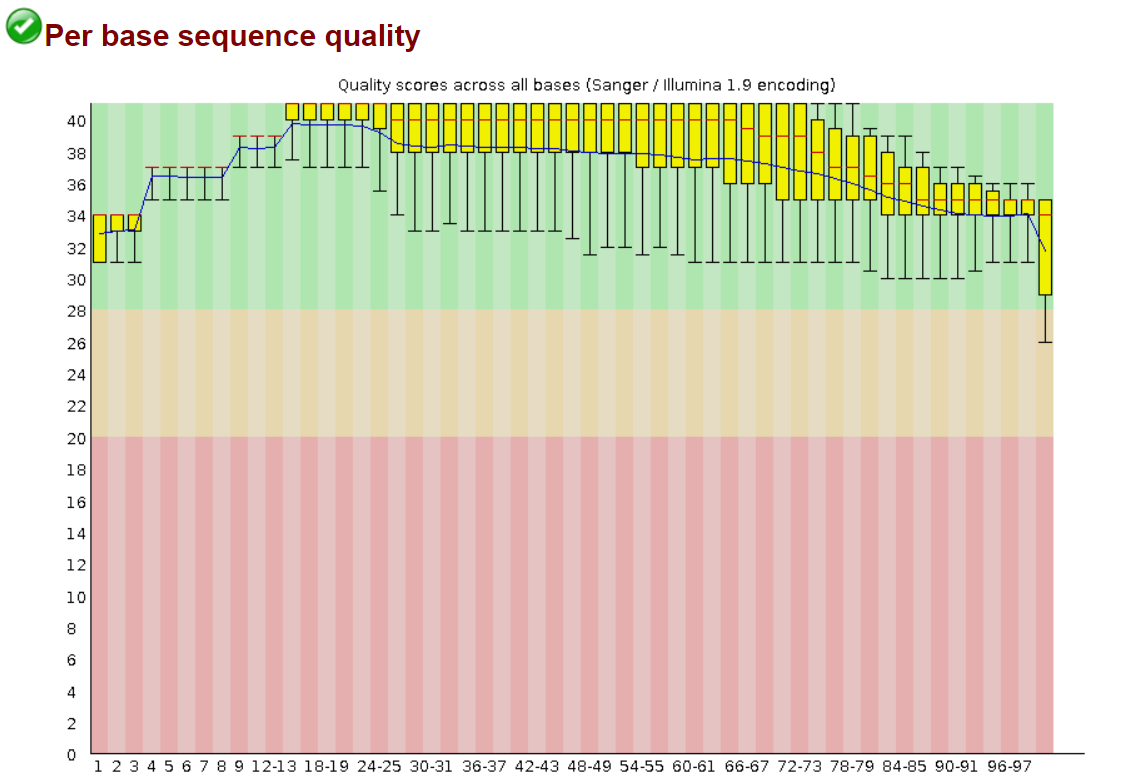
Есть еще такие картинки:
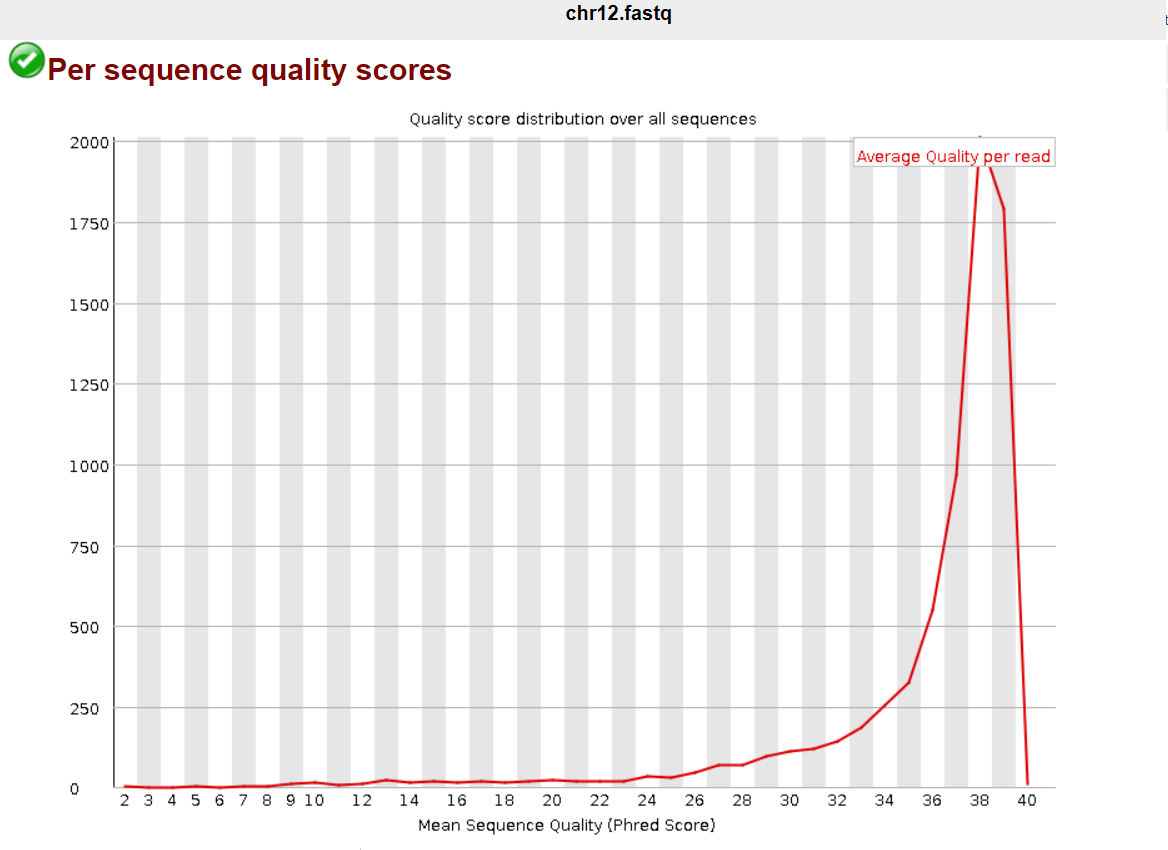
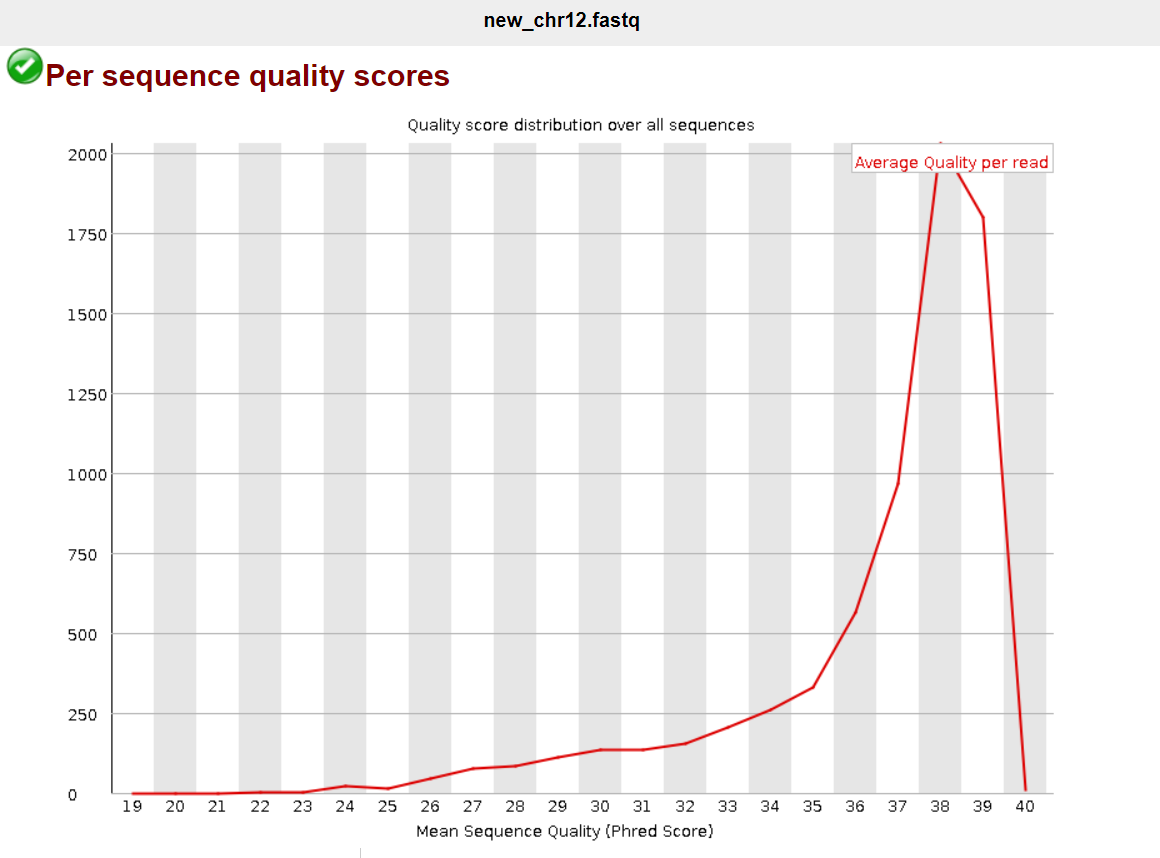
Остальные графики не приведены, ибо разницы никакой нет. Доля плохих ридов была ничтожна, как попытки Соколова облегчить себе участь. Значит, кроме непосредственно качества оснований, ничего особо не улучшилось и теоретически можно было и не триммировать.
До триммирования: 7157 ридов. После триммирования: 7023 ридов. По статистике flagstat, картировано на референс 7003 чтения (99.72%). Статистика hisat2:
7023 reads; of these:
7023 (100.00%) were unpaired; of these:
20 (0.28%) aligned 0 times
7003 (99.72%) aligned exactly 1 time
0 (0.00%) aligned >1 times
Картировалось хорошо, потому что однозначно.
Task 2
В результате поиска полиморфизмов инделей не обнаружилось, все 35 полиморфизмов были snp. Вот 3 достаточно интересных snp:
#CHROM POS ID REF ALT QUAL FILTER INFO FORMAT chr12_s.bam
chr12 9833628 . C T 221.999 . DP=70;VDB=0.496659;SGB=-0.693147;RPB=1;MQB=1;MQSB=1;BQB=1;MQ0F=0;AF1=1;AC1=2; DP4=0,1,11,54;MQ=60;FQ=-184.988;PV4=1,0.23432,1,0.237727 GT:PL 1/1:255,158,0
chr12 66343810 . C T 10.4247 . DP=1;SGB=-0.379885;MQ0F=0;AF1=1;AC1=2;DP4=0,0,1,0;MQ=60;FQ=-29.9905 GT:PL 1/1:40,3,0
chr12 66359752 . C A 221.999 . DP=133;VDB=0.00905334;SGB=-0.693147;RPB=0.941835;MQB=1;MQSB=1;BQB=0.99997; MQ0F=0;AF1=1;AC1=2;DP4=2,1,54,74;MQ=60;FQ=-281.989;PV4=0.575578,0.383652,1,0.295616 GT:PL 1/1:255,255,0
Как можно заметить, качество 1 и 3 snp очень хорошее (221.999), а у 2 - не очень (10.4247). Похожая ситуация с глубиной чтения: хорошая у 1 (70) и 3 (133) и нехорошая (всего 1) у 2.
В файлах, полученных на основе данных RefGene, были такие категории snp:
| exonic | 3 |
|---|---|
| intronic | 25 |
| ncRNA-intronic | 1 |
| UTR3 | 6 |
В базе данных SNP138 из 35 полиморфизмов нашлось 28 (rs).
Из экзонных 2 были несинонимичны (1 и 3 строки) и 1 синонимичный (4 строка). Первый (9822387:C -> G, 1 экзон) приводит к 2 изменениям в любых белковых продуктах гена CLEC2D: c.C57G:p.N19K. Второй (9833524: C -> G, 2 экзон) приводит к 2 изменениям в белковых продуктах гена CLEC2D:c.C67G:p.L23V.
SNP попали в гены RPSAP52, CLEC2D, HMGA2, TMEM263. (согласно RefGene). Средняя частота найденных snp - 0,341781849 (согласно 1000 Genomes).
Клиническая аннотация (GWAS) нашла 4 snp. ClinVar не нашел ничего.
Name=Type 1 diabetes chr12 9833628 9833628 C T hom 221.999
Name=Brain structure chr12 66343810 66343810 C T hom 10.4247
Name=Height chr12 66359752 66359752 C A hom 221.999
Name=Bone mineral density chr12 107367225 107367225 T C hom 221.999
При поиске дополнительной информации о генах оказалось, что некоторые мутации гена HMGA2 действительно ассоциированы с низкорослостью, что подтверждалось на мышах.[1][2]








