Практикум 12. Анализ транскриптомов
Task 1
Работа велась по сборке hg19 по 12 хромосоме. Использованные команды:
fastqc chr12.1.fastq | Процедура проверки качества ридов |
|---|---|
java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr12.1.fastq chr12.1_trim.fastq TRAILING:20 MINLEN:50 | Отброс ридов малой длины, обрезка с конца ридов оснований плохого качества |
fastqc chr12.1_trim.fastq | Проверка качества ридов после триммирования |
hisat2-build chr12.fasta chr12 | Индексирование референса |
hisat2 -x chr12 -U chr12.1.fastq --no-softclip -S chr12_rna.sam | Выравнивание ридов на референс |
samtools view -b -o chr12_rna.bam chr12_rna.sam | Перевод картирования в бинарный формат |
samtools sort chr12_rna.bam chr12_rna_sorted | Сортировка картирования |
samtools index chr12_rna_sorted.bam | Индексирование картирования |
htseq-count -i gene_id -s no -m union -f bam chr12_rna_sorted.bam gencode.v19.chr_patch_hapl_scaff.annotation.gtf -o chr12_rna_count.sam |
Счет ридов: -i какой атрибут используется для индексирования (gene_id); -f формат входного файла (bam); -s взяты ли данные со специфичной цепи или нет (нет); -m счет если: union - из генов легло только на 1; i._strict - лег полностью только на 1 ген, i._nonempty - пересекается с геном, но можно понять, что относится только к нему. |
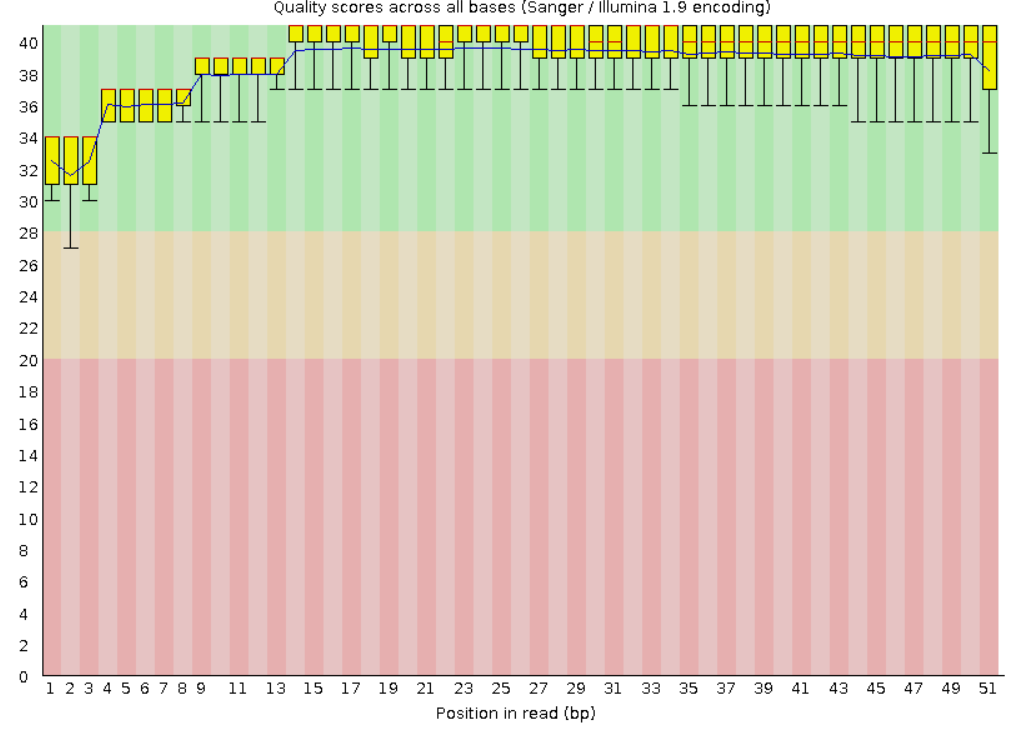
Качество ридов до триммирования:[1]
Качество ридов после триммирования:[2]
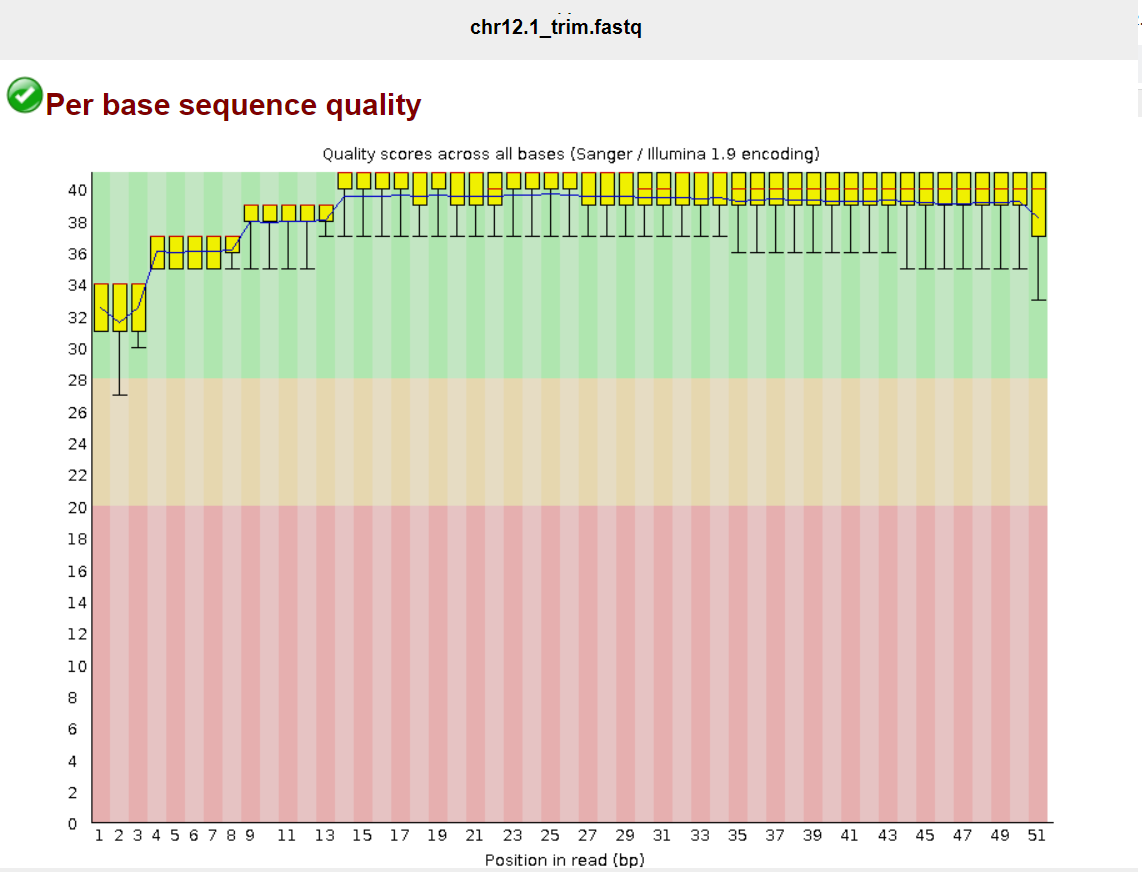
Программа FastQC говорит, что с ридами все очень плохо, а после триммирования даже хуже (при том что качество отдельных оснований очень хорошее и вообще не изменилось).
До триммирования: 16451 рид. После триммирования: 16403 ридов. Работали с ридами до триммирования. Статистика hisat2:
16451 reads; of these:
16451 (100.00%) were unpaired; of these:
396 (2.41%) aligned 0 times
16055 (97.59%) aligned exactly 1 time
0 (0.00%) aligned >1 times
Картировалось хорошо, потому что однозначно.
В результате работы программы Htseq-count с приведенными в таблице выше параметрами был получен файл. В этом файле были такие строчки:
| XF:Z:ENSG00000110955.4 | 15749 | ATP synthase F1 subunit beta |
|---|---|---|
| XF:Z:ENSG00000076108.7 | 10 | Bromodomain adjacent to zinc finger domain 2A |
| XF:Z:ENSG00000207031.1 | 8 | Small nucleolar RNA, C/D box 59A |
| XF:Z:__no_feature | 288 | - |
| XF:Z:__not_aligned | 396 | - |
Как можно заметить, были риды, откартировавшиеся на один из трех генов, были риды, которые откартировались, но непонятно на что, были риды, не откартировавшиеся вообще. Далее в Ensembl мы искали, что это за гены и какие у них продукты. Оказалось, что большинство ридов были откартированы на ген, кодирующий одну из субъединиц АТФ-синтазы. Также интресно, что чуть-чуть ридов попали на ген одной из малых ядрышковых РНК типа C/D box. (У гена всего один продукт - snRNA, она и попала в проанализированную часть транскриптома, видимо, метилировала что-то). Про продукт второго гена BAZ2A известно, что он может связываться с гистоном H3, таким образом регулирует эпигенетические процессы и вообще его экспрессия ассоциирована с различными видами рака.








