Практикум 3. Структуры нуклеиновых кислот
Задание 1
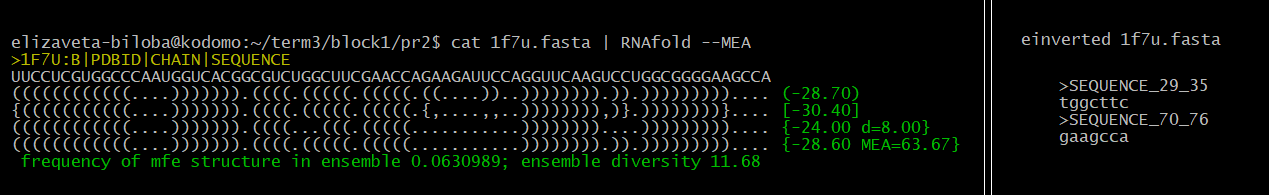
Требовалось найти инвертированные участки в заданной структуре тРНК. Это было проделано 2 способами: с помощью команды 'einverted' (minimum score threshold = 20) пакета EMBOSS и при помощи программы RNAfold (с опцией --MEA), осуществляющей алгоритм Зукера. Анализ результатов приведён в таблице 1.
| features | Structure_positions | einverted (pred) | RNAfold (pred) |
| acceptor stem | 5'-901-907-3' 5'-966-972-3' 7 pairs | 0/7 | 5/7 |
| d-stem | 5'-910-913-3' 5'-922-925-3' 4 pairs | 0/4 | 2/4 |
| t-stem | 5'-949-953-3' 5'-961-965-3' 5 pairs | 0/5 | 2/5 |
| anticodon stem | 5'-926-931-3' 5'-939-944-3' 6 pairs | 0/6 | 2/6 |
| WC bp number | 18 | 7 | 23 |
Нетрудно заметить, что даже с оптимально выбранными параметрами поиска программа einverted плохо справляется с задачей предсказания вторичной структуры. К сожалению, программа RNAfold при меняющихся параметрах выдавала одну и ту же неправильную структуру (и это грустно). Поэтому мы решили воспользоваться алгоритмом Зукера с сайта "http://unafold.rna.albany.edu" и выбрать там удобные параметры (percent of suboptimality = 15). Результат оказался на порядок лучше.
((((((((((((....)))))))...(((((.......))))).....(((((.......)))))..)))))....
В акцепторном стебле совпадают 5 из 7 пар оснований, в d-стебле - 2 из 4, в t-стебле - 5 из 5, в антикодоновом стебле - 5 из 6. Таким образом, алгоритм Зукера неплохо предсказывает вторичную структуру тРНК.
Задание 2
Требовалось посчитать количество контактов ДНК-белок. Это делалось методами Jmol. Краткая информация о том, как проходила работа содержится в файле.
| contact protein- | polar | non-polar | all |
| -deoxyribose | 4 | 41 | 45 |
| -phosphate res | 54 | 48 | 102 |
| -nb res (big) | 7 | 28 | 35 |
| -nb res (small) | 0 | 0 | 0 |
Далее нужно было получить изображение ДНК-белковых контактов с помощью программы nucplot. Со структурой 1tro_old.pdb (старый формат) возникли сложности (Segmentation fault), зато с похожей структурой 1trr_o.pdb (старый формат) все получилось хорошо.
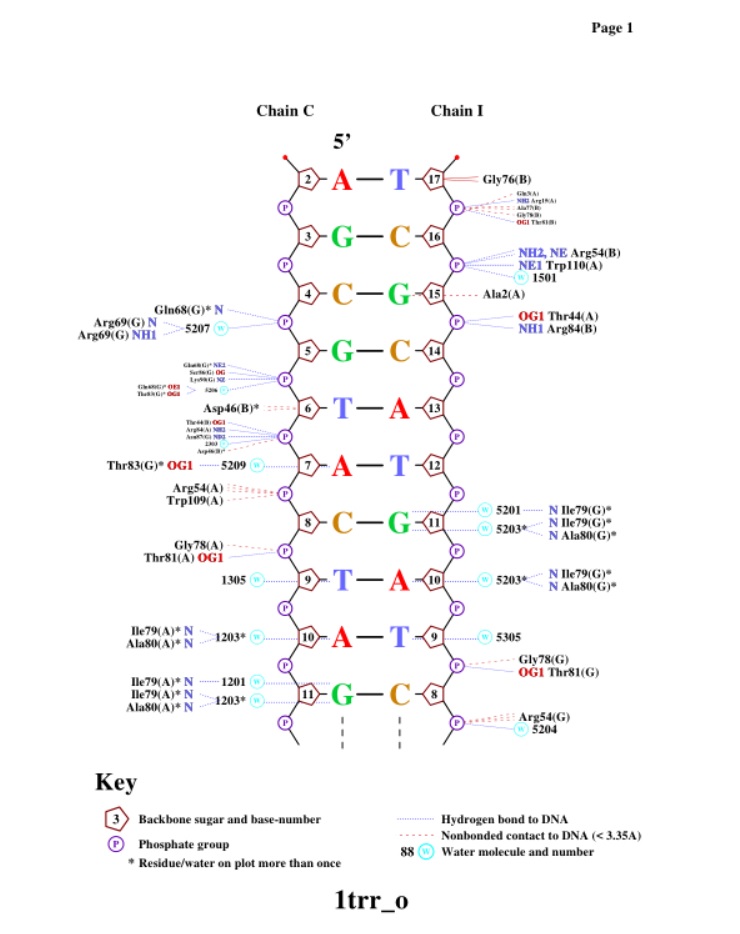
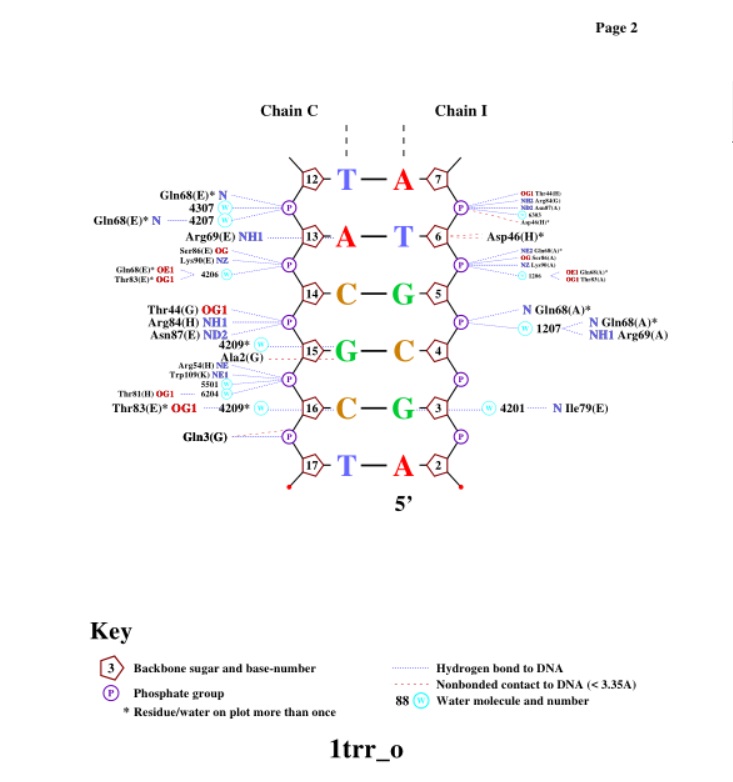
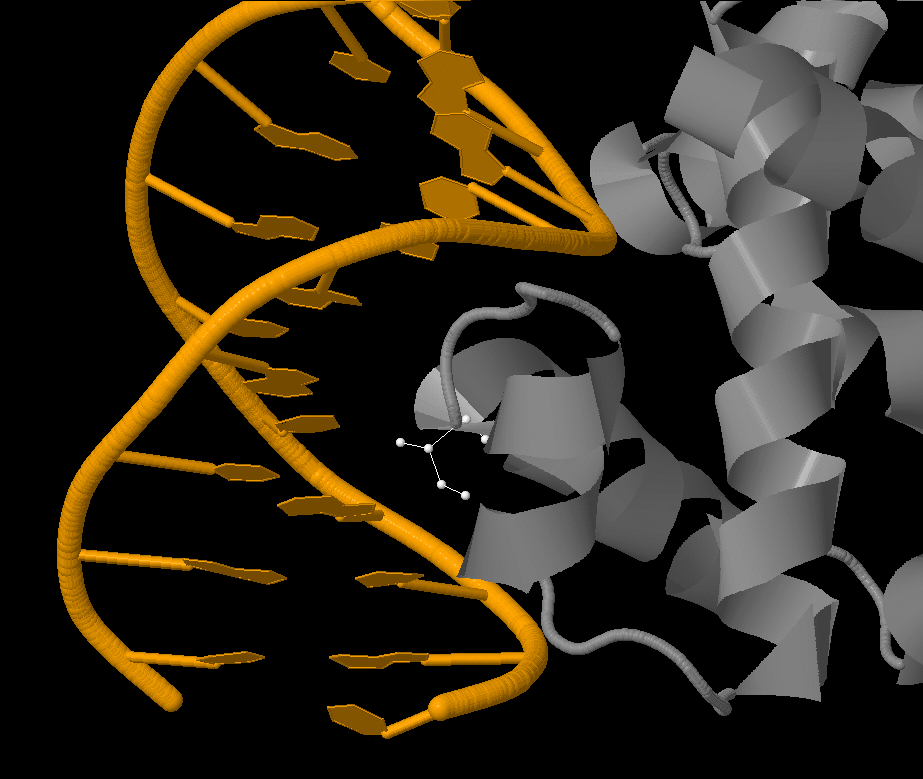
Как можно заметить, структура обладает симметрией, поэтому мы нашли несколько остатков одной аминокислоты (Ile), каждый из которы вступает в 3 взаимодействия с ДНК. Мы выбрали остаток Ile79(A), посредством воды 1201 и 1203 взаимодействующий с азотистыми основаниями аденином (10) и гуанином (11) цепи C.
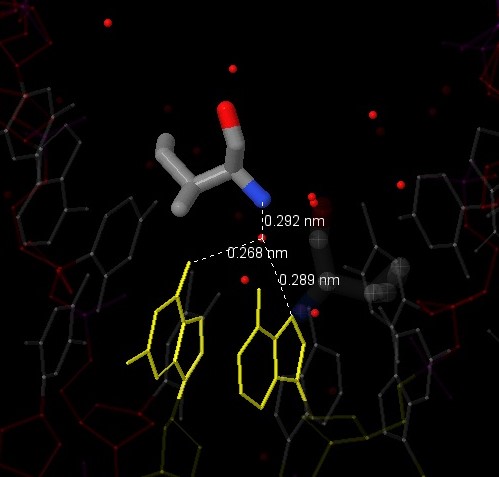
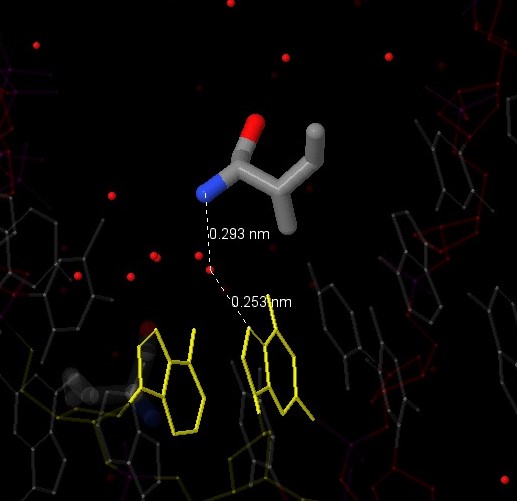
Нам кажется, что именно этот остаток (а также ILE79 на других цепях) наиболее вероятно помогает определить последовательность ДНК: он вступает в глубокое взаимодействие с азотистыми основаниями вблизи 3'-конца. На рисунке белым цветом показана локализация остатка Ile79(C) в белке и относительно ДНК.