Практикум 4. Секвенирование по Сэнгеру
Задание 1
Смысл практикума заключался в обработке результатов секвенирования по Сенгеру и получении КОНСЕСНСУСНОЙ последовательности. Для этого требовалось открыть в программе визуализации обработки хроматограмм, полученных при секвенировании прямой и обратной цепей участка последовательности ДНК. Мы пользовались программой GeneStudio.
Файл с прямой цепью ДНК - 45_F.ab1, с обратной - 45_R.ab1.
В среднем отношение сил сигнала и шума составляет, по нашему мнению, 4:1. При этом максимальный уровень шума наблюдается в начале и в конце хроматограммы (примерно 2:1). Наиболее высокие и наиболее низкие сигнальные пики наблюдались у пуриновых онований, в то время как высота пиков пиримидиновах оснований оставалась на примерно одном уровне.
При просмотре хроматограммы прямой цепи обнаружилась непрочтенная область с 5'-конца длиной 23 основания (рис.1). Отыскались некоторые проблемные нуклеотиды, при внимательном просмотре хроматограммы, однако, они однозначно определялись. Также нашлись некоторые полиморфизмы, идущие по счету 261 (r), 386(r)(рис.2), 451(r), 471(w), 588(r), 675(r), 687(y), 693(r). В основном они представляют собой дуализмы a/g или t/c. Скорее всего, это связано с некоторой вероятностью реакции хроматограммы на основания одной природы (пуриновые или пиримидиновые), а не просто на какое-то конкретное основание, из-за чего возникают неопределённости и ошибки интерпретации сигнала.
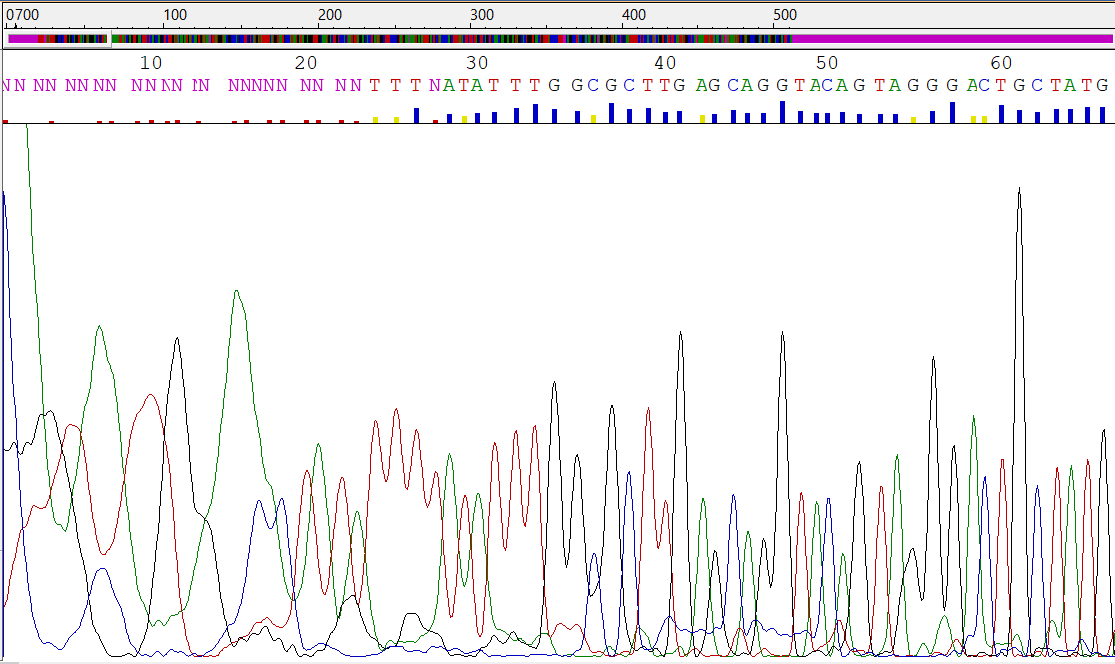
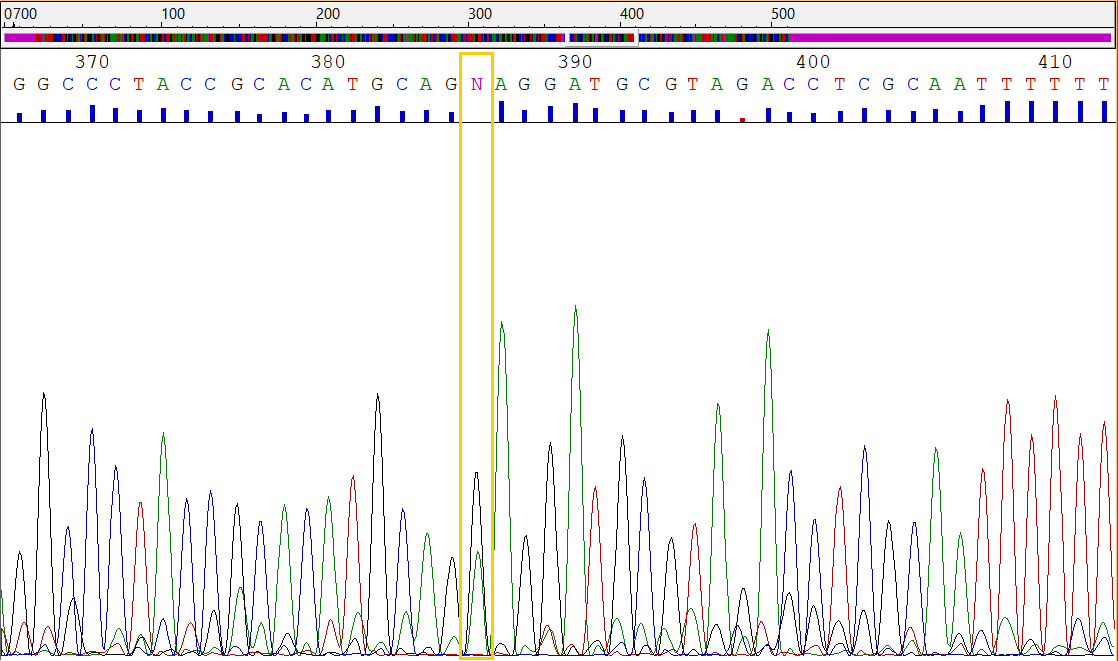
Просмотр хроматограммы обратной цепи выявил непроченную область на 3'-конце длиной 20 нуклеотидов. Полиморфизмы встречаются реже, чем на прямой цепи, например 678(y). В большем количестве были обнаружены проблемные нуклеотиды (пример - рис.3).
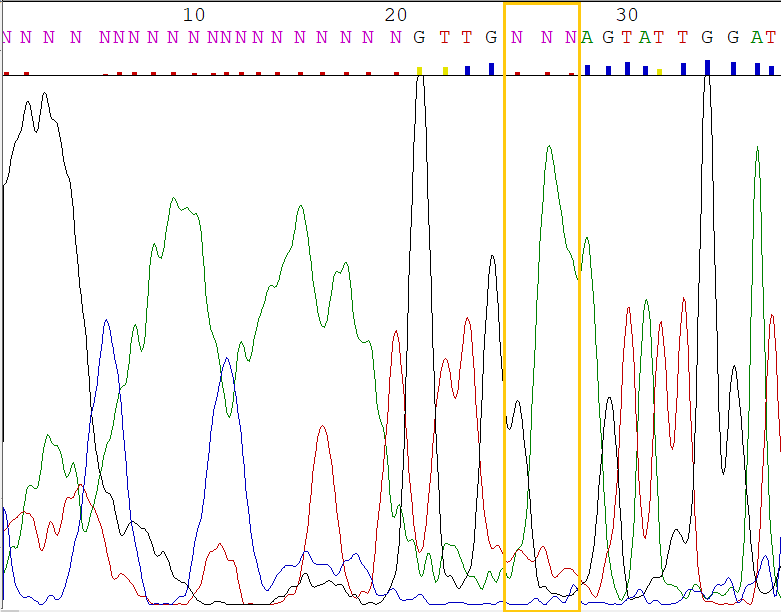
Восстановленные таким образом последовательности:
Задание 2
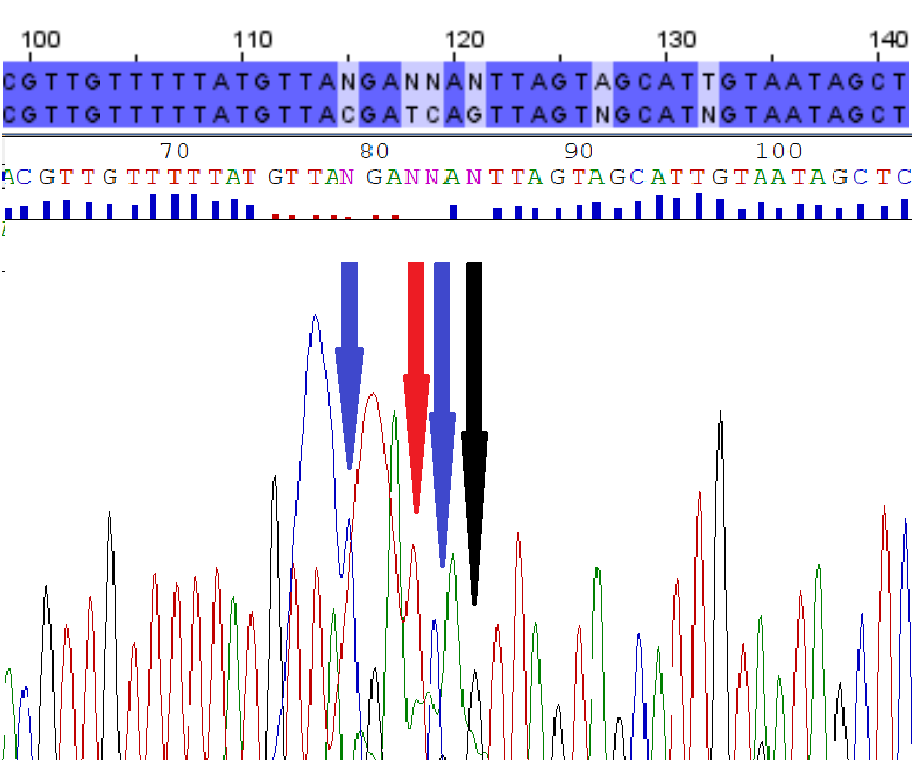
Далее требовалось выравнять последовательности прямой и обратной цепей. Мы воспользовались методами программы Jalview (так было привычнее). Для начала для обеих цепей были получены обратные к ним цепи (то есть 45_F|revcomp - это сгенерированная программой Jalview обратная комплементарная цепь к прямой цеии, а 45_R|revcomp - к обратной цепи). Затем были проведены 2 выравнивания: 45_F с 45_R|revcomp и 45_R с 45_F|revcomp. На основании первого выравнивания мы вручную восстановили последовательность прямой цепи (в местах, где на прямой цепи стоит неопределенный нуклеотид, а на 45_R|revcomp нуклеотид определен). Точно так же восстанавливалась последовательность обратной цепи (для примера - основания под номерами 115, 118, 119 и 121 в выравнивании определились нами как соответственно C, T, C и G). С помощью выравниваний мы подтвердили наличие и длины плохо прочтенных областей 5'-конца прямой и 3'-конца обратной цепей, а также определили плохо прочтенную область 3'-конца прямой (55 нуклеотидов) и, соответственно, 5'-конца обратной цепи (52 нуклеотида). Последовательность, полученную с помощью данных выравнивания, мы считали и считали консенсусной.
Файлы с выравниваниями:
![]()
![]()
Решения для проблемных нуклеотидов обратной цепи:
КОНСЕНСУСНАЯ последовательность, полученная с помощью данных выравнивания и восстановленная на нечитаемых концах:








