Практикум 6. Мотивы и профили (часть 2)
Задача, поставленная перед нами, заключалась в поиске высоко консервативных последовательностей TRS в геноме коронавируса. Для исследования мы выбрали Middle East respiratory syndrome-related coronavirus - коронавирус, впервые описанный в 2012 году, вызывающий респираторный синдром с высокой фатальностью. Провели исследование для двух вариантов генома коронавируса - штамма England 1 и референсного генома HCoV-EMC.
Штамм England 1
С сайта NCBI были скачаны файлы .gb с аннотацией и координатами генов и .fasta геномной последовательности RefSeq.
Далее для каждого из 9 генов были выбраны координаты upstream regions, которые определялись так: для первого гена полипротеина orf1ab - 1 основание в референсной последовательности до -1 основания от ORF, для остальных генов (поздних) - 101 нуклеотид до ORF ([-101:-1]). Программой seqret пакета EMBOSS выделили последовательности upstream regions.
Для поиска мотивов была выполнена команда:
meme all_MERS_us.fasta -oc dir/ -dna -nmotifs 3 -minw 6
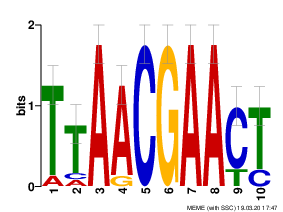
На выходе были получены файлы с отчетом в форматах .html и .txt и диаграммы LOGO в виде картинок .png. При просмотре отчета оказалось, что первый мотив найден во всех upstream regions, и он содержит 5 консервативных позиций при длине 10. E-value для последовательностей порядка E-5 - E-6, для мотива 7.4e-011. LOGO говорит о высоком информационном содержании мотива. MEME выдает IC 16.5 бит.
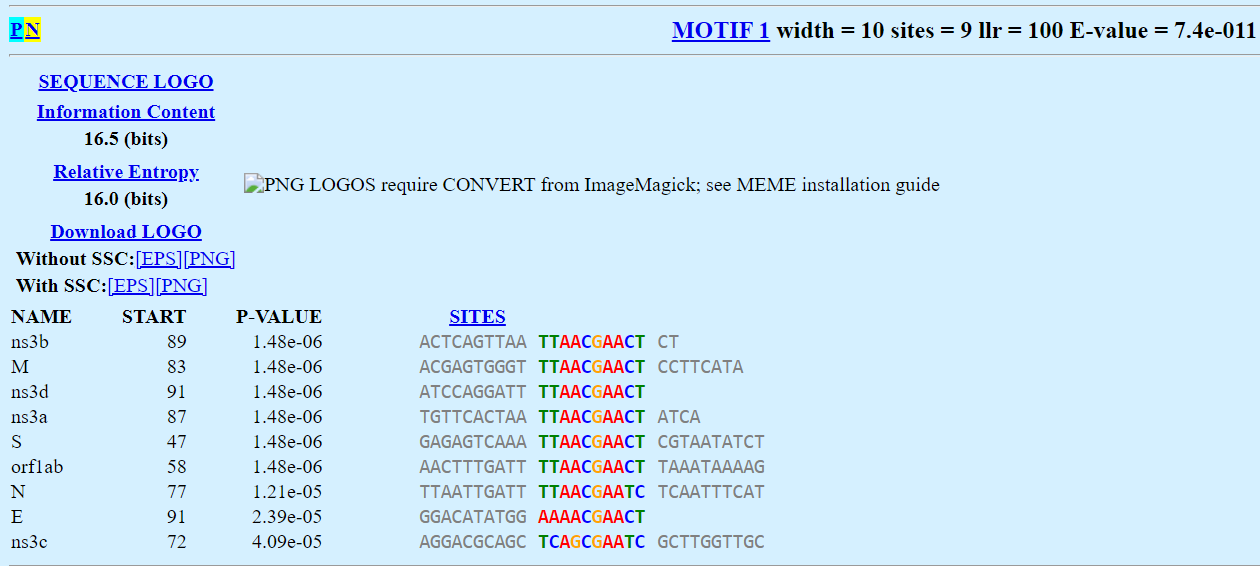
Второй мотив был найден в 4 последовательностях, причем 9/24 позиций были консервативны, E-value для каждой последовательности порядка E-10, а для мотива 6.0e+001. IC мотива 30 бит.
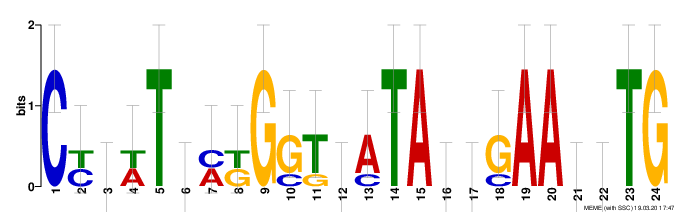
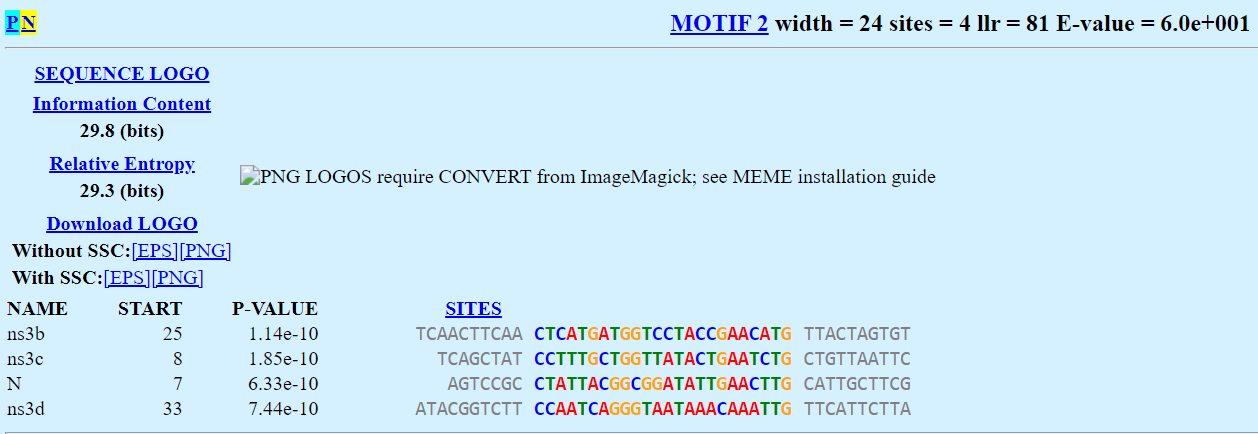
Третий мотив найден в четырех последовательностях, в нем 6/11 консервативных позиций, E-value последовательностей порядка E-6, мотива 2.1e+002. IC 17.8 бит.
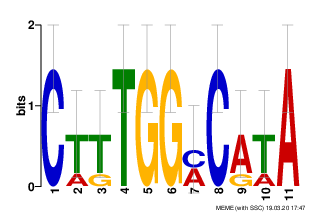

MEME выдает расположение мотива в последовательностях. Высчитано комбинированное p-value: чем меньше, тем наверняка менее случайна последовательность, а значит наверняка она содержит много интересных мотивов. Больше всего интересных мотивов в upstream regions для генов ns3b, ns3c, ns3d, N. Заметно, что для 7/9 1 мотив находится в непосредственной близости к ORF последующего гена. Можно предположить, что мотив является очень интересным сайтом. 2 мотив интересен тем, что там, где он был найден (ns3b,ns3c, ns3d, N), он имеет достаточно жесткую позицию относительно мотива 1, примерно на 35-40 оснований вверх по течению. Расположение мотива 3 менее регулярно.
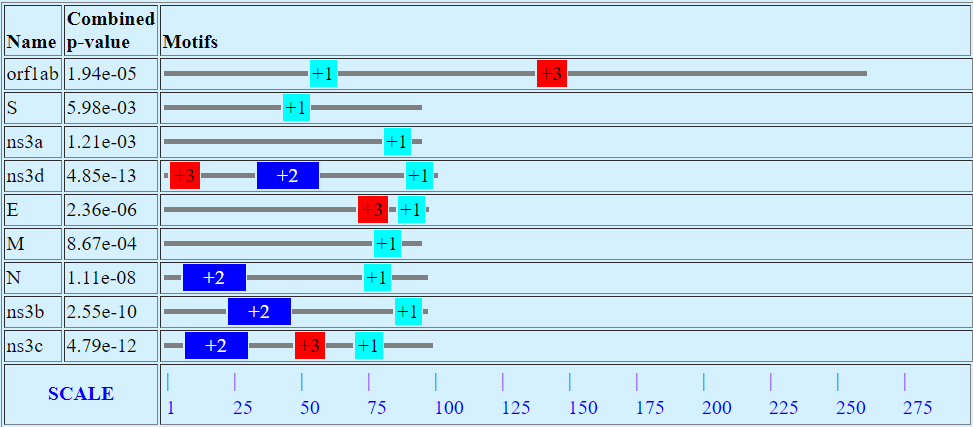
При других значениях длин фрагментов (не 101, а 151 и 71) мотивы 2 и 3 терялись в некоторых последовательностях совсем и находились совсем другие мотивы. А вот 1 мотив оставался при любых длинах фрагментов.
|
Name |
Combined p-value |
Motifs |
||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| orf1ab | 1.94e-05 |
|
||||||||||||||||||||||||
| S | 5.98e-03 |
|
||||||||||||||||||||||||
| ns3a | 1.21e-03 |
|
||||||||||||||||||||||||
| ns3d | 4.85e-13 |
|
||||||||||||||||||||||||
| E | 2.36e-06 |
|
||||||||||||||||||||||||
| M | 8.67e-04 |
|
||||||||||||||||||||||||
| N | 1.11e-08 |
|
||||||||||||||||||||||||
| ns3b | 2.55e-10 |
|
||||||||||||||||||||||||
| ns3c | 4.79e-12 |
|
||||||||||||||||||||||||
| SCALE |
| |||||||||||||||||||||||||
HCoV-EMC
Те же действия мы повторили для другого штамма (Human betacoronavirus 2c EMC/2012), незначительно отличающегося от первого:
| 1 штамм | 2 штамм |
|---|---|
| orf1ab | orf1ab |
| S | S |
| NS3A | NS3 |
| NS3B | NS4A |
| NS3C | NS4B |
| NS3D | NS5 |
| E | E |
| M | M |
| N | N |
| - | orf8b |
Сначала мы вырезали из последовательности генома вируса последовательности:
$ cat sequence2.txt | cut -c 1-279 > orf1ab.fasta $ cat sequence2.txt | cut -c 21355-21456 > S.fasta $ cat sequence2.txt | cut -c 25431-25532 > NS3.fasta $ cat sequence2.txt | cut -c 25751-25852 > NS4A.fasta $ cat sequence2.txt | cut -c 25992-26093 > NS4B.fasta $ cat sequence2.txt | cut -c 26739-26840 > NS5.fasta $ cat sequence2.txt | cut -c 27489-27590 > E.fasta $ cat sequence2.txt | cut -c 27752-27853 > M.fasta $ cat sequence2.txt | cut -c 28465-28566 > N.fasta $ cat sequence2.txt | cut -c 28661-28761 > orf8b.fasta
Далее был создан файл mylist c названиями файлов, файлы были слиты в один командой seqret @mylist all.fasta.
Meme был запущен командой meme all.fasta -dna -nmotifs 3 -minw 6 -maxw N -dna ememe -snucleotide1.
Получены 3 мотива:

Первый мотив был найден во всех последовательностях, E-value = 9.8e-010.
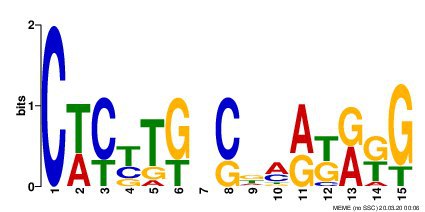
Второй мотив был найден во всех последовательностях, E-value = 6.6e+001.
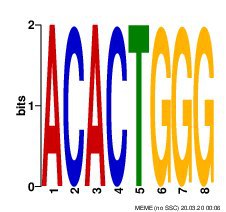
Третий мотив был найден лишь в 2 последовательностях, E-value = 1.2e+003.
Сравнение и обобщение
Первый мотив 2-ого штамма был схож с мотивом, полученным для 1-ого штамма, другие два значительно отличались, В первом мотиве была найдена искомая высококонсервативная последовательность CS (Core Sequence). Core Sequence — это последовательность из 6 нуклеотидов, обнаруженная в сигналах TRS-L (TRS от Transcription-Regulating Sequence) и TRS-B. TRS-L и TRS-B являются сигналами перестройки РНК для получения мРНК поздних генов. Есть 2 варианта CS: 5′-CUAAAC и 5′-ACGAAC [1]. В первом мотиве обоих штаммов видна последовательность 5′-ACGAAC.
Кто выполнил работу
Елена Птицына выполнила поиск мотивов в геноме HCoV-EMC и выяснила факт принадлежности найденного в двух геномах мотива классу TRS.
Зайцева Елизавета выполнила поиск мотивов в геноме штамма England 1 и разместила отчет о работе на своей страничке.
Использованные материалы
[1] "Coronavirus N Protein N-Terminal Domain (NTD) Specifically Binds the Transcriptional Regulatory Sequence (TRS) and Melts TRS-cTRS RNA Duplexes." Nicholas E.Grossoehme, Lichun Li, Sarah C.Keane, Pinghua Liu, Charles E.Dann, Julian L.Leibowitz, David P.Giedroc. Journal of Molecular Biology, Volume 394, Issue 3, 4 December 2009, Pages 544-557
