Построение парных выравниваний. Поиск по сходству
Создание выборки гомологов белка декстрансукразы L. citreum KM20
Для нахождения гомологов анализируемого белка использовалась программа protein BLAST. Некоторые настройки были изменены для лучшего представления результатов, сами настройки можно посмотреть здесь (файл с сохранёнными настройками).
Так как программа выдавала много потенциальных последовательностей-гомологов (12.395), некоторые настройки были изменены, например бактерии рода Streptococcus не показываются. Из такой небольшой выборки белков (229), белки с покрытием >80% нашлись, причём довольно большое количество, - 89. Единственное, что стоит отметить, - это то, что некоторые белки 'повторяются', так как до сих пор в базах данных остались данные о декстрансукразе, как о гликозил гидролазе, поэтому первые результаты с покрытием в 99% и идентичностью в 99% в той же бактерии, L. citreum, по сути являются старыми артефактами, которые не были удалены.
| Таблица 1. Краткое сравнение белков | |||||
|---|---|---|---|---|---|
| Организм | Длина выравнивания | Bit score | Процент идентичных/сходных остатков, % | E-value | Выравнивание |
| Leuconostoc gelidum | 1791 | 3661 bits(9493) | 99/99 | 0.0 | Лучшее |
| Leuconostoc fallax | 1163 | 1077 bits(2785) | 51/63 | 0.0 | Случайное |
| Oenococcus oeni | 1040 | 859 bits(2220) | 47/61 | 0.0 | Случайное |
| Weissella cibaria | 203 | 32.3 bits(72) | 25/37 | 7.3 | Худшее |
Множественное выравнивание 24 последовательностей
Для выполнения этого задания были взяты 24 последовательности. В этом fasta-файле лежат все последовательности. Сам jvp-проект находится здесь. Блоки отмечены 'B'. На N- и C-концах были длинные невыровненные участки, которые впоследствии были вырезаны, осталась часть с консервативными колонками, блоками. Наличие гэпов на N- и C-концах никак не говорит о том, что последовательности не гомологичны.
Глобальное и локальное выравнивания
Чтобы выполнить это задание, нужно было воспользоваться программами Needle и Water через сервер Kodomo с помощью Putty. Входные файлы: две последовательности в fasta-формате, величины штрафов за первый гэп (10.0) и за продление гэпа (0.5), также имя выходного файла.
Отличия Needle и Water:
Использованные последовательности: WP_041761905.1 (мой белок) и WP_063083270.1.
Глобальное парное выравнивание, построенное с помощью Needle

Глобальное парное выравнивание, вырезанное из множественного

Локальное парное выравнивание, построенное с помощью Water

Локальное парное выравнивание, построенное с помощью Blast
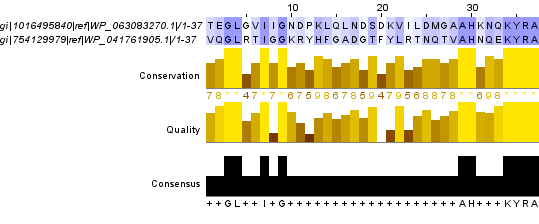
4 выравнивания, объединённых в группы

{ссылка на jalview-проект с 4 окнами, выравниваниями}
{ссылка на jalview-проект с объединёнными выравниваниями}
Выравнивание полученных выравниваний
Участки, найденные программами BLAST и Water совпадают почти полностью. Также на рис. 1 представлен участок, на котором выравнивания различаются.

 |
{ссылка на jalview-проект с выровненными выравниваниями}
Парные выравнивания двух заведомо негомологичных белков
Для выполнения этого задания были взяты два белка: NP_953118.2 и WP_041761905.1.
Ниже представлены выравнивания заведомо негомологичных белков с использованием программ Water и Needle. Видно, что выравнивания, полученные 2 программами в целом совпадают. Что касается отдельных выравниваний, можно сказать, что совпадения нашлись, но как таковые блоки отсутствуют. Иными словами полученные данные в результате выравниваний не информативны.

{ссылка на jalview-проект с выравниванием заведомо негомологичных поледовательностей}
Финальный Jalview-проект
{cсылку на jalview-проект с тремя окнами (множественное выравнивание, парные выравнивания моего белка с гомологом, парные выравнивания моего белка с белком, заведомо негомологичным ему)}
⌘
© Emir Radkevich, 2016