Uniprot и Refseq protein
Почему Refseq ID декстрансукразы не работает?
В прошлом семестре я столкнулся с проблемой касательно ID моего белка. Полностью я описал её уже тут. Единственное, что хотелось бы добавить, - это то, как же я искал свой белок в Uniprot. Зайдя на сайт NCBI, узнал новый ID для декстрансукразы (WP_041761905.1), однако, такого ID Uniprot не распознал, поэтому пришлось поискать с помощью названия фермента (декстрансукраза). Непонятно, почему сайт не узнаёт этот ID, однако даже в строчке DR нет строчки с названием Refseq.
Забегая вперёд, напишу, что строчка с PDB ID также отсутствует (см. табл. 1).
В таблице 1 показано количество белков в кластерах Uniref50, Uniref90, Uniref100. Сами эти кластеры
представляют собой группу белков, объединённых по своей схожести. То есть в кластере Uniref100 содержится 1 белок,
потому что 100%-схожий белок с анализируемым может быть только один, - сам белок (как правило). Что касается
кластеров Uniref90 и Uniref50, ситуация аналогична.
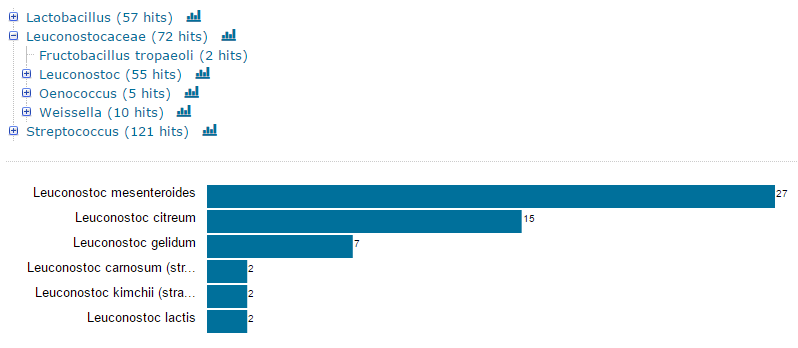
Полезно будет показать, какие организмы эволюционно ближе всего к Leuconostoc citreum KM20, если сравнивать последовательность
только этого белка (см. рис. 1). Результаты ожидаемые: род Leuconostoc оказался самым близкородственным, несмотря на то, что совпадений больше у рода Streptococcus.
Также, совпадение последовательности >90% характено только для Leuconostoc mesenteroides, причём декстрансукраза Leuconostoc citreum совпадает как с нативной,
так и с мутантной формой фермента Leuconostoc mesenteroides.
Refseq ID не найден, задание 4 выполнить со своми белком не могу. Поэтому я решил взять белок однокурсницы, чтобы посмотреть, чем отличаются записи RefSeq и Uniprot. В результате можно сказать, что информация, представленная RefSeq, скуднее, чем Uniprot. Например в записи RefSeq отсутсвует молеклярная масса белка, описание его функций, ссылки на ID в других базах данных. Однако, присутствует сама последовательность белка, информация о сайтах связывания (в features), некоторые комментарии.
Для просмотра изменения истории взял свой белок. Всего насчитывается 52 версии. При сравнивании близких версий (50 и 52 например) можно заметить небольшие изменения, однако, если сравнить 52 и 1 версии, то различия уже куда значительнее. Такие строчки как DE, OS, RA, OC, GN и многие другие в ранней версии отсутствуют. Если сравнить версии, разница между которыми менее 3 лёт, критичных пробелов не обнаруживается, разве что добавляются некоторые идентификаторы DR.
В прошлом семестре я столкнулся с проблемой касательно ID моего белка. Полностью я описал её уже тут. Единственное, что хотелось бы добавить, - это то, как же я искал свой белок в Uniprot. Зайдя на сайт NCBI, узнал новый ID для декстрансукразы (WP_041761905.1), однако, такого ID Uniprot не распознал, поэтому пришлось поискать с помощью названия фермента (декстрансукраза). Непонятно, почему сайт не узнаёт этот ID, однако даже в строчке DR нет строчки с названием Refseq.
Забегая вперёд, напишу, что строчка с PDB ID также отсутствует (см. табл. 1).
| Таблица 1. Информация о белке WP_041761905.1 [1] | |||
|---|---|---|---|
| Общая информация | Информация о Uniref-кластерах | ||
| Uniprot ID | B1MZT6_LEUCK | Uniref-100 | |
| Uniprot AC | B1MZT6 | ID кластера | UniRef100_B1MZT6 |
| Refseq ID | - | Количество белков | 1 |
| PDB ID | - | Uniref-90 | |
| Длина, AA | 1791 | ID кластера | UniRef90_B1MZT6 |
| Молекулярная масса, Da | 199.986 | Количество белков | 1 |
| Рекомендованное название | Dextransucrase (дектсрансукраза) | Uniref-50 | |
| Последняя дата изменения | 16.03.2016 | ID кластера | UniRef50_B1MZT6 |
| Локус | LCK_01211 | Количество белков | 1 |
 |
| Таблица 2. Результаты поиска | |
|---|---|
| Критерий поиска | Количество белков |
| Поиск по RecName (1) | 148 |
| Поиск по RecName + family (2) | 53 |
| Поиск по RecName + phylum (3) | 136 |
| Поиск по RecName + названия (4) | 4 |
| Поиск по RecName + трипсин (5) | 11.159 |
| Поиск по RecName + ингиботоры трипсина (6) | 2.429 |
- Использованные команды:
- (1) - {name:dextransucrase}
- (2) - {name:dextransucrase taxonomy:"Leuconostocaceae [81850]"}
- (3) - {name:dextransucrase taxonomy:"Firmicutes [1239]"}
- (4) - {name:myoglobin taxonomy:"Ciliophora [5878]"} - 3 и {name:myoglobin taxonomy:"Fungi [4751]"} - 1
- (5) - {name:trypsin}
- (6) - {name:"trypsin inhibitor"}
| Таблица 3. Информация о найденных белках | |||
|---|---|---|---|
| Номер поиска | Общее количество белков | Количество аннотированных белков | Количество неаннотированных белков |
| (1) | 148 | 10 | 138 |
| (2) | 53 | 4 | 49 |
| (3) | 136 | 10 | 126 |
| (4) | 4 | 3 | 1 |
| (5) | 11.159 | 301 | 10.858 |
| (6) | 2.429 | 191 | 2.238 |
Refseq ID не найден, задание 4 выполнить со своми белком не могу. Поэтому я решил взять белок однокурсницы, чтобы посмотреть, чем отличаются записи RefSeq и Uniprot. В результате можно сказать, что информация, представленная RefSeq, скуднее, чем Uniprot. Например в записи RefSeq отсутсвует молеклярная масса белка, описание его функций, ссылки на ID в других базах данных. Однако, присутствует сама последовательность белка, информация о сайтах связывания (в features), некоторые комментарии.
Для просмотра изменения истории взял свой белок. Всего насчитывается 52 версии. При сравнивании близких версий (50 и 52 например) можно заметить небольшие изменения, однако, если сравнить 52 и 1 версии, то различия уже куда значительнее. Такие строчки как DE, OS, RA, OC, GN и многие другие в ранней версии отсутствуют. Если сравнить версии, разница между которыми менее 3 лёт, критичных пробелов не обнаруживается, разве что добавляются некоторые идентификаторы DR.
- Как обозначаются различные отхождения в химической структуре на Uniprot
- Нестандартные аминокислоты обозначаются 'NON_STD' в строчке 'FT'. Селеноцистеин принято обоначать буквой 'U', пирролизин - 'O'.
- Фосфорилирование отображается в формате 'text' в строчке 'FT', как 'MOD_RES...Phospho{name of amino acid}'. Чаще всего фософрилируются S, T, Y.
- Дисульфидные мостики обозначаются как 'DISULFID' в строчке 'FT'
⌘
© Emir Radkevich, 2016