Изображение выравниваний
Откуда брал файлы и что с ними делал изначально
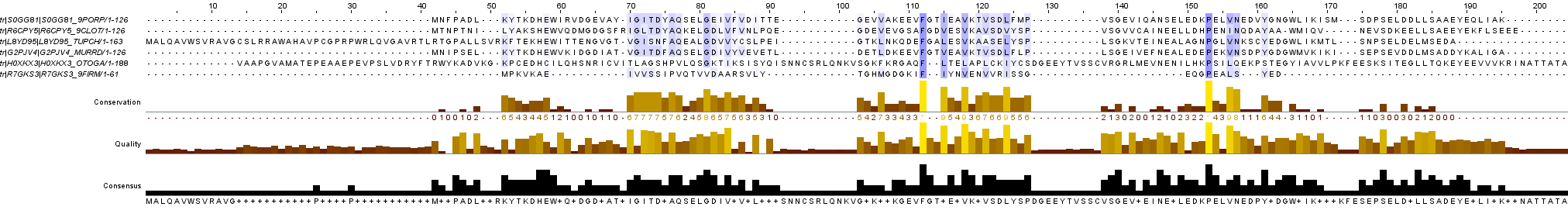
Было дано 6 исходных идентификаторов белков, которые нужно было скачать в Fasta-формате. Это можно было сделать несколькими путями, я выбрал самый простой: добавил в корзину все 6 идентификаторов, а потом скачал одним файлом в Fasta-формате (на самом деле ниже я напишу, почему выбрал такой путь скачивания файлов). После установки нужной программы (JalView) и скачивания Fasta-файла я загрузил его и получил на выход то, что видно на рисунке слева (так как файл плохо видно на самой страничке, лучше открыть в новом окне и при помощи зума уже анализировать). На N- и C-концах имеются гэпы (см. рис. ниже), которые точно можно удалить, потому что не играют никакой роли в анализe выравнивания (это также можно понять, если попробовать раскрасить выравнивания со значением порога консервативности в 0%). С помощью последовательности действий Web Service - Alignment - Muscle with Defaults строим множественные выравнивания.
Подытог выравнивания
После построения множественных выравниваний и раскрашивания с помощью разных методов (BLOSUM62 и ClustalX), замечаем, что одна последовательность явно выбивается из общего ряда: HOXKX3_OTOGA (неизвестный белок Otolemur, примата). На самом деле под вопросом был ещё один белок, который достаточно сильно отличался от остальных, однако при выборе порога консервативности в 30%, наиболее консервативные области красились и у этого белка.
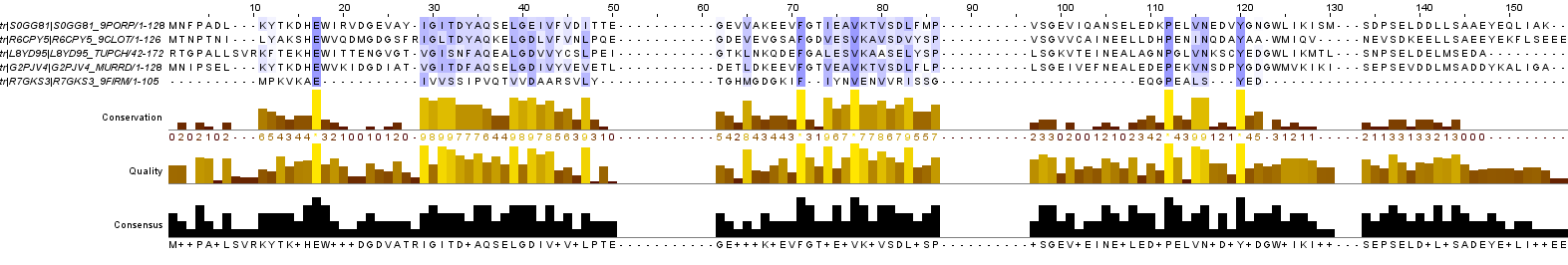
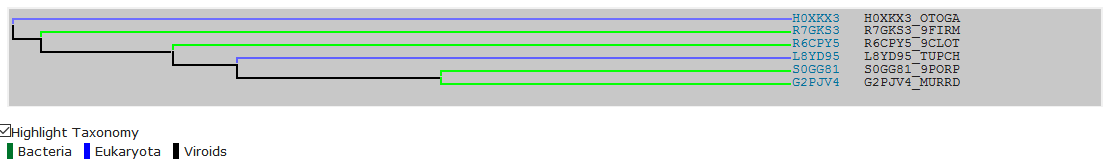
Как и писал выше, я не случайно выбрал такой путь скачивания Fasta-файла. При внимательном изучении результатов выравнивания, было достаточно сложно понять, считать ли большое количесто гэпов у белка R7GKS3_9FIRM "отклонением", и как следствие "лишним"? Для решения этой проблемы я воспользовался Tree construction на сайте Uniprot, где и получил убедительный и окончательный ответ. Оказывается, что белок с большим количеством гэпов является азот-регуляторным белком, в то время как 4 белка являются белками, ответственными за расщепление глицина, а один белок представляет собой белок с неизвестной функцией. Итак, давайте запишем всё в табличку:
| Таблица 1. Идентификаторы белков, общая информация | |||
|---|---|---|---|
| Идентификатор белка | Название организма | Группа организмов | Функция белка |
| HOXKX3_OTOGA | Otolemur garnettii | Animalia | Неизвестная функция |
| R7GKS3_9FIRM | Catenibacterium sp. | Bactreia | Азот-регуляция |
| R6CPY5_9CLOT | Clostridium sp. | Bactreia | Расщепление глицина |
| L8YD95_TUPCH | Tupaia chinensis | Animalia | Расщепление глицина в митохондрии |
| SOGG81_9PORP | Parabacteroides goldsteinii | Bactreria | Расщепление глицина |
| G2PJV4_MURRD | Muricauda ruestringensis | Bacteria | Расщепление глицина |
 |
Необходимые файлы, которые получились в ходе работы
ID "лишней" последовательности: HOXKX3_OTOGA
{ссылка на 6 последовательностей в Fasta-формате}
{ссылка на выравнивание, полученное во 2 задании в формате msf}
{ссылка на выравнивание, полученное во 2 задании в формате fasta}
{ссылка на проект JalView}
*Файлы msf и fasta будут не раскрашены, потому что сам формат файла не предполагает сохранение таких опций как раскраска.
⌘
© Emir Radkevich, 2016