Комплексы ДНК-белок
Предсказание вторичной структуры заданной тРНК
Ниже представлена таблица, в которой можно увидеть сравнение реальной и предсказанной вторичной структуры тРНК (для предсказания использовались программа einverted и алгоритм Зукера).
*Программа einverted запрашивала значения множетсва параметров, однако параметр, который наиболее сильно влиял на предсказание вторичной структуры, - Minimum score treshold. Стандартное значение для этого параметра - 50. После ввода 50 в качестве значения параметра, выводом оказался пустой файл, что неудивительно, есил вспомнить, что программа изначально работает с ДНК последовательностями, которые как правило длиннее, чем РНК последовательности. Собственно поэтому нужно было изменить это значение [50] в меньшую сторону, причем значительно. В итоге максимальным порогом стало значение 14, при котором предсказание было таким же, как и при 0.
| Таблица 1. Реальная и предсказанная вторичная структура тРНК из файла 1I9V.pdb | |||
|---|---|---|---|
| Участок структуры тРНК | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'- 1-7 - 3' 5' - 66-72 - 3' Всего 7 пар | Предсказано 0 из 7 пар | Предсказано 7 из 7 пар |
| D-стебель | 5' - 10-13 - 3' 5' - 22-25 - 3' Всего 4 пары | Предсказано 0 из 4 пар | Предсказано 4 из 4 пар |
| T-стебель | 5' - 49-53 - 3' 5' - 61-65 - 3' Всего 5 пар | Предсказано 5 пар из 5. Начало и конец совпадают абсолютно | Предсказано 4 из 5 пар. Начало и конец не совпадают, смещение на одну пару |
| Антикодоновый стебель | 5' - 40-44 - 3' 5' - 26-30 - 3' Всего 5 пар | Предсказано 5 пар из 5. Начало и конец не совпадают, предсказано 5 лишних пар | Предсказано 5 из 5 пар |
| Общее число канонических пар оснований | 23 | 13 | 23 |
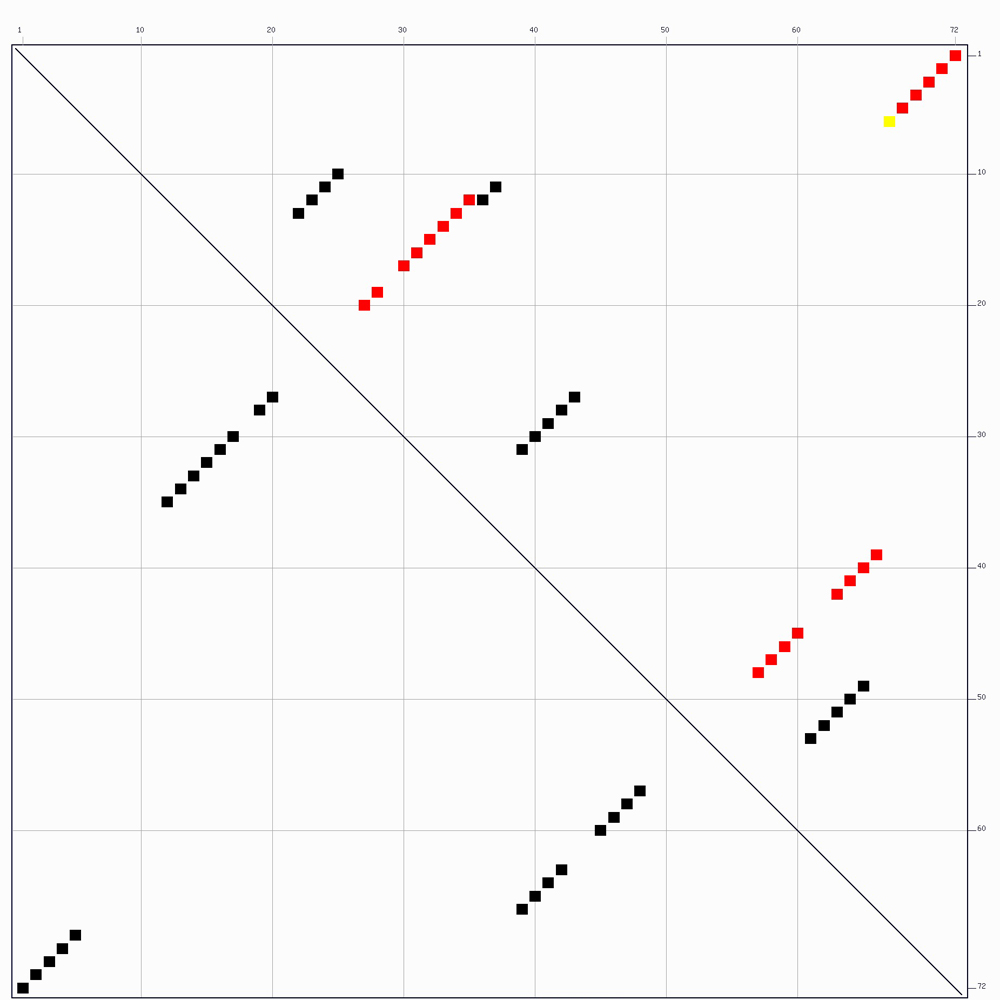 |
Рис. 1. Построенная тРНК, согласно предсказанию алгоритма Зукера. (картинка слева)
*Красным кругом отмечены нуклеотиды, которые были неправильно идентифицированы как пара оснований, а синим - нуклеотиды, которые были идентифицированы алгоритмом как неспаренные нуклеотиды.
Рис. 2. Точечный (квадраты) график, который отражает вероятность спаривания нуклеотидов (построено программой mfold). (картинка справа)
*Разными цветами обозначены разные значения энергии спаривания нуклеотидов (красный цвет обозначает оптимальную энергию (-22.9 ccal/mol), черный - энергию, отклоняющуюся на 0.9 ccal/mol в бОльшую сторону). На графике видно, что имеет место ошибка, так как например 1 и 72 нуклеотиды не образуют пару, хотя согласно этому графику могут (но с малой энергией!).
Поиск ДНК-белковых контактов в заданной структуре
|
|
Перед тем как нажать на кнопку, нажмите на картинку, чтобы запустить PDB файл! Нажимать после запуска апплета (первая кнопка - запуск скрипта, вторая - продолжение скрипта): Требовалось визуализировать следующие сеты: Set1: множество атомов кислорода 2'-дезоксирибозы; Set2: множество атомов кислорода в остатке фосфорной кислоты; Set3: множество атомов азота в азотистых основаниях {Ссылка на 1 скрипт} {Ссылка на 2 скрипт} *Действие 2 скрипта в апплете не показано. |
| Таблица 2. Контакты разного типа в комплексе 1MDM.pdb | |||
|---|---|---|---|
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 3 | 13 | 16 |
| остатками фосфорной кислоты | 9 | 18 | 27 |
| остатками азотистых оснований со стороны большой бороздки | 4 | 11 | 15 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 2 | 2 |
Согласно данным таблицы 2 видно, что белок контактирует с остовом ДНК чаще, чем с остатками азотистых оснований. Это можно объяснить специфическим пространственным расположением взаимодействующих участков ДНК. Можно также заметить, что неполярных контактов больше, чем полярных.
С помощью программы nucplot была составлена следующая популярная схема ДНК-белкового взаимодействия:
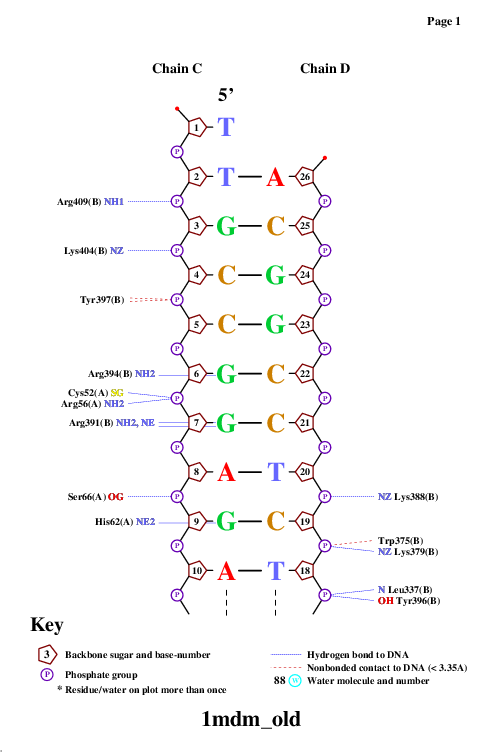
На картинке видно, что наибольшее количество связей образует tyr397. В качестве распознавателя, возьмем аминокислоту, которая непосредственно связывается с азотистым основанием, то есть arg394.
*На самом деле в выводе было 3 изображения, однако 2 из них приходились на другие цепи белка.
 |
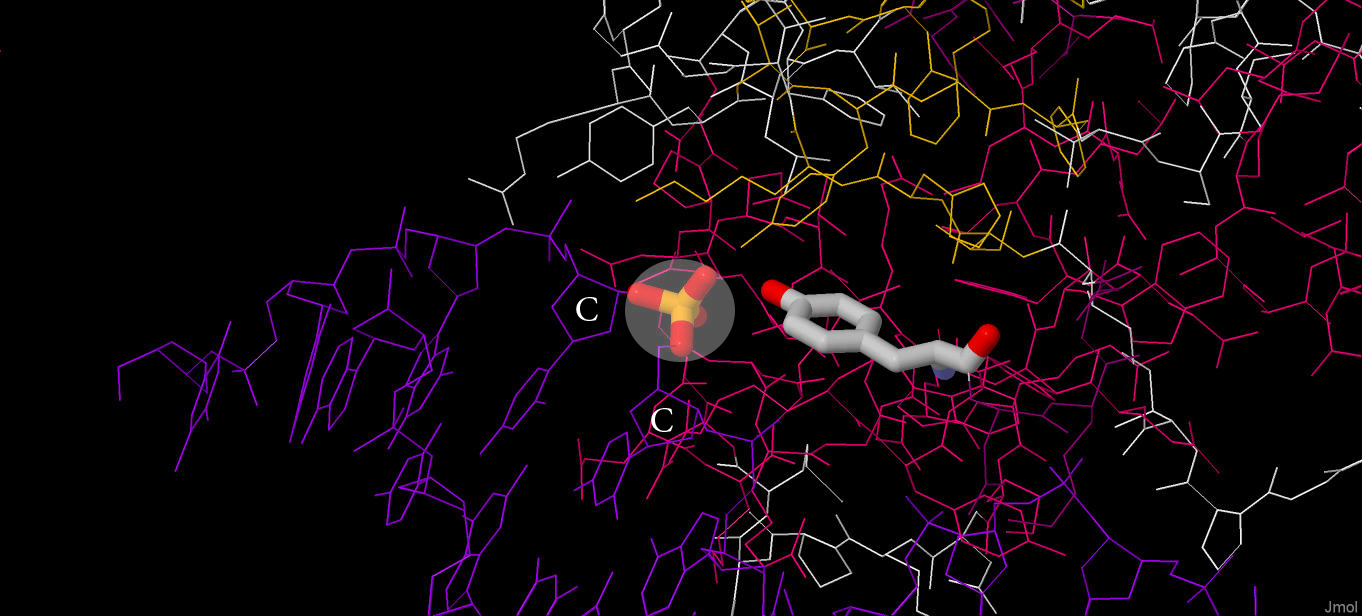 |
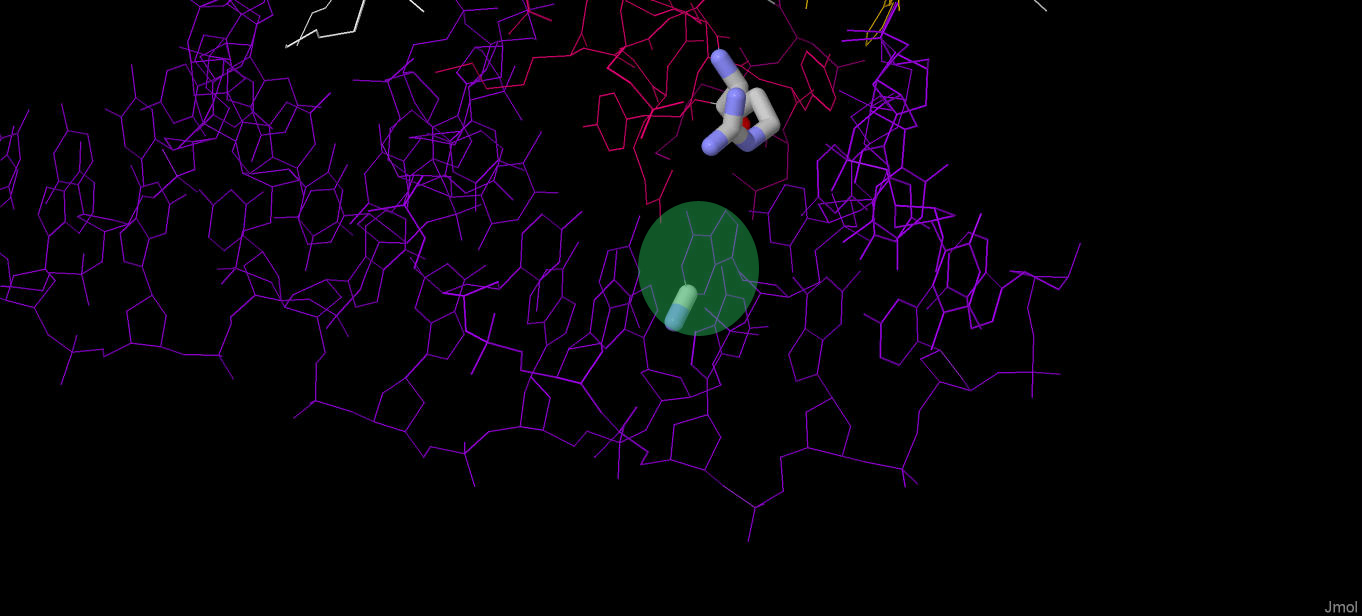
Рис. 3. Связывание arg394 с N2 гуанина. (картинка сверху)
*В зелёный кружок взят гуанин, который как раз и участвует в образовании контакта с аргинином.
Рис. 4. Связывание tyr397 с фосфатом. (картинка снизу)
*В белый круг взят фосфат, который участвует в образовании контактов с тирозином. Также, белыми буквами обозначены соседние нуклеотиды, соединенные этим фосфатом.
Изображения были получены с помощью этого скрипта, а также этого.
⌘
© Emir Radkevich, 2016