Филогенетическое дерево
Сравнение различных методов построения деревьев алгоритмом Neighbor-joining
Сравнивались три метода в программе Jalview и собственно Neighbor-joining в MEGA. Результаты приведены ниже в таблице 1, а также под таблицей приведено изображение реального дерева для сравнения:
| Таблица 1. Деревья, построенные разными методами Neighbor-joining | |
|---|---|
| Метод | Картинка из Jalview/MEGA |
| Neighbor-joining using % Identity | 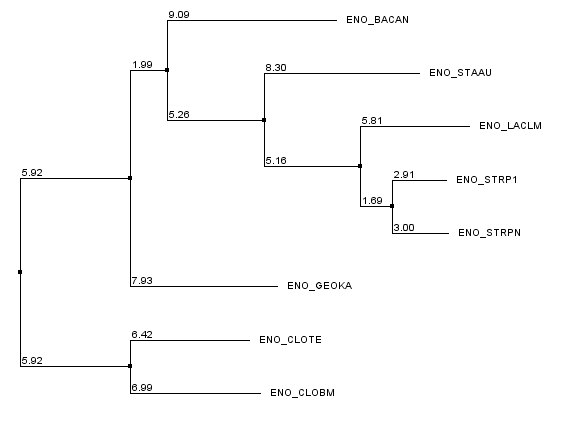 |
| Neighbor-joining using BLOSUM62 | 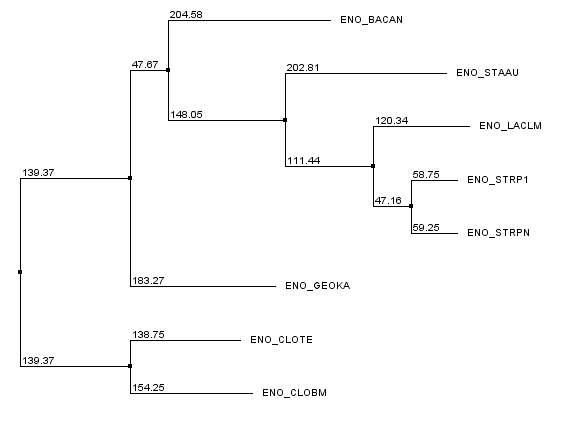 |
| Neighbor-joining using PAM 250 | 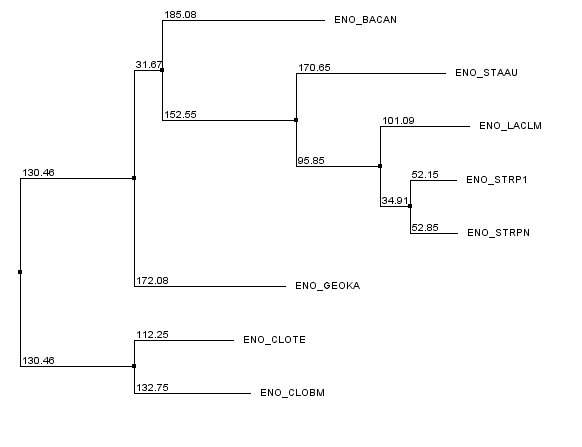 |
| Neighbor-joining in MEGA |
*Картинка под таблицей - изображение реального дерева.
Различие в PAM 250 и BLOSUM62 в том, что эти методы используют различные матрицы расстояний, а % Identify - просто процент одинаковых позиций. К сожалению не удалось увидеть серьезных различий (хотя все-таки различия есть) между всеми этими методами, хотя предполагается, что PAM 250 и BLOSUM62 должны работать лучше.
Различие между MEGA и Jalview: с первого взгляда кажется, что различие состоит в том, что лист GEOKA в случае MEGA стоит в группе с CLOTE и CLOBM, а в случае Jalview - со всеми остальными, но это не так. Если мы переукореним дерево, полученное с помощью MEGA в ветвь {CLOTE, CLOBM}, то получим такое же дерево по топологии, как и все деревья, полученные с помощью Jalview. Так что никаких топологических различий в деревьях нет.
Итог: Несмотря на то, что в данном случае не удалось увидеть хоть каких-нибудь серьезных различий в топологии деревьев, построенных разными методами. Наверно действительно хороший результат MEGA продемонстрировал бы, если бы анализировались не одна, и не две, а несколько последовательностей, хотя утверждать такое нельзя.
⌘
© Emir Radkevich, 2016