Поиск по PDB и содержание PDB файлов
Occupancy ≠ 1
Поле occupancy отражает долю молекул, находящихся в данной конформации. В болбшинстве случаев этот коэффициент равен единице, что означает, что во всех молекулах данный атом находится в данной точке.
Необходимо было найти pdb файл, в котором есть атомы с коэффициентом occupancy ≠ 1. Для этого в PDB был произведен расширенный поиск с параметрами "Experimental method: X-ray diffraction" и "Resolution ≤ 1". В результате была выбрана структура с PDB ID 6CNW. Найденные строчки cо значением resolution ≤ 1:
ATOM 793 H AILE A 51 13.575 -24.186 3.495 0.56 6.91 H ATOM 794 H BILE A 51 13.616 -24.208 3.460 0.44 6.91 HПервые три числа — x, y, z координаты, четвертое — коэффициент occupancy. В 56% молекул атом находится в точке с координатами из верхней строчки, а в 44% — из нижней.
Отсутствующие остатки
Зачастую подвижные участки белка не расшифровываются в РСА эксперименте, поэтому их координаты не включаются в pdb-файл и перечисляются в поле Missing residues. Для структуры с PDB ID 3LFU найдены следующие отсутствующие остатки:
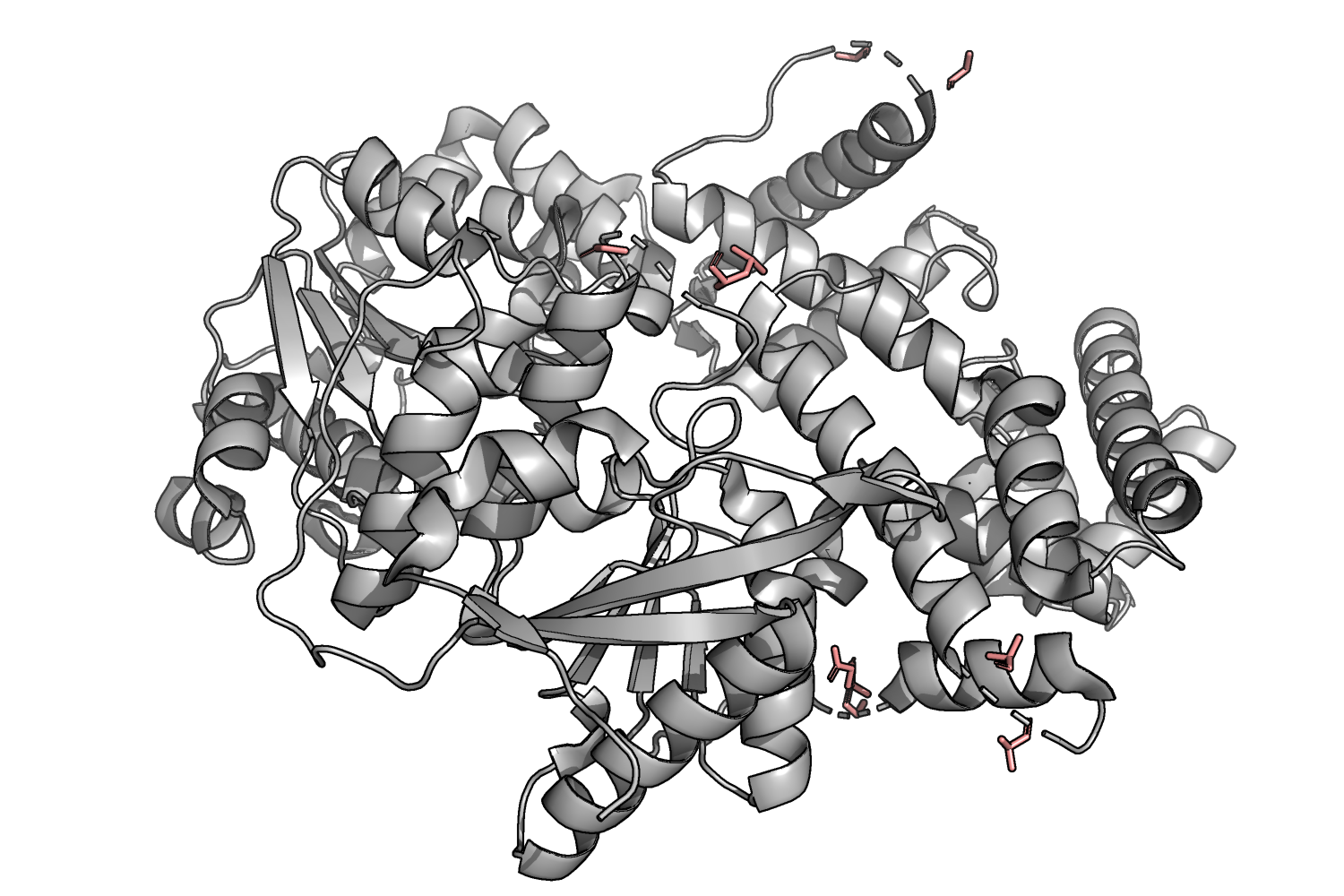
REMARK 465 MISSING RESIDUES REMARK 465 THE FOLLOWING RESIDUES WERE NOT LOCATED IN THE REMARK 465 EXPERIMENT. (M=MODEL NUMBER; RES=RESIDUE NAME; C=CHAIN REMARK 465 IDENTIFIER; SSSEQ=SEQUENCE NUMBER; I=INSERTION CODE.) REMARK 465 REMARK 465 M RES C SSSEQI REMARK 465 MET A 1 REMARK 465 ASP A 2 REMARK 465 GLN A 160 REMARK 465 SER A 161 REMARK 465 TYR A 162 REMARK 465 GLY A 163 REMARK 465 TYR A 521 REMARK 465 ASN A 522 REMARK 465 GLU A 523 REMARK 465 GLU A 524 REMARK 465 ASP A 525 REMARK 465 ALA A 542 REMARK 465 ASP A 587 REMARK 465 GLU A 588Видно, что есть два нерасшифорванных стрэтча (160-163 и 521-525) и еще несколько отсутствующих остатков. На картинке ниже видно, что все они находятся на поверхности белка в петлях между элементами вторичной структуры (розовым отмечены остатки, фланкирующие отсутствующие остатки):
Отличия в последовательностях кристаллизованного белка и белка в UniProt
C PDB структурой белка связаны три последовательности: (1) последовательность природного белка из Uniprot; (2) последовательность белка, который кристаллизовали (может отличаться наличием тэгов, быть частью природного белка); (3) та часть последовательности (2), которую удалось кристаллизовать. Необходимо было привести пример, в котором (1) и (2) не совпадают. Для этого в PDB был проведен поиск с параметрами "Experimental method: X-ray" и "Sequence features: wild-type protein". Для структуры с PDB ID 5XWM найдены следующие строчки в полях DBREF и SEQADV:
DBREF 5XWM A 1 377 UNP Q9BS26 ERP44_HUMAN 30 406 DBREF 5XWM B 1 377 UNP Q9BS26 ERP44_HUMAN 30 406 DBREF 5XWM C 1 377 UNP Q9BS26 ERP44_HUMAN 30 406 DBREF 5XWM D 1 377 UNP Q9BS26 ERP44_HUMAN 30 406 SEQADV 5XWM GLY A -5 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM SER A -4 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM HIS A -3 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM MET A -2 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM ALA A -1 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM SER A 0 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM GLY B -5 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM SER B -4 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM HIS B -3 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM MET B -2 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM ALA B -1 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM SER B 0 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM GLY C -5 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM SER C -4 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM HIS C -3 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM MET C -2 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM ALA C -1 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM SER C 0 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM GLY D -5 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM SER D -4 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM HIS D -3 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM MET D -2 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM ALA D -1 UNP Q9BS26 EXPRESSION TAG SEQADV 5XWM SER D 0 UNP Q9BS26 EXPRESSION TAGВ поле DBREF указывается соответствие между текущей последовательностью (прописанной в поле SEQRES) и последовательностями других баз данных (в данном случае — UniProt). Поле SEQADV показывает отличия последовательности данного pdb-файла, указанной в поле SEQRES, от последовательности, описанной в поле DBREF.
В данном случае, видно, что последовательности отличаются наличием экспрессионного тэга.
Худший и лучший B-фактор
B-фактор отражает подвижность атома и пропорционален величине "размазывания" его электронной плотности. Значения ниже 10 Å говорят о малой подвижности атома, выше 50 Å — о высокой подвижности. Для структуры с PDB ID 3LFU ниже представлены строчки с самыми высокими и низкими значениями В-фактора (последнеее число в строчке):
ATOM 1902 N ILE A 245 45.109 19.546 -10.940 1.00 13.59 N ATOM 1903 CA ILE A 245 44.083 19.053 -10.022 1.00 13.30 C ATOM 1904 C ILE A 245 42.973 18.475 -10.872 1.00 12.93 C ATOM 1905 O ILE A 245 42.717 18.917 -11.997 1.00 13.00 O ATOM 1906 CB ILE A 245 43.547 20.122 -9.039 1.00 14.20 C ATOM 1907 CG1 ILE A 245 42.814 21.231 -9.815 1.00 13.86 C ATOM 1908 CG2 ILE A 245 44.713 20.638 -8.134 1.00 12.86 C ATOM 1909 CD1 ILE A 245 41.912 22.144 -9.027 1.00 16.69 C ATOM 4224 N GLY A 543 23.202 -15.353 14.037 1.00 75.43 N ATOM 4225 CA GLY A 543 22.241 -15.491 12.947 1.00 75.88 C ATOM 4226 C GLY A 543 22.015 -14.213 12.159 1.00 76.03 C ATOM 4227 O GLY A 543 22.101 -13.109 12.706 1.00 76.22 O
⌘
© Emir Radkevich, 2018