Определение вторичной структуры
Определение вторичной структуры
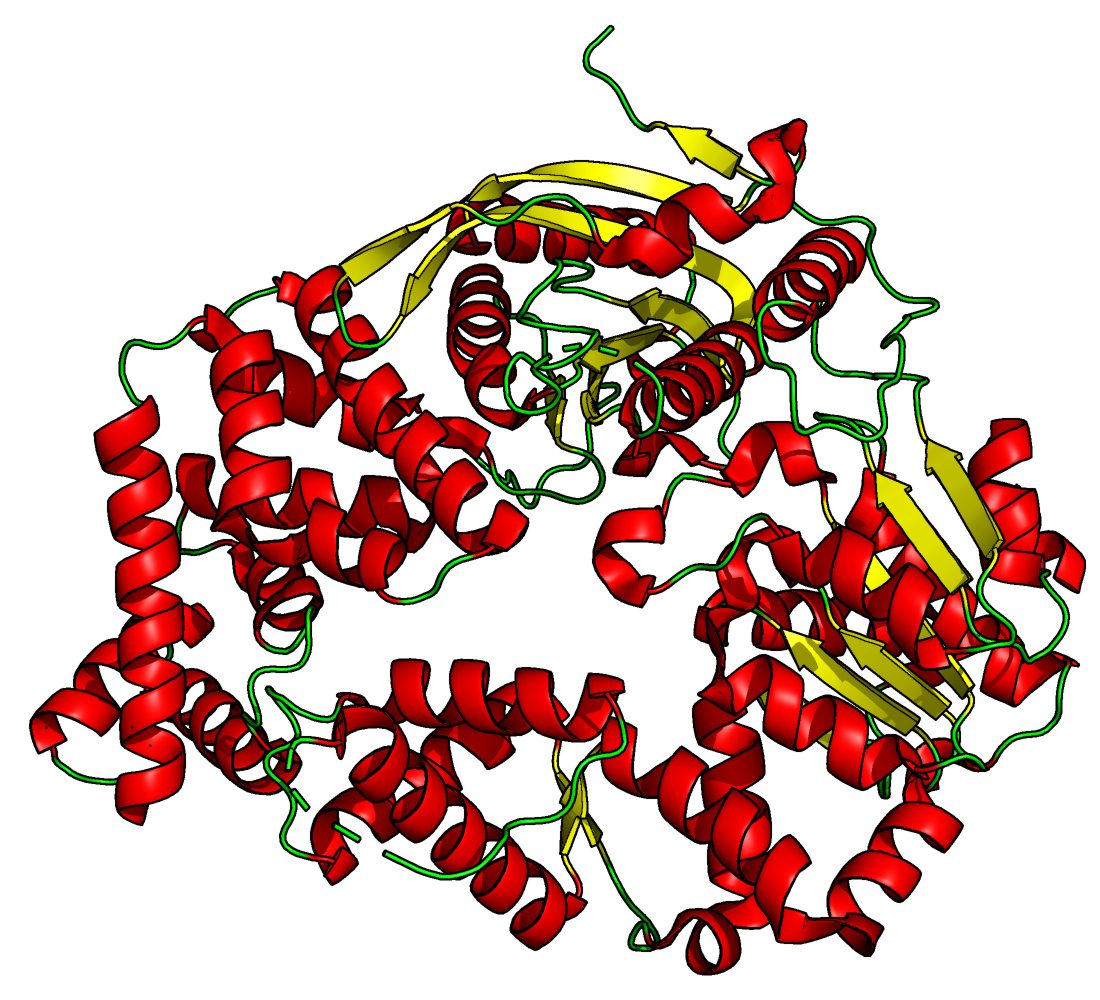
Для анализа был взят белок с PDB ID 3LFU. Исследуемая гидролаза показана на рисунке ниже. Элементы вторичной структуры покрашены следующим образом: α-спирали покрашены в красный цвет, β-стрэнды в желтый цвет, неупорядоченные участки в зеленый цвет.
Вторичные структуры определялись при помощи программ DSSP [1] и Stride [2]. Файлы с результатами: stride.out, dssp.out. Полученные файлы сравнивались с pdb файлом.
Также, в таблице 1 проиллюстрировано сравнение выдачи DSSP, Stride и содержимого pdb файла (розовым отмечены случаи несовпадения с содержимым pdb файла).
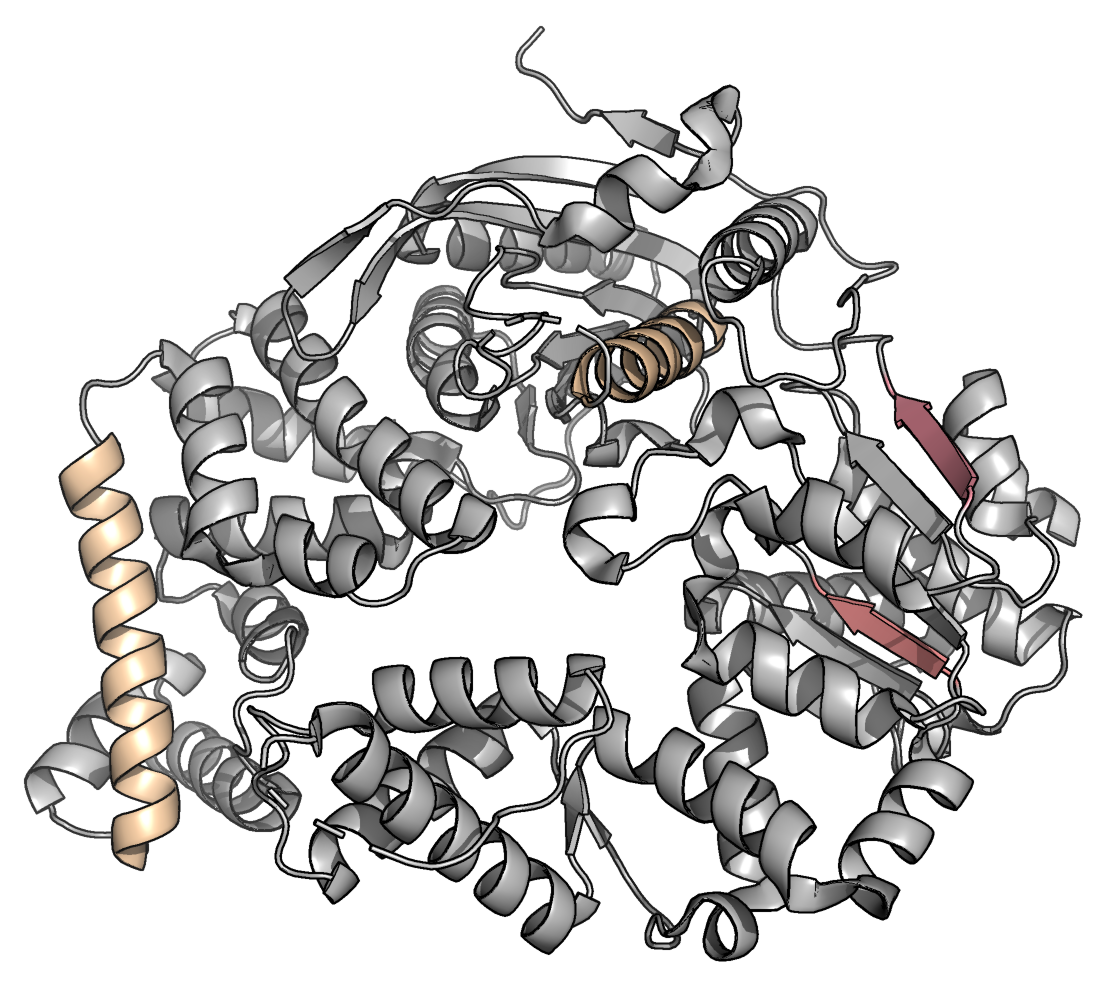
| Таблица 1. Сравнение выдачи DSSP, Stride и содержимого pdb файла | |||
|---|---|---|---|
| Элемент вторичной структуры | Границы в pdb файле | Границы в DSSP | Границы в Stride |
| α-спираль на поверхности белка | Ala451-Thr472 | Gly452-Glu471 | Gly452-Thr472 |
| α-спираль внутри белка | Arg591-Thr604 | Leu592-Val603 | Arg591-Thr604 |
| β-стрэнд на поверхности белка | Glu275-Leu279 | Glu275-Leu279 | Glu275-Leu279 |
| β-стрэнд внутри белка | Asn216-Val219 | Asn216-Val219 | Asn216-Val219 |
Исходя из данных, представленных в таблице 1, видно, что β-стрэнды определены с одинаковой разметкой во всех случаях. Также, видно, что α-спираль на поверхности белка во всех случаях имеет различные границы (различие на самом деле невелико — всего 1 а.о.). В принципе, это неудивительно наблюдать, так как концы элемента вторичной структуры, находящегося на поверхности белка, вероятнее будут размечаться по-разному, в отличие от элемента, находящегося внутри белка.
Что касается разметки α-спирали внутри белка, видно, что разметка в случае использования DSSP отличается от остальных разметок. По-видимости, это связано с тем, что малоструктурированные концевые участки, как и в случае с α-спиралью на поверхности белка, размечаются хуже, чем внутренний участок α-спирали.
Ссылки на литературу
1. Touw WG, Baakman C, Black J, Beek TAH, Krieger E, Joosten RP, Vriend G — A series of PDB related databases for everyday needs. Nucl Acids Res (2015); 43(Database issue): D364-D368.
2. Heinig M, Frishman D — STRIDE: a Web server for secondary structure assignment from known atomic coordinates of proteins. Nucl Acids Res (2004); 32: W500-2.
⌘
© Emir Radkevich, 2018