Нуклеотидные банки данных
Задание 1
В качестве примера эукариотического организма был выбран Manacus vitellinus — золотистый короткокрылый манакин.
Рис. 1. Manacus vitellinus

На данный момент существует две сборки генома и 4 проекта по секвенированию единственного образца SAMN02299332.
Для рассмотрения была выбрана сборка GCA_000692015.2 (genbank accession).
Характеристика использованного для сборки образца
Идентификатор образца: SAMN02299332 | Название образца: BGI_N305 | SRA (Seqquence Read Archive): SRS466478
Вид организма: Manacus vitellinus
Таксономическое положение:
- Eukaryotes (надцарство Эукариоты)
- Metazoa (царство Животные)
- Chordata (тип Хордовые)
- Craniata (п/тип Позвоночные)
- Aves (класс Птицы)
- Neognathae (н/отряд Новонёбные)
- Passeriformes (отряд Воробьинообразные)
- Pipridae (семейство Манакиновые)
- Manacus (род Манакин)
- Manacus vitellinus (вид Золотистый короткокрылый манакин)
- Manacus (род Манакин)
- Pipridae (семейство Манакиновые)
- Passeriformes (отряд Воробьинообразные)
- Neognathae (н/отряд Новонёбные)
- Aves (класс Птицы)
- Craniata (п/тип Позвоночные)
- Chordata (тип Хордовые)
- Metazoa (царство Животные)
Особенности: образец BGI_N305 выделен в Панаме из самки Manacus vitellinus.
Перечень проектов, использующих образец:
- PRJNA341382
- PRJNA335611
- PRJNA253835
- PRJNA212872
Предоставлен: 2 августа 2013 года BGI (Beijing Genomics Institute)
Характеристика проекта по секвенированию
- Идентификатор проекта: PRJNA212872
- Тип проекта: сборка и секвенирование генома
- Выполнен: BGI (Beijing Genomics Institute)
- Зарегистрирован: 13.05.2014
- Актуальность: применительно к эволюции
- RefSeq проект: PRJNA253835
Характеристика сборки GCA_000692015.2
- Идентификатор сборки: GCA_000692015.2 (GenBank) | WGS-проект: JMFM02000000
- Полная длина последовательности: 1 145 854 002
- Суммарная длина гэпов сборки: 29 843 881
- Гэпы между скэффолдами: 0
-
Общее число N50 L50 Контиги 138 155 43 697 6 945 Скэффолды 92 755 2 558 866 124 - Таблица контигов: в формате [xlsx] доступна для скачивания
- Самый длинный контиг: JMFM02021993 имеет длину 468443
- Самый короткий контиг: 249 контигов имеют длину 200
- Последовательность контига: для JMFM02000023 в формате [FASTA] доступна для скачивания
- Таблица скэффолдов: в формате [xlsx] доступна для скачивания
- Самый длинный скэффолд: scaffold11 имеет длину 12468512
- Самый короткий скэффолд: 244 штуки имеют длину 200
Задание 2
GenBank, EMBL и DDBJ разработали общие стандарты[1] для формата аннтоаций. Ключевой задачей Feature Tables, так называемых таблиц особенностей, помимо непосредственно предоставляемого описания, является также возможность быстрого и удобного оперирования данными.
Спектр особенностей, описываемых в этих таблицах, очень широк и включает участки, которые:
- связаны с выполнением биологических функций;
- связываются с другими молекулами;
- связаны с репликацией последовательности;
- являются продуктом рекомбинации различных последовательностей;
- являются узнаваемыми повторяющимися единицами;
- обладают вторичной или третичной структурой;
- вариабельны или были пересмотрены и изменены.
Общий синтаксис таблиц особенностей:
Key Location/Qualifiers CDS 23..400 /product="alcohol dehydrogenase" /gene="adhI"
Key — ключ;
Location — расположение в последовательности;
Qualifiers — классификаторы с конкретными дополнительными деталями (для каждого ключа — свои, причём имеются как обязательные, так и необязательные).
Задание заключалось в описании десяти выбранных ключей, используемых в таблицах особенностей.
Описание ключа в документации INSDC может включает в себя такие особенности, как:
- обязательные (mandatory qualifiers) и дополнительные (optional qualifiers) классификаторы;
- те организмы, у которых этот ключ может присутствовать в аннотации последовательности (organism scope);
- те молекулы, в последовательности которых может быть данный ключ (molecule scope);
- ссылки на цитирования и исследования
- комментарий с пояснениями
| Ключ | Значение | Пример |
| 1 | ||
| CDS | кодирующая последовательность | FEATURE Location/Qualifiers
CDS join(544..589,688..>1032)
/product="T-cell receptor beta-chain" |
| Объединив отрезок 544-589 и отрезок, начинающийся с 688-го нуклеотида и продолжающийся после 1032-го, получим нуклеотидную последовательность, кодирующую бета-цепь белка рецептора Т-клетки. | ||
| 2 | ||
| rep_origin | ориджин репликации | FEATURE Location/Qualifiers
rep_origin 3617..3892
/standard_name="ori1"
/experiment="experimental evidence, no additional
details recorded"
/citation=[2] |
| Квалификатор /standard_name встречается часто с различными ключами
и используется для записи общепринятых стандартных названий.
Квалификатор /experiment вкратце отражает суть эксперимента, подтверждающего наличие особенности. /citation — цитирование. Список References приводится в начале аннотации. | ||
| 3 | ||
| sig_peptide | кодирующая последовательность сигнального пептида | FEATURE Location/Qualifiers
sig_peptide 9146..9226
/locus_tag="L2_14"
/note="putative" |
| Квалификатор /note позволяет прокомментировать особенность. В нашем случае, например, сообщается о том, что наличие указанной особенности лишь предполагается. | ||
| 4 | ||
| assembly_gap | гэп между двумя компонентами сборки генома | FEATURE Location/Qualifiers
assembly_gap 1813..4923
/estimated_length=3111
/gap_type="within scaffold"
/linkage_evidence="unspecified" |
| В отличие от предыдущих случаев, все три квалификатора являются для данного ключа обязательными. /estimated length — длина гэпа; /gap_type — тип гэпа (например: внутри скэффолда / вне скэффолда и другие); /linkage_evidence — тип свидетельства, подтверждающего наличие связи в гэпе сборки (например: "pcr", "paired-ends" и другие). В нашем случае такого свидетельства нет. | ||
| 5 | ||
| source | биологический источник образца | FEATURES Location/Qualifiers
source 1..574052
/organism="Cynoglossus semilaevis"
/mol_type="genomic DNA"
/isolate="Cse_v1.0"
/db_xref="taxon:244447"
/chromosome="6"
/tissue_type="blood"
/country="China"
/collection_date="Sep-2009" |
| Ключ source является обязательным. Их может быть несколько,
но тогда объединение их locations покрывает всю последовательность.
В классификаторах этого ключа содержится разнообразная информация о том откуда, где, когда и как был получен образец. | ||
| 6 | ||
| D-loop | D-петля; область митохондриальной ДНК, в которой короткий участок РНК спарен с одной цепью ДНК, как бы замещая в этом месте вторую цепь 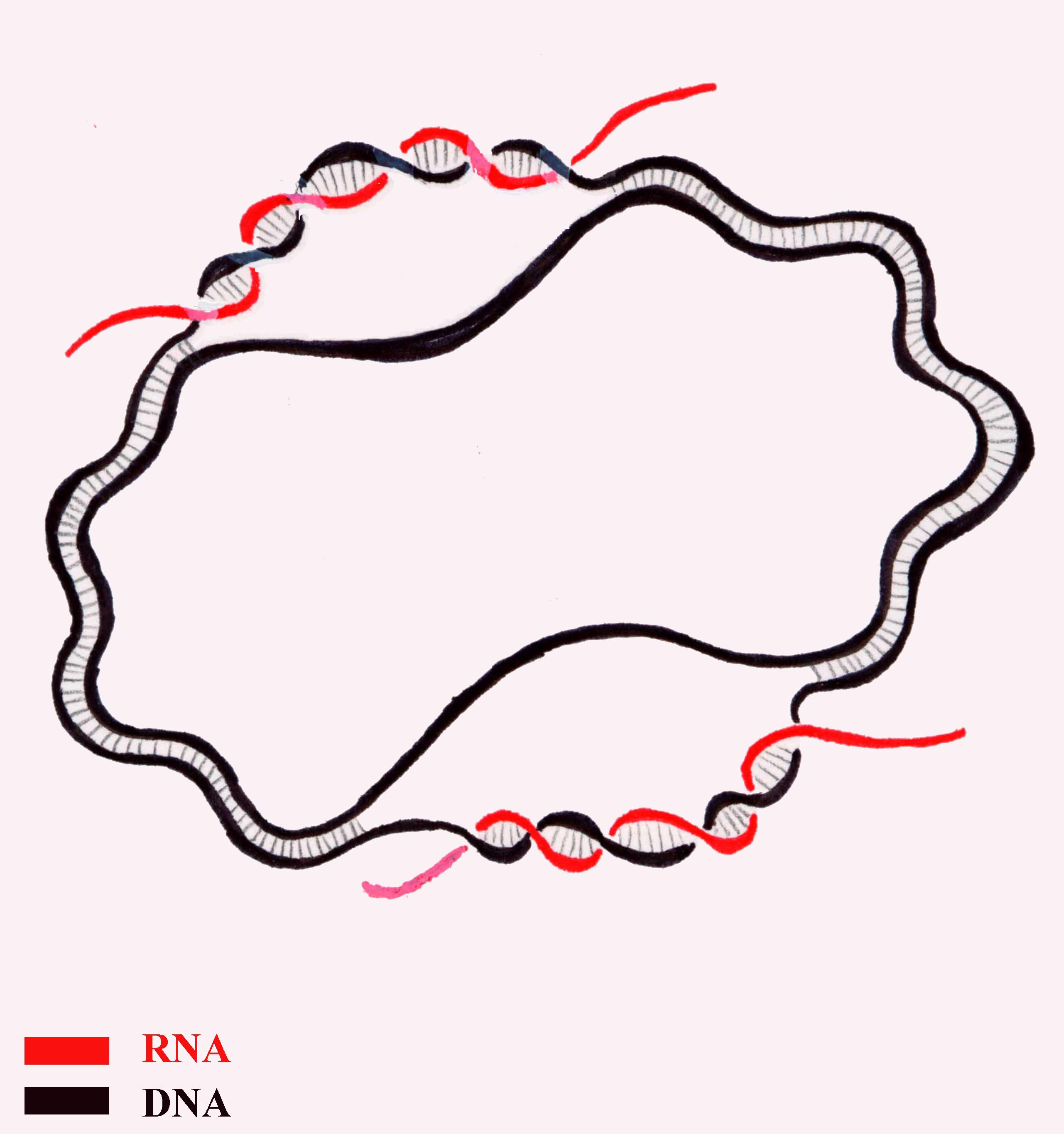 |
FEATURES Location/Qualifiers
D-loop 15784..16659
/note="control region" |
| Ключ также используется для описания смещения участка одной из цепей дуплексной ДНК какой-либо одноцепочечной молекулой в реакции, катализируемой белком RecA. | ||
| 7 | ||
| misc_recomb | участки, добавленные в геном или выпавшие из него вследствие рекомбинации | FEATURES Location/Qualifiers
misc_recomb 5083..5084
/locus_tag="lambdap70"
/gene="NinI" |
| Учитываются сайты общей, сайтоспецифичной и репликативной рекомбинации. | ||
| 8 | ||
| variation | полиморфизм | FEATURES Location/Qualifiers
variation 467
/locus_tag="lambdap89"
/replace="t"
/gene="O"
/note="t in ric5b; c in wild-type" |
| Полиморфизм как однонуклеотидный, так и длин рестрикционных фрагментов (RFLP). Если задать классификатор /replace="", это будет означать делецию. В общем случае это замена нуклеотида. | ||
| 9 | ||
| repeat_region | участок генома, содержащий повторы | FEATURES Location/Qualifiers
repeat_region 80..401
/rpt_type=DISPERSED |
| Для /rpt_type есть большое количество возможных значений, характеризующих структуру и распределение повторов. | ||
| 10 | ||
| stem_loop | шпилька или стебель | FEATURES Location/Qualifiers stem_loop join(978..988,1..17) |
| Location задаётся через объединение двух участков цепи, которые образуют стебель. | ||
Задание 3

Для того чтобы исследовать генетические механизмы, влияющие на развитие эпилепсии у большинства людей, в 2010 году при финансировании от NINHS (National Institute of Neurological Disorders and Stroke) был создан так называемый "Center without Walls" и запланировано исследование Epi4K, имевшее своей целью отсеквенировать и проанализировать геномы и фенотипы 4000 больных людей[3].
В команде проекта участвовало более 60 учёных с трёх континентов (Северная Америка, Евразия, Австралия).
К маю 2013 года в исследованиях приняли участие 4199 людей. Цель достигнута. Но исследования в указанной области всё ещё продолжаются.
Сайт проекта доступен по ссылке
Ссылка на последнюю публикацию: здесь
Задание 4
Для таксона Rhodophyta были составлены[2] запросы в NCBI, по которым находятся все полные митохондриальные геномы (83 штуки) в GenBank и RefSeq соответственно:
- (rhodophyta[ORGN] AND mitochondrion[TITLE] AND ("complete genome"[TITLE] OR "complete sequence"[TITLE])) AND srcdb_genbank[PROP]
| 49 находок | - (rhodophyta[ORGN] AND mitochondrion[TITLE] AND ("complete genome"[TITLE] OR "complete sequence"[TITLE])) AND srcdb_refseq[PROP]
| 34 находки |
В качестве представителя был выбран Chondrus crispus (см. Рис. 2), также известный как "ирландский мох". Из этой водоросли выделяется каррагинан, который в течение сотен лет используется в качестве пищевой добавки. По сути это альтернатива желатина растительного происхождения.
Рис. 2. Chondrus crispus

Для Chondrus crispus имеется полный митохондриальный геном, accession number в RefSeq NC_001677.
Рис. 3. Митохондриальный геном
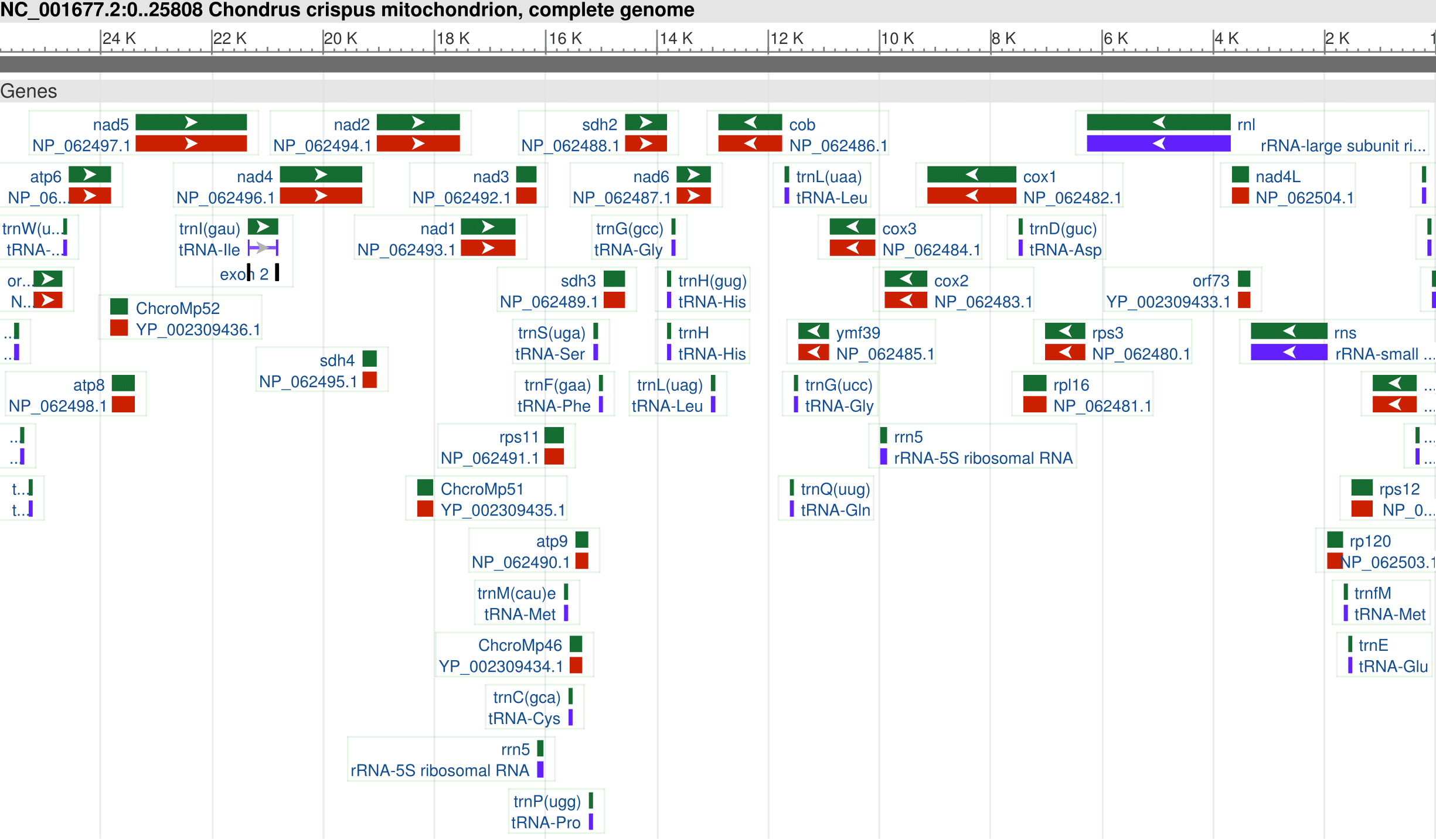
Таблица генов митохондриального генома этого организма в формате [xlsx] доступна для скачивания. На одном из листов приведены все гены, а на втором — только белок-кодирующие.
Задание 5
Таблица 2 содержит информацию о размерах геномов по разным таксонам.
Таблица 2
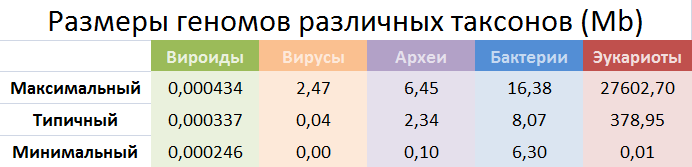
Файл [xlsx], в котором содержатся расчёты, доступен по ссылке.
[1] INSDC Documentation
[2] Fields available for all Sequence Databases
[3] Gene discovery in 4,000 genomes