Предсказание генов прокариот
Задание 1
Мне была выдана плазмида с идентификатором INSDC: CP015440, принадлежащая Anoxybacillus amylolyticus. Это термофильная бактерия, умеющая синтезировать амилазу.
Описание плазмиды: размер — 65295 bp; число генов: 83 (из них белок-кодирующих: 64, тРНК-кодирующих: 19).
Информацию о последовательности плазмиды и об особенностях (features) я извлекла в два файла — [fasta] и [gff] соответственно.
seqret embl:CP015440 -feature CP015440.fasta
Выполнение приведённой команды создаёт не только указанный CP015440.fasta файл, содержащий последовательность, но и одноимённый CP015440.gff файл с особенностями, что связано с указанием параметра -feature.
Затем я использовала программу Prodigal, которая на вход получает [fasta]-последовательность, а на выходе даёт файл genecoords.sco [sco]-формата (параметр -f sco), содержаший координаты предсказанных генов.
prodigal.windows -i CP015440.fasta -f sco -o genecoords.sco
Для последующего сравнения предсказания с аннотацией был написан Python-скрипт prodigal_analysis.py, доступный для скачивания. Запуск производится обычно командой python prodigal_analysis.py, после
чего программа сама спросит имена входных файлов (с аннотированными генами и предсказанными) и имя выходного файла:
Выходной файл out.txt выглядит так:
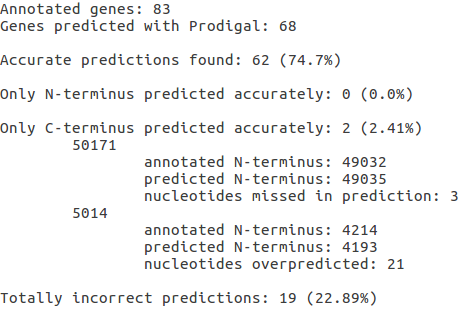
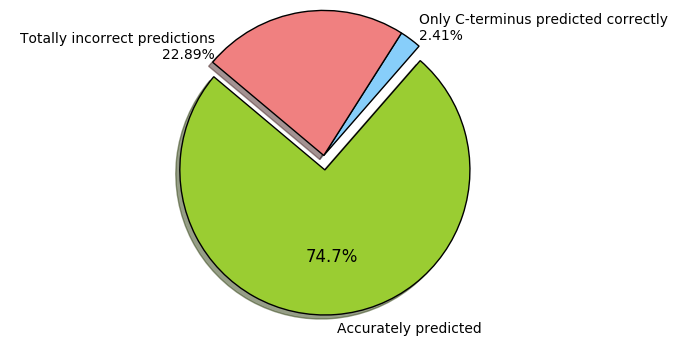
Значения в скобках — это проценты от всех предсказанных с помощью Prodigal генов. По этим значениям с помощью matplotlib.pyplot была построенна диаграмма, приведённая на Рис. 1.
Рис. 1. Анализ предсказания Prodigal
Скрипт работает следующим образом. Сначала из обоих файлов извлекаются координаты: начало гена, конец, ориентация цепи. Эти координаты в одном и том же формате (строка с тремя значениями с табулятором в качестве разделителя) сохраняются в специальных множествах. Затем рассматривается пересечение этих множеств. Получаем количество абсолютно точных предсказаний. Затем для учёта ориентации цепи в анализе верных предсказаний N- и С- концов координаты получают соответствующее направлению цепи положительное или отрицательное значение. Создаются множества аннотированных и предсказанных N- и C-концов. Опять-таки верность предсказания равносильна тому, что какое-то значение содержится в пересечении множеств. Так подсчитываются случаи с точно определенным одним из концов. Оставшиеся же случаи, очевидно, относятся к типу Totally incorrect.
В моем случае получилось 2 предсказания, в которых верно был предсказан только С-конец. (см. Рис. 1.) Посмотрим, что пошло не так с N-концом.
Ген 1: (49032..50171). Здесь, как следует из вывода скрипта, предсказателем были пропущены три первые нуклеотида. Посмотрим на Рис. 2.
Рис. 2. Пропуск первых трёх нуклеотидов

Цветом выделен аннотированный ген. Он, как мы видим, начинается с GTG (цепь прямая, поэтому это соответствует N-концу). Этот кодон в Таблице трансляции №11 (ее использование аннотировано) является стартовым (см. Рис. 3).
Рис. 3. Таблица трансляции №11

Но как следует из комментариев к таблице, более распространена инициация с ATG (AUG). Эти три нуклеотида и идут в позициях 4-6 (Рис. 2). Можно предположить, что именно поэтому Prodigal посчитал начало гена именно таким и, вследствие чего, ошибся.
Ген 2: (4214..5014). В предсказании этого гена Prodigal дал координаты начала на 7 триплетов раньше (21 нуклеотид). На Рис. 4 приведена последовательность гена (коричневым), а также оранжевым выделены три нуклеотида, которые были предсказаны как начало гена.
Рис. 4. Начало гена предсказано на 21 нуклеотид раньше

Нетрудно заметить, что ситуация сложилась ровно такая же: программа предпочла начать ген с ATG, а не с GTG.