Предсказание генов эукариот
Задание 1
Для выполнения задания практикума мне был выдан контиг с идентификатором NW_014547703, для которого была скачана последовательность в формате [fasta]. Контиг взят из генома Alligator mississippiensis — американского, или миссисипского аллигатора. Краткая таксономия и изображение A.mississippiensis приведены ниже.
 |
>superkingdom Eukaryota
>kingdom Metazoa
>phylum Chordata
>subphylum Vertebrata
>group Sauropsida
>order Crocodylia
>family Alligatoridae
>genus Alligator
|
| Источник: Gareth Rasberry / CC BY-SA 3.0 |
Краткая информация о контиге:
- Размер контига: 834972 bp
- Число генов: 19
- Число кодирующих последовательностей: 25
В данном контиге был найден ген, для которого предсказан альтернативный сплайсинг:
- Ген: LOC102560820
- Расположение: complement(366,835..397,657)
- Длина: 30,823
- Описание: кодирует DDI1-подобный белок
DDI1[1] — ген, кодирующий одноимённый (DNA damage inducible 1) белок, который участвует во взаимодействии c убиквитинирующей системой. Это может происходить либо с помощью N-концевого убиквитин-подобного домена, либо С-концевого убиквитин-ассоциированного домена. Помимо этого DDI1 также имеет центральный участок, схожий с ретровирусной аспартатной (по строению активного центра) протеазой. Его функции состоят в регулировании клеточного цикла и в контроле секреции белков.
В изучаемом контиге было предсказано наличие четырёх изоформ для продукта гена LOC102560820. (см. Рис. 1).
Рис. 1. Ген LOC102560820

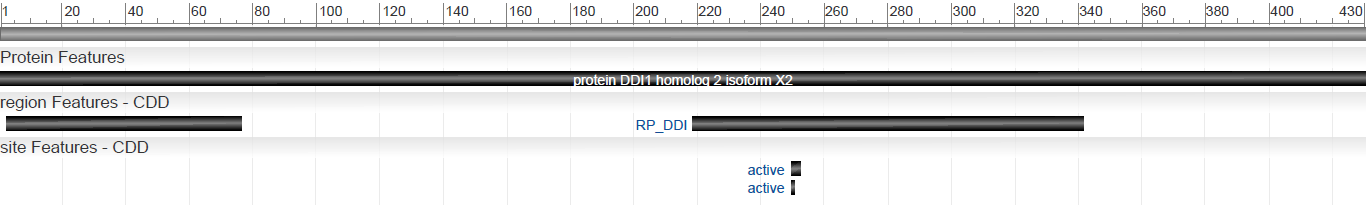
Также в геномном браузере были получены изображения мРНК и белка для одной из изоформ (X2). Они приведены на Рис. 2 и Рис. 3.
Рис. 2. мРНК XM_006261687.1 и продукт её трансляции
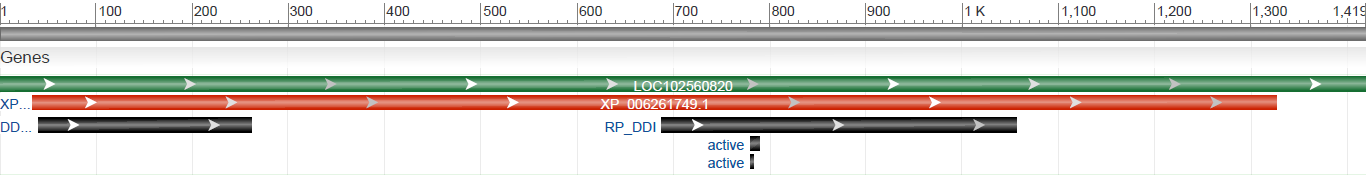
Рис. 3. Изоформа X2 белка XP_006261749.1
Задание 2
Далее файл с последовательностью контига был проанализирован с помощью AUGUSTUS. Параметры запуска можно видеть ниже:

Таким образом, нужно задать параметры модели, указать, что именно и каким образом предсказывать — нужны ли предсказания UTR, какую из цепей анализировать, насколько детально прорабатывать альтернативные транскрипты и какие структуры генов мы ожидаем (полные/частичные).
Для получения параметров модели был выбран организм Gallus gallus, или chicken, — курица. Такой выбор был обусловлен тем, что список видов, предлагаемый AUGUSTUS невелик, и из него самым близким к изучаемому виду оказался вид G. gallus. Выбор, на первый взгляд, не самый очевидный, но убедиться в близости организмов можно, обратившись к кладограмме на Рис. 4. Gallus gallus относится к ветви Aves (Птицы), а A. mississippiensis — к Crocodilia.
Рис. 4. Кладограмма
Параметр предсказания UTR был включен, что, впрочем, ничего не изменило, так как в AUGUSTUS нет файлов с оптимизированными параметрами UTR для Gallus gallus. Соответствующее предупреждение сервера можно также видеть в параметрах запуска.
По завершении работы AUGUSTUS выдал [tar.gz]-архив с шестью файлами. Их содержание:
| Расширение файла | Содержание |
| *.aa | предсказание генов в виде fasta-последовательности белков |
| *.cdsexons | предсказанные экзоны в fasta-последовательности ДНК |
| *.codingseq | предсказание генов в виде fasta-последовательности кодирующих участков ДНК |
| *.gbrowse | трэк-файл предсказания для GBrowse |
| *.gff | предсказание генов в обычном [gff]-формате (General Feature Format) |
| *.gtf | предсказание генов в [gtf]-формате ([gff] 2-ой версии) |
Основным файлом предсказания будем считать [gff]-файл, доступный для скачивания.
На Рис. 5 приведено сравнение предсказанных генов (снизу) с аннотацией (сверху).
Рис. 5. Сравнение предсказания AUGUSTUS с аннотацией

Предсказно 19 генов. Столько же и аннотировано. Но, взглянув на рисунок выше, можно понять, что это можно считать случайным совпадением. Количество генов действительно совпадает, только вот расхождения в координатах иногда очень и очень существенные. Ярким примером является ген EIF4G3 (располагается в отрезке 500k-700k). На соответстующем отрезке AUGUSTUS предсказал 4 гена. Два из них имеют подозрительно маленькую длину относительно остальных генов, что уже может натолкнуть на мысль о неточности предсказания, если не иметь аннотации.
Ген LOC106738970 (подписан как L и располагается в отрезке 230k-240k) не был предсказан вообще.
Отметим, что произошла и обратная ситуация: на отрезке, в котором гены не аннотированы вообще, AUGUSTUS предсказывает небольшой ген g6, располагающийся в области 145k-148k.
Из характерных "ошибок" AUGUSTUS можно указать на склонность склеивать гены, расстояние между которыми невелико. Тому видим целых три примера:
- FBLIM (261072..280034) и TMEM82 (286784..292514) предсказываются как единый g8 (261735..291942)
- SLC25A34 (293588..301743) и PLEKHM2 (308285..356483) —> единый g9 (295121..356483)
- SH2D5 (439360..445085) и HP1BP3 (461997..482872) —> единый g13 (439360..478534)
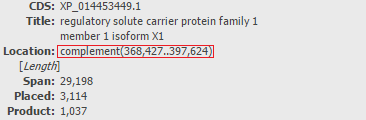
Для выбранного же в Задании 1 гена LOC102560820 (366835..397657) предсказание (см. ниже)получилось не таким уж плохим: g10 (368427..397624). (Указанные гены располагаются на обратной цепи). В предсказании начала гена происходит ошибка на 33 нуклеотида, а для конца — на 1592.

Заметим, что одна из изоформ продукта этого гена кодируется, согласно аннотации, как раз в таких же координатах (см. ниже). Так что можно предположить, что AUGUSTUS не очень точно справляется с ситуациями альтернативного сплайсинга, отдавая в процессе предсказания предпочтение лишь определенному участку гена, соответствующему одной из изоформ.
[1] — NCBI: The retroviral proteinase active site and the N-terminus of Ddi1 are required for repression of protein secretion.