Прочтение последовательностей по Сэнгеру
Данные в виде файлов с хроматограммами формата [ab1] были получены из капиллярного секвенатора по Сэнгеру. Для просмотра и редактирования автоматического прочтения этих хроматограмм использовалась программа Chromas.
Задание 1. Прочтение последовательности ДНК
Файлы данного задания:
- исходные [ab1]-файлы с автоматическим прочтением прямой и обратной последовательностей;
- полученные после редактирования автоматического прочтения [FASTA]-файлы с прямой и обратной-комплементарной последовательностями;
- [jvp]-проект с выравниванием отредактированных последовательностей.
В следующей таблице приведены нечитаемые участки. Для обратной последовательности — после замены на комплеменатрную.
| Нечитаемый участок | 5'-конец | 3'-конец |
| Прямая цепь | 1-35 (35 нуклеотидов) | 382-386 (5 нуклеотидов) |
| Обратная цепь | 1-4 (4 нуклеотида) | 349-382 (34 нуклеотида) |
Общая характеристика хроматограммы прямой цепи:
- мощность сигнала в среднем составляет 1000, мощность шума — 150;
- отношение сигнала к шуму составляет примерно 6 к 1;
- сигнал распределен вдоль последовательности равномерно;
- иногда пурины дают значительно более высокий сигнал;
- распределение шума тоже можно назвать равномерным.
Общая характеристика хроматограммы обратной цепи (после замены на комплементарную):
- мощность сигнала в среднем составляет 1000, мощность шума — 100;
- отношение сигнала к шуму составляет примерно 10 к 1;
- сигнал распределен вдоль последовательности равномерно;
- распределение шума неравномерное: во второй половине хроматограммы он повышен относительно среднего.
После работы с обеими хроматограммами можно сказать, что качество второй значительно лучше. Там я встретилась с меньшим количеством проблемных участков. Причиной тому можно считать хорошее отношение сигнал-шум.
Отчёт о проблемах при редактировании автоматического прочтенияОпишу некоторые из проблемных ситуаций, возникших в ходе редактирования автоматического прочтения последовательностей ДНК. Я работала как с прямой, так и с обратной-комплементарной последовательностью. В ходе работы возникали, помимо прочих, такие случаи, когда однозначный выбор нуклеотида был невозможен. Тогда на этой позиции в [FASTA]-файле ставился один из так называемых вырожденных кодов (ambiguity codes[1]):
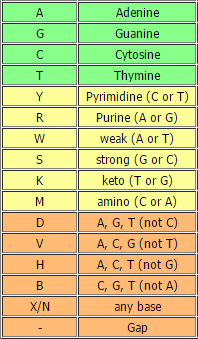
В Таблице 1 приведены несколько проблемных ситуаций и иллюстрации, их поясняющие. В верхней части двойных изображений всегда показана прямая, а снизу — обратная-комплеменатрная цепочка. Участки выровнены. (Выравнивание после удаления нечитаемых участков такое, что 1-ый нуклеотид прямой цепи соответствует 60-ому нуклеотиду обратной-комплементарной.
Таблица 1
| Проблема | Описание | Изображение |
| Слишком высокий шум затрудняет автоматическое определение нуклеотида | В 176-ой позиции прямой цепи нуклеотид был неопознан из-за перекрывания пиков сигнала и шума. Но при обращении к обратной-комплементарной цепи всё становится понятно — там этот участок не является проблемным, и мы чётко видим пик тимина. В [FASTA]-файл в этой позиции поставлена t. | 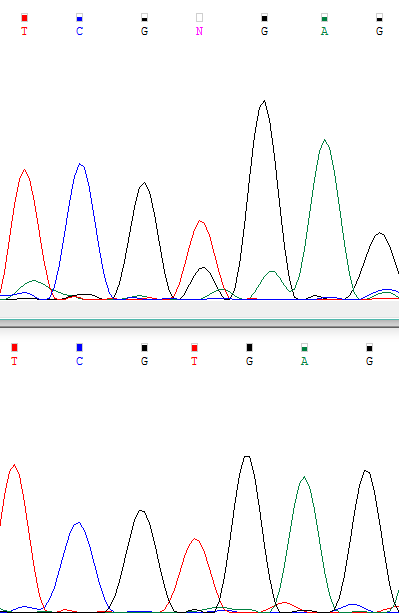 |
| Слишком высокий шум затрудняет автоматическое определение нуклеотида | Проблема остаётся той же. При рассмотрении обратной-комплементарной цепи в позиции 37 нуклеотид не распознан программой. Но только теперь всё осложняется тем, что аналогичный участок прямой цепи отсутствует (такое выравнивание), и поэтому проверка по ней оказывается невозможной. В [FASTA]-файл в этой позиции поставлен вырожденный код v (not t). | 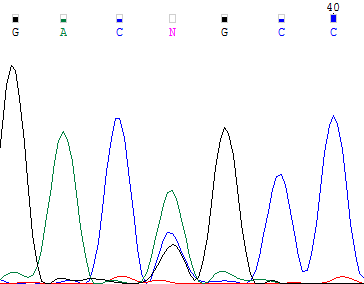 |
| Размытые пики и их перекрывание | На обратной цепи не распознаны нуклеотиды в позициях 342 и 344. На самом деле, если посмотреть на хроматограмму, то становится ясно, что у программы в позиции 342 возникают проблемы из-за широкого размытого пика тимина, а в позиции 344 — из-за сильного перекрывания неясно выраженного пика гуанина и более четкого пика аденина. Здесь нас опять-таки выручает неплохое качество аналогичного участка прямой цепи. Нуклеотид 342 определен как t, а 344 — как g. Последнее обстоятельство особенно интересно тем, что пик гуанина на хроматограмме самой обратной-комплементарной цепи был более размыт. | 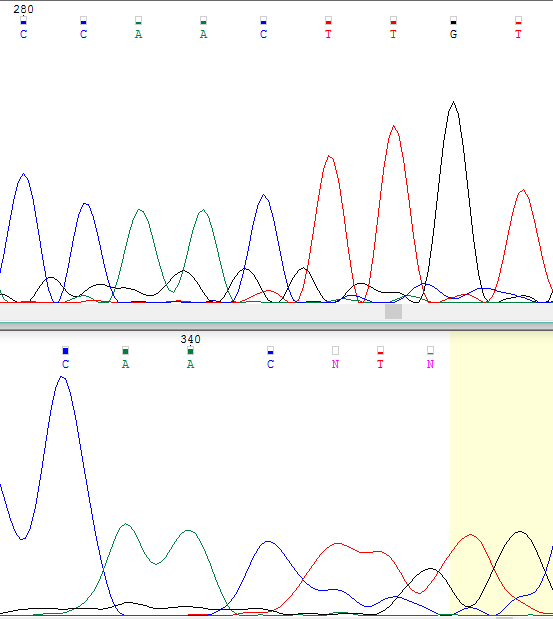 |
| Делеция нуклеотидов | На этом изображении мы видим три неопределённых нуклеотида 297-299 на обратной-комплементарной цепи. В целом участок полностью проблемный. Особенные неприятности вызывают широкие и очень размытые пики аденина и гуанина. Но в целом понятно, что 297 — a, в 298,299 — g. Нехарактерно большие расстояния между пиками указывают на делецию двух нуклеотидов — A и G, аналогичные которым в прямой цепи имеют номера 241 и 242. (На этом месте в выравнивании будет гэп.) | 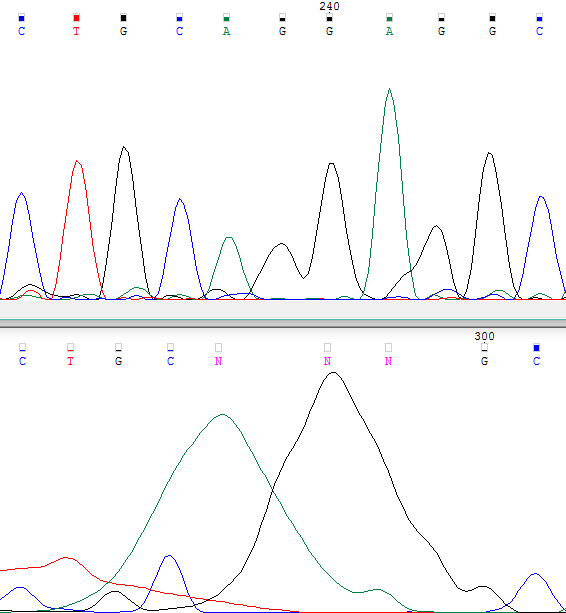 |
После проведенного редактирования полученные [FASTA]-последовательности прямой и обратной-комплементарной цепей были выровнены с ипользованием Muscle with defaults, а полученное выравнивание раскрашено по нуклеотидам.

Консенсусная последовательность:
>Consensus/1-405 Percentage Identity Consensus TAAAAMGACGGCCAGTATGGCTCGTACCAAGCAGACVGCCCGTAAATCTACCGGAGGCAAGGCCCCCCGCAA GCAGCTGGCCACCAAGGCTGCGCGCAAGTCTGCGCCCGCTACYGGAGGAGTCAAGAAGCCTCACAGGTACAG GCCCGGTACCGTCGCTCTCCGTGAGATCCGTCGTTACCAGAAGAGCACTGAGCTCCTCATCCGCAAGCTGCC TTTCCAGCGCTTGGTTCGTGAGATCGCTCAGGACTTCAAGACTGATCTCCGCTTCCAGTCTTCCGCCGTCAT GGCCCTGCAGGAGGCATCTGAGGCTTACCTCGTCGGTCTCTTCGAGGATACCAACTTGTGCGCCATCCACGC CAAGCGAGTCACTATTATGCCYAAGGATATGTCATASCTGTTTCY
Замечание
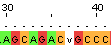
На мой взгляд, стоит отметить то печальное обстоятельство, что прогамма Jalview не поддерживает использование вырожденных кодов. В нашем случае это не помешало построению выравнивания, поскольку вырожденные коды ставились мной при редактировании в тех позициях, для которых отсутствовал соответствующий участок на другой цепи. Соответственно в выравнивании на этих местах в любом случае находятся гэпы, и нам в принципе неважно, какие нуклеотиды могут быть напротив них (см. Рис. 1).
Рис. 1
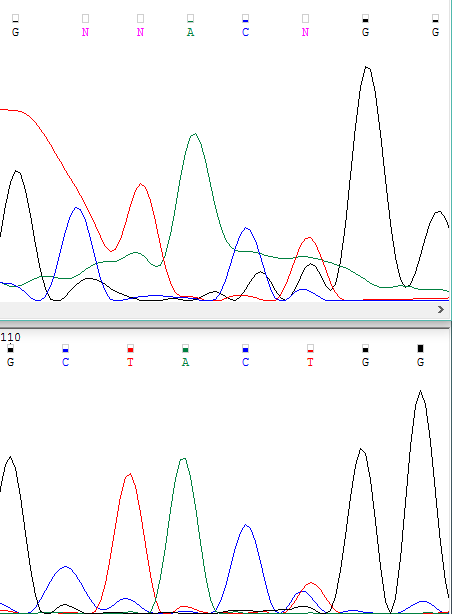
Но встретилось и исключение — рассматриваемые участки были прочтены в обеих последовательностях, но в одной и той же позиции там возникли проблемы. Соответственно, однозначно определить нуклеотид не удаётся и в прямой, и в обратной цепи (см. Рис. 3). Тогда в выравнивании имеем два вырожденных кода друг напротив друга (см. Рис. 2).
Рис. 2

Рис. 3
Три неопределённых нуклеотида. Два первых определяются по обратной-комплементарной цепи однозначно, а третий на ней вышел плохо, и проверить уже не представляется возможным.
Таким образом, в моём случае при построении выравнивания ничего плохого не произошло. Но есть ряд существенных недочётов, связанных с нераспознаванием вырожденных кодов программой Jalview. Так, например, при построении выравниваний может быть выбрано не наилучшее из возможных, потому что не учитывается сходство. Когда мы строим выравнивания белков, у нас есть определенная матрица, задающая сходство аминокислот. Соответственно, выравнивания, в которых аминокислоты схожи между собой, оказываются весомее.
В случае работы с нуклеотидными последовательностями, содержащими вырожденные коды, это не учитывается. При выборе итогового выравнивания наравне будут идти выравнивание, в котором Y (C/T) стоит напротив C, и выравнивание, в котором напротив этого Y оказался G. Хотя очевидно, предпочтение надо отдать первому варианту.
Задание 2. Пример нечитаемой хроматограммы
На рисунке ниже приведён пример действительно плохой по качеству хроматограммы. Её файл NN_G10.ab1 доступен для скачивания.
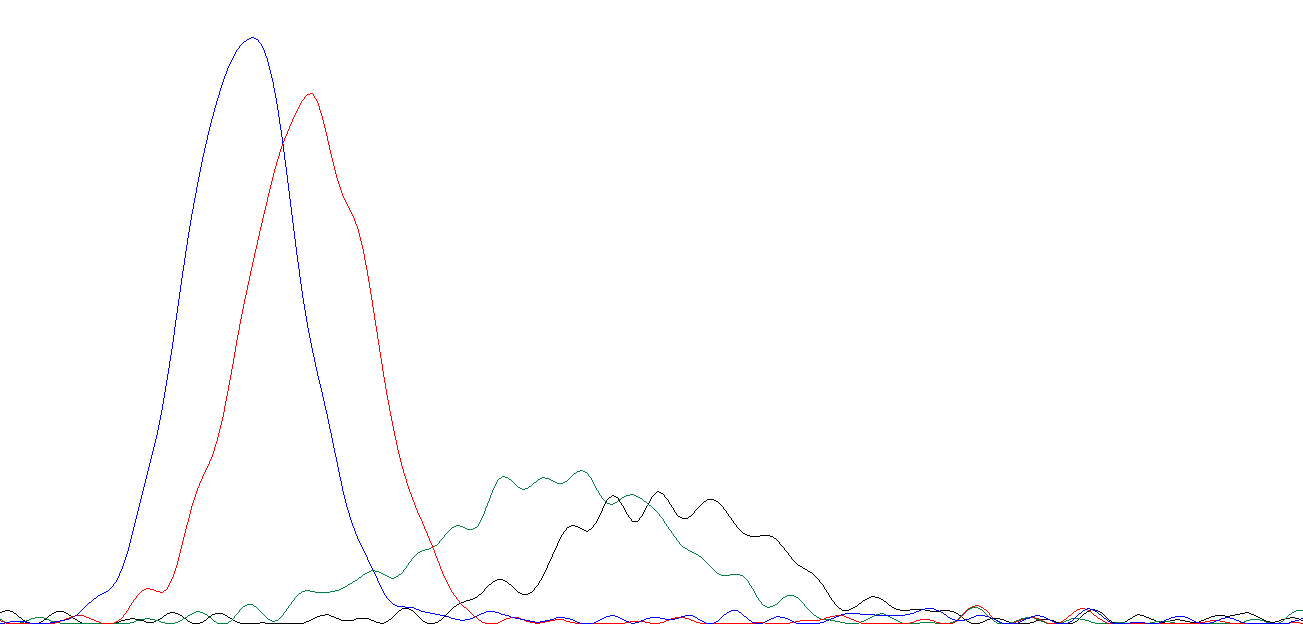
Как видим, автоматического прочтения не произошло. И это неудивительно, при просматривании хроматограммы на всем протяжении шум неотличим от сигнала, встречаются огромные пики, размытые и нечёткие. Возможно, в образце содержались разные цепочки ДНК.
[1] IUPAC ambiguity codes