Кристалл
1. Взаимодействия с субъединицами соседних ячеек
Идентификатор изучаемого белка в банке данных PDB: 5W0H.
Секция CRYST1 в [pdb]-файле описывает геометрические параметры кристаллографической ячейки[1].
Для моего белка содержимое этого поля выглядит следующим образом:
CRYST1 33.430 38.203 72.722 90.00 90.00 90.00 P 21 21 21 4
Первые три числа (1-3) описывают величины модулей векторов a, b, c, которые представляют собой тройку некомпланарных векторов, составляющих базис кристаллографической ячейки. Они описывают трансляционные симметрии кристалла. Имеют размерность [Å].
Следующие три числа (4-6) описывают величины углов α, β, γ между указанными векторами и выражаются в градусах. В силу того, что в нашем примере все углы прямые, кристаллографическая ячейка эксперимента представляет собой прямоугольный параллелепипед.
Далее следуют четыре значения: P 21 21 21.
Они описывают тип симметрии ячейки в формате нотации Германа-Могена[2] (англ. Hermann–Mauguin).
В последнем же числе закодировано так называемое Z value, а именно — число цепей в ячейке (для рассматриваемой структуры: 4).
Для восстановления соседних образов ассиметрической единицы в PyMol использовалась команда symexp.
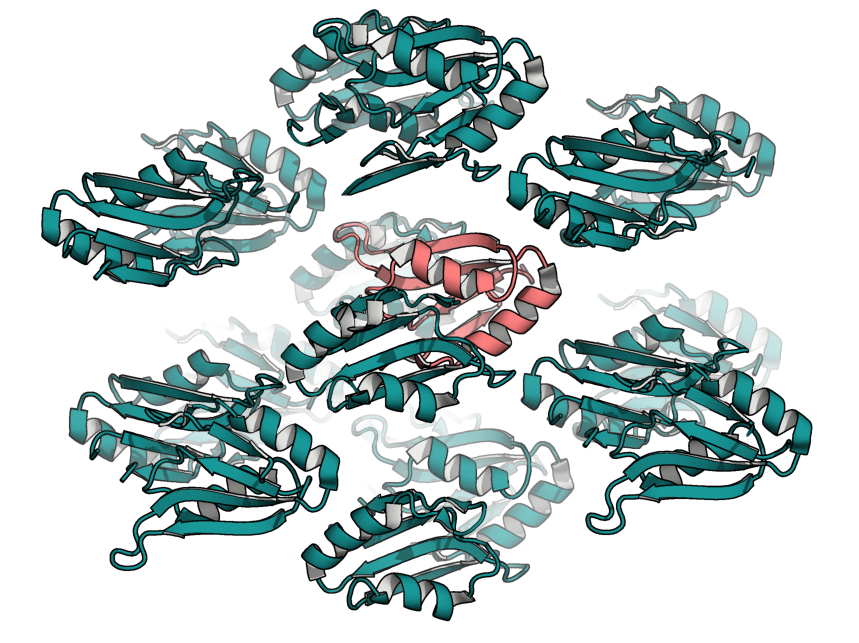
Результат приведен на Рис. 1. Также показаны границы элементарной ячейки (см. Рис.2).
Рис. 1. Структура кристалла: несколько ячеек в окружении 12Å
|
|
Розовым цветом выделена ассиметрическая единица.
Рис. 2. Структура кристалла: несколько ячеек в окружении 25Å

Обведены границы одной элементарной ячейки кристалла.
Далее был проведен поиск контактов (полярных связей) между соседними по кристаллу субъединицами. Оказалось, что одна ассиметрическая единица взаимодействует с двумя другими. Иллюстрации при разных положениях камеры приведены на Рис. 3.
Рис. 3. Контакты ассиметрической единицы с соседями по кристаллу
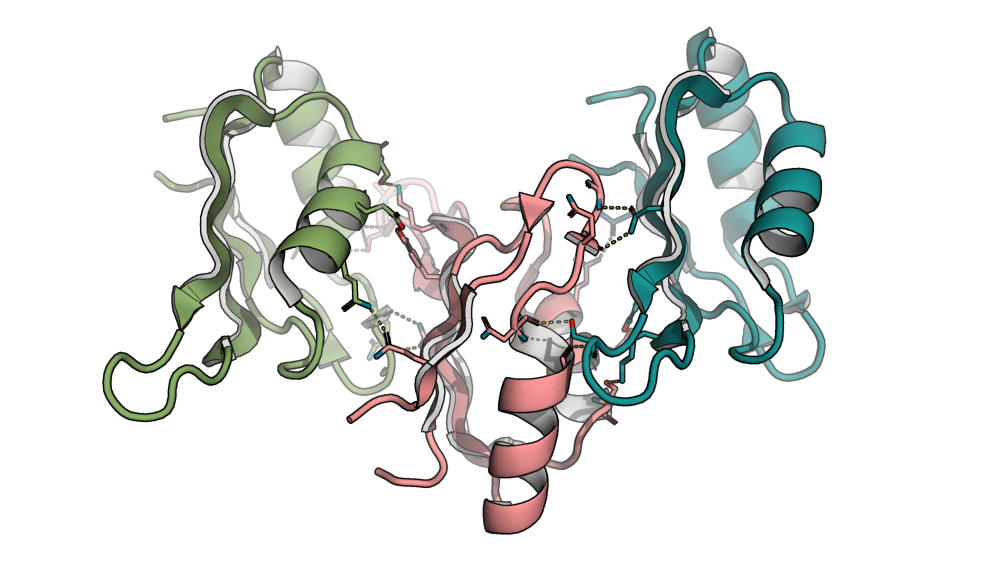
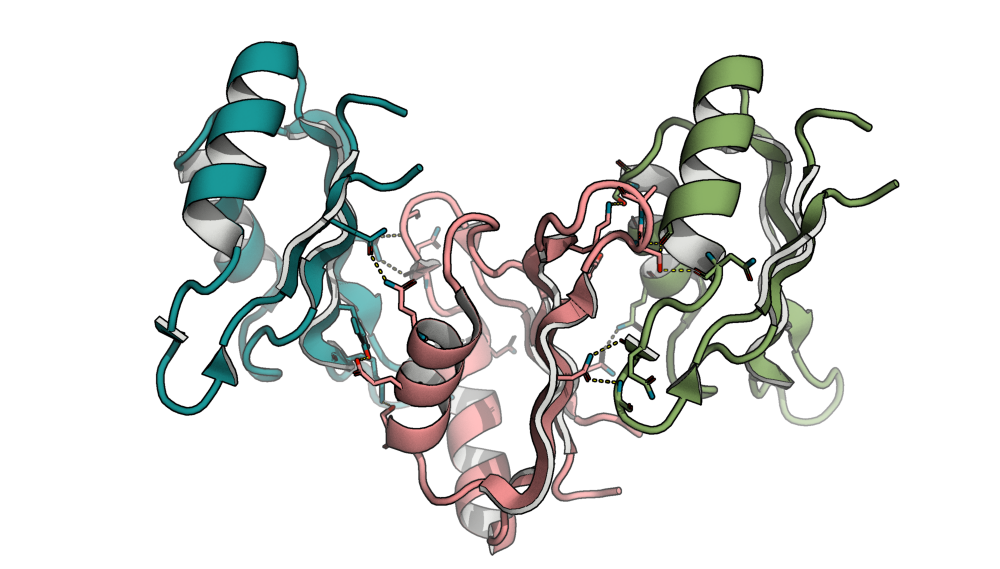
Найденные контакты исходной (розовой) ассиметрической единицы с синей и зелёной единицами представлены на Рис. 4 и Рис. 5 соответственно.
Рис. 4. Визуализация контактов с соседней единицей #1
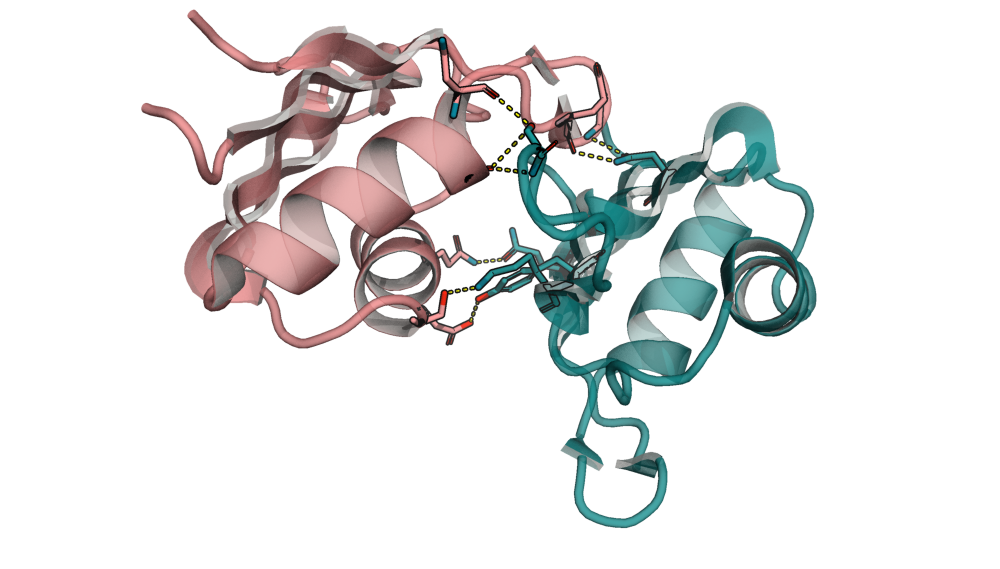
Рис. 5. Визуализация контактов с соседней единицей #2
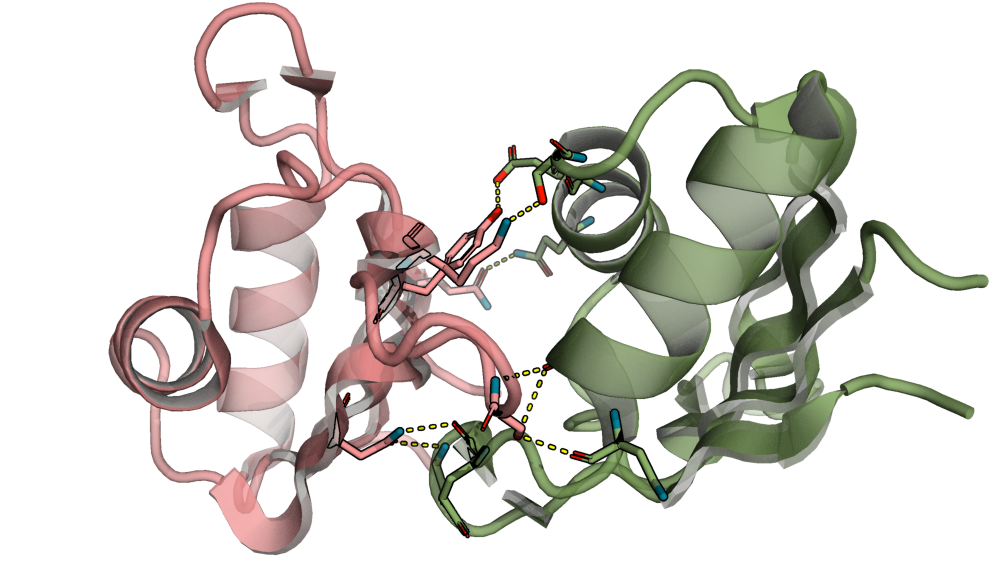
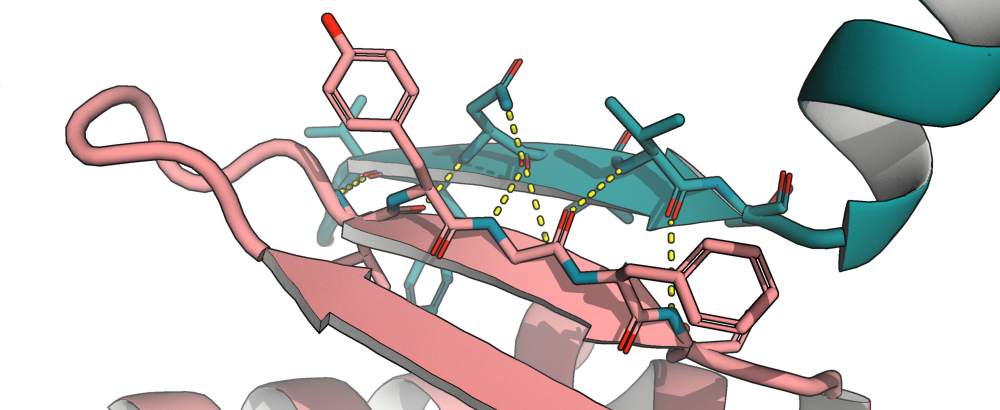
Анализ двух поверхностей контакта показывает, что связи одни и те же в обоих случаях. Это 8 водородных связей, созданных как атомами боковых цепочек, так и атомами остова полипептидной цепи.
Однако, биологичность данных связей остается под вопросом. В PDB сообщается, что биологическая сборка (англ. biological assembly) состоит из одной ассиметрической единицы. При этом экспериментальной проверки данного факта не проводилось: это отмечено в поле "Biological Assembly Evidence".
На самом деле[3] изучаемая структура представляет собой один домен большой субъединицы U2AF2 фактора сплайсинга. При этом данный фактор организован так, что рассматриваемый домен (RRM2) встречается там один раз. Узнавание РНК комплексом U2AF2 реализуется парой RRM1-RRM2, где RRM1 схож, но не эквивалентен RRM2. Возможно, было бы интересно посмотреть на структурную организацию такого RRM1-RRM2 комплекса и узнать, насколько паттерн связей совпадает с наблюдаемым в кристалле RRM2. Но на данный момент нет никаких оснований считать контакты соседних ассиметрических единиц аналогичным наблюдаемым в природе при, например, олигомеризации RRM2. В литературе не отмечены свидетельства протекания подобного процесса.
2. Странное поведение X-Ray структур
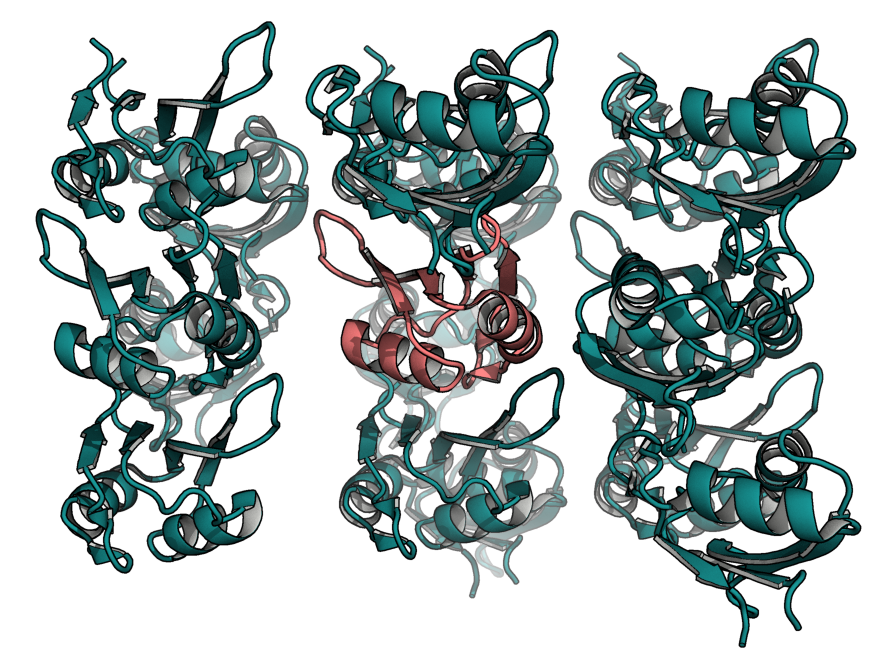
Для рассмотрения была взята структура 1MNM, представленная комплексом белка с ДНК. Общий вид её представлен на Рис. 6.
Рис. 6. Структура 1MNM
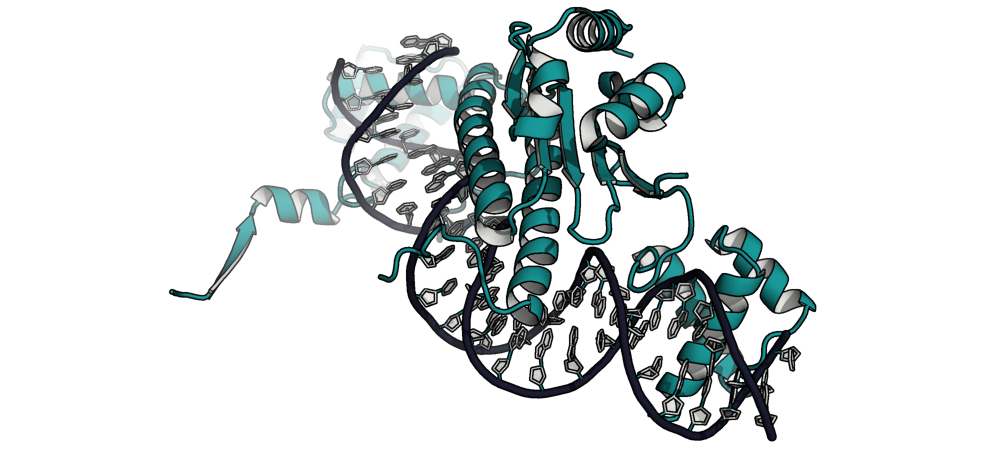
Можно заметить, что один тяж бета-листа (с левого края) занимет крайне странное пространственное положение. Для выяснения причин подобного поведения были восстановлены соседние ячейки кристалла. Изображение двух соседних субъединиц приведено на Рис. 7.
Рис. 7. Соседствующие в кристалле субъединицы
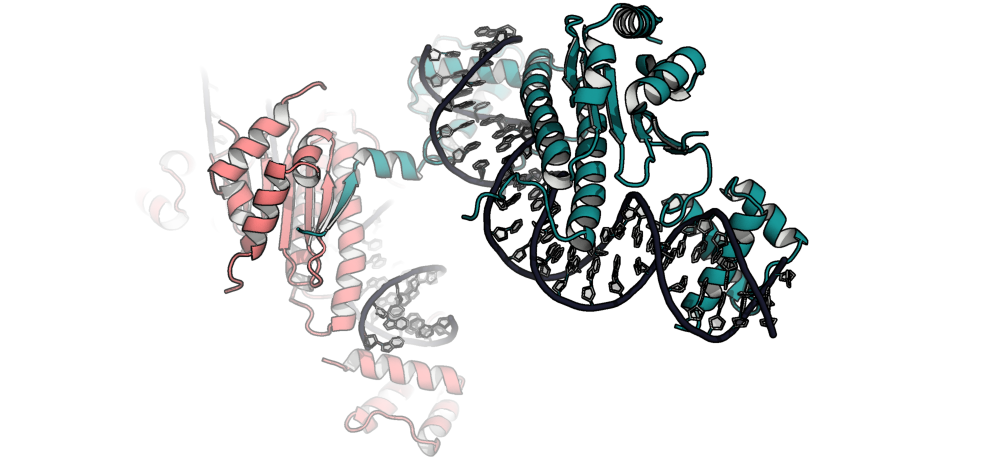
Становится очевидным, что данный тяж является одной из складок бета-листа, образующегося при спаривании двух отдельных ассиметрических единиц. Для наглядности были также проиллюстрированы контакты, опосредующие образование бета-листа (Рис. 8).
Рис. 8. Контакт между двумя единицами
[1] WWPDB: Crystallographic and Coordinate Transformation Section
[2] Wiki: Hermann–Mauguin notation
[3] Cancer-Associated Mutations Mapped on High-Resolution Structures of the U2AF2 RNA Recognition Motifs