PDB-файл
1. Occupancy
PDB ID: 5YCE с разрешением 0.77Å. Ниже приведена выдержка из файла, иллюстрирующая наличие остатка GLN8 в двух альтернативных положениях (A и B) с occupancy соответствующих атомов в 0.56 и 0.44 соответственно.
ATOM 71 N AGLN A 8 20.283 -1.236 -11.217 0.56 7.41 N ANISOU 71 N AGLN A 8 522 1191 1101 36 -99 -129 N ATOM 72 N BGLN A 8 20.250 -1.258 -11.233 0.44 7.52 N ANISOU 72 N BGLN A 8 663 1112 1081 83 -33 -162 N ATOM 73 CA AGLN A 8 20.872 -1.675 -12.477 0.56 8.23 C ANISOU 73 CA AGLN A 8 647 1278 1200 19 34 -238 C ATOM 74 CA BGLN A 8 20.836 -1.591 -12.516 0.44 7.82 C ANISOU 74 CA BGLN A 8 589 1218 1163 117 -43 -213 C ATOM 75 C AGLN A 8 19.787 -2.300 -13.369 0.56 7.53 C ANISOU 75 C AGLN A 8 530 1146 1183 205 -52 -290 C ATOM 76 C BGLN A 8 19.826 -2.354 -13.392 0.44 7.65 C ANISOU 76 C BGLN A 8 557 1097 1254 160 3 -282 C ATOM 77 O AGLN A 8 19.687 -1.977 -14.565 0.56 7.52 O ANISOU 77 O AGLN A 8 569 1174 1114 187 47 -323 O ATOM 78 O BGLN A 8 19.815 -2.180 -14.607 0.44 7.60 O ANISOU 78 O BGLN A 8 579 1012 1298 -8 130 -421 O ATOM 79 CB AGLN A 8 22.024 -2.665 -12.239 0.56 8.94 C ANISOU 79 CB AGLN A 8 716 1361 1321 160 -27 -261 C ATOM 80 CB BGLN A 8 22.176 -2.325 -12.399 0.44 8.15 C ANISOU 80 CB BGLN A 8 510 1347 1238 125 -314 -178 C ATOM 81 CG AGLN A 8 22.757 -3.075 -13.539 0.56 11.24 C ANISOU 81 CG AGLN A 8 1214 1499 1556 216 -39 -205 C ATOM 82 CG BGLN A 8 22.938 -2.322 -13.725 0.44 10.31 C ANISOU 82 CG BGLN A 8 945 1477 1493 200 -76 124 C ATOM 83 CD AGLN A 8 22.059 -4.179 -14.343 0.56 12.69 C ANISOU 83 CD AGLN A 8 1378 1635 1810 206 203 -119 C ATOM 84 CD BGLN A 8 24.274 -3.016 -13.635 0.44 11.88 C ANISOU 84 CD BGLN A 8 1206 1646 1662 126 298 224 C ATOM 85 OE1AGLN A 8 21.320 -5.003 -13.797 0.56 14.93 O ANISOU 85 OE1AGLN A 8 1725 1919 2029 145 356 -27 O ATOM 86 OE1BGLN A 8 25.224 -2.668 -14.328 0.44 13.10 O ANISOU 86 OE1BGLN A 8 1319 1854 1804 293 337 346 O ATOM 87 NE2AGLN A 8 22.324 -4.216 -15.653 0.56 13.69 N ANISOU 87 NE2AGLN A 8 1661 1756 1783 371 424 -92 N ATOM 88 NE2BGLN A 8 24.354 -4.003 -12.769 0.44 12.08 N ANISOU 88 NE2BGLN A 8 1255 1661 1673 282 326 294 N
Высокое разрешение модели говорит в пользу существования в кристалле действительно двух типов молекул: 56% с A-GLN8 и 44% — с B-GLN8, положения атомов которых различаются.
2. Missing residues
PDB ID: 6MWR с разрешением 3.3Å. Ниже приведена выдержка из заголовка PDB-файла, которая подтверждает наличие не расшифрованных в ходе эксперимента остатков. Указано имя остатка, цепь, которой он принадлежит, и его предполагаемый номер в этой цепи.
M RES C SSSEQI REMARK 465 MET A 0 REMARK 465 ARG A 1 REMARK 465 GLN A 249 REMARK 465 SER A 250 REMARK 465 SER A 251 REMARK 465 ASN A 252 REMARK 465 ASP B 98 REMARK 465 MET B 99 REMARK 465 ALA C 1 REMARK 465 GLY C 2 REMARK 465 ILE C 225 REMARK 465 LYS C 226 REMARK 465 THR C 227 REMARK 465 ASP C 228 REMARK 465 VAL C 229 REMARK 465 ILE C 230 REMARK 465 THR C 231 REMARK 465 MET C 232 REMARK 465 ASP C 233 REMARK 465 PRO C 234 REMARK 465 LYS C 235 REMARK 465 ASP C 236 REMARK 465 ASN C 237 REMARK 465 ALA C 238 REMARK 465 SER C 239 REMARK 465 GLY C 240 REMARK 465 LEU C 241 REMARK 465 VAL C 242 REMARK 465 PRO C 243 REMARK 465 ARG C 244 REMARK 465 ALA D 4 REMARK 465 VAL D 210 REMARK 465 LYS D 211 REMARK 465 THR D 212 REMARK 465 ASP D 213 REMARK 465 SER D 214 REMARK 465 THR D 215 REMARK 465 ASP D 216 REMARK 465 HIS D 217 REMARK 465 VAL D 218 REMARK 465 LYS D 219 REMARK 465 PRO D 220 REMARK 465 LYS D 221 REMARK 465 GLU D 222 REMARK 465 THR D 223 REMARK 465 GLU D 224 REMARK 465 ASN D 225 REMARK 465 THR D 226 REMARK 465 LYS D 227 REMARK 465 GLN D 228 REMARK 465 PRO D 229 REMARK 465 SER D 230 REMARK 465 LYS D 231 REMARK 465 SER D 232 REMARK 465 ALA D 233 REMARK 465 SER D 234 REMARK 465 GLY D 235 REMARK 465 LEU D 236 REMARK 465 VAL D 237 REMARK 465 PRO D 238 REMARK 465 ARG D 239
Итого, был пропущен 61 остаток (при общем количестве их в 850). В структурах с низким разрешением не должно быть удивительным встретить подобную проблему.
3. Кристаллизованный vs природный белок
PDB ID: 6TLD с разрешением 1.61Å — это структура белка гистондеацетилазы 8 из организма Schistosoma mansoni в комплексе с неким ингибитором. Экспрессия белка была организована в E.coli.
Поле SEQADV отражает отличия между последовательностью из SEQRES-записи в PDB-файле и последовательностью из базы данных, указанной в поле DBREF (в нашем случае UNP — UniProt)[1].
Ссылка на UniProt-последовательность: (ID: A5H660_SCHMA).
Рис. 1. UniProt и PDB записи: соответствие
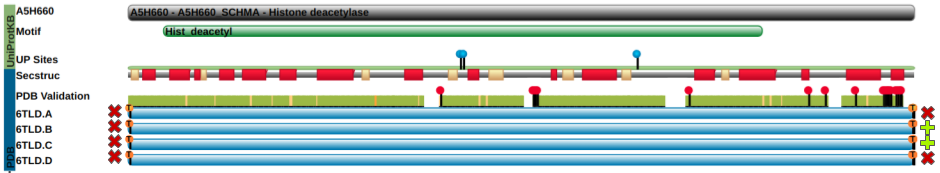
На Рис. 1, взятом из UniProt с изменениями, наглядно показано соотношение между PDB-структурами, UniProt-записью и гистондеацетилазным мотивом. Итак, каждая из 4 цепей структуры 6TLD (A, B, C, D) соответствует A5H660_SCHMA. Однако, можно заметить на концах репрезентации цепей маленькие индикаторы-флажки "T", которые обозначают наличие экспрессионного тэга. То же подтверждается записью из полей DBREF и SEQADV, приведенной ниже.
DBREF 6TLD A 1 440 UNP A5H660 A5H660_SCHMA 1 440 DBREF 6TLD B 1 440 UNP A5H660 A5H660_SCHMA 1 440 DBREF 6TLD C 1 440 UNP A5H660 A5H660_SCHMA 1 440 DBREF 6TLD D 1 440 UNP A5H660 A5H660_SCHMA 1 440 SEQADV 6TLD HIS A 0 UNP A5H660 EXPRESSION TAG SEQADV 6TLD GLY A 441 UNP A5H660 EXPRESSION TAG SEQADV 6TLD SER A 442 UNP A5H660 EXPRESSION TAG SEQADV 6TLD LEU A 443 UNP A5H660 EXPRESSION TAG SEQADV 6TLD VAL A 444 UNP A5H660 EXPRESSION TAG SEQADV 6TLD PRO A 445 UNP A5H660 EXPRESSION TAG SEQADV 6TLD ARG A 446 UNP A5H660 EXPRESSION TAG SEQADV 6TLD HIS B 0 UNP A5H660 EXPRESSION TAG SEQADV 6TLD GLY B 441 UNP A5H660 EXPRESSION TAG SEQADV 6TLD SER B 442 UNP A5H660 EXPRESSION TAG SEQADV 6TLD LEU B 443 UNP A5H660 EXPRESSION TAG SEQADV 6TLD VAL B 444 UNP A5H660 EXPRESSION TAG SEQADV 6TLD PRO B 445 UNP A5H660 EXPRESSION TAG SEQADV 6TLD ARG B 446 UNP A5H660 EXPRESSION TAG SEQADV 6TLD HIS C 0 UNP A5H660 EXPRESSION TAG SEQADV 6TLD GLY C 441 UNP A5H660 EXPRESSION TAG SEQADV 6TLD SER C 442 UNP A5H660 EXPRESSION TAG SEQADV 6TLD LEU C 443 UNP A5H660 EXPRESSION TAG SEQADV 6TLD VAL C 444 UNP A5H660 EXPRESSION TAG SEQADV 6TLD PRO C 445 UNP A5H660 EXPRESSION TAG SEQADV 6TLD ARG C 446 UNP A5H660 EXPRESSION TAG SEQADV 6TLD HIS D 0 UNP A5H660 EXPRESSION TAG SEQADV 6TLD GLY D 441 UNP A5H660 EXPRESSION TAG SEQADV 6TLD SER D 442 UNP A5H660 EXPRESSION TAG SEQADV 6TLD LEU D 443 UNP A5H660 EXPRESSION TAG SEQADV 6TLD VAL D 444 UNP A5H660 EXPRESSION TAG SEQADV 6TLD PRO D 445 UNP A5H660 EXPRESSION TAG SEQADV 6TLD ARG D 446 UNP A5H660 EXPRESSION TAG
Остатки 1-440 полностью соответствуют последовательности UniProt. Однако, сообщается о том, что на N-конец каждой цепи вводился "нулевой" His. В свою очередь, на каждый С-конец цепи вводился тэг из 6 остатков: 441-Gly Ser Leu Val Pro Arg-446. Сопоставление с Missing residues показало, что упомянутые нулевые гистидины не были расшифрованы в окончательной структуре, впрочем, как и С-концевые тэги для цепей A и D. Это отображено плюсами и крестиками на Рис. 1.
Для наглядности на Рис. 2 приведена структура цепи B и ее C-концевой тэг.
Рис. 2. Структура 6TLD (цепь B)
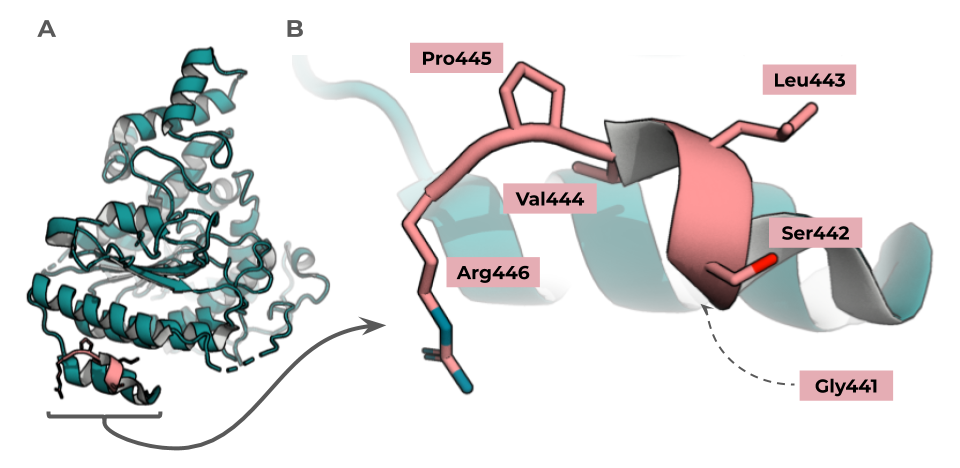
(А): Общая структура цепи, розовым выделен тэг; (B): Тэг и его аминокислотный состав; Gly направлен от зрителя.
4. Экстремальные B-факторы
PDB ID: 5UIW с разрешением 2.2Å — структура С-С рецептора хемокина 5 (CCR5) комплексе с антагонистом хемокина. Этот рецептор является трансмембранным белком, и для него было получена ориентация в мембране из базы данных OPM[2].
Указанный белок располагается в плазматической мембране эукариотических клеток, что проиллюстрировано на Рис. 5 с отрисовкой (взятых из OPM) положительной и отрицательной сторон мембраны.
Рис. 3. Структура 5UIW
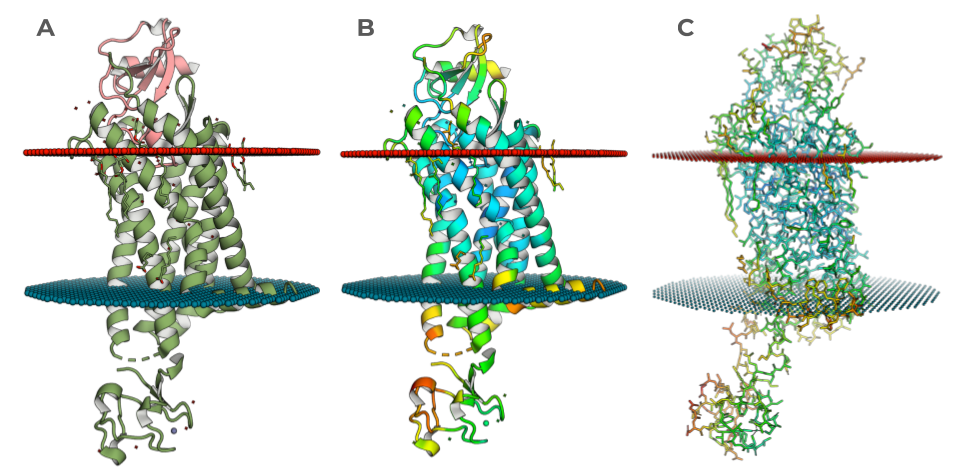
Показана структура комплекса в мембране (слой красных сфер: граница, обращенная к внеклеточному пространству, слой синих сфер: граница, обращенная к внутренней цитоплазме клетки).
(А): Зеленым: рецептор хемокина, розовым: антагонист хемокина; (B): Раскраска по величине B-фактора (более теплые цвета соответствуют бóльшим значениям);
(C): То же, палочная модель.
Хорошо прослеживается, как внутри мембраны преобладают низкие значения B-фактора, особенно при погружении глубже. Теплее становится в областях контакта белка и липидов мембраны. И уж совсем высокие значения наблюдаются там, где белок свободен от мембраны — будь то цитоплазма или внеклеточное пространство.
На Рис. 4 проиллюстрированы остатки, атомы которых попали в топ-10 самых низких и самых высоких значений B-фактора соответственно. Ими стали остатки (1) Phe109 из глубины трансмембранной части и (2) Asp1036 из участка, обращенного в цитоплазму.
Рис. 4. Экстремальные значения B-факторов
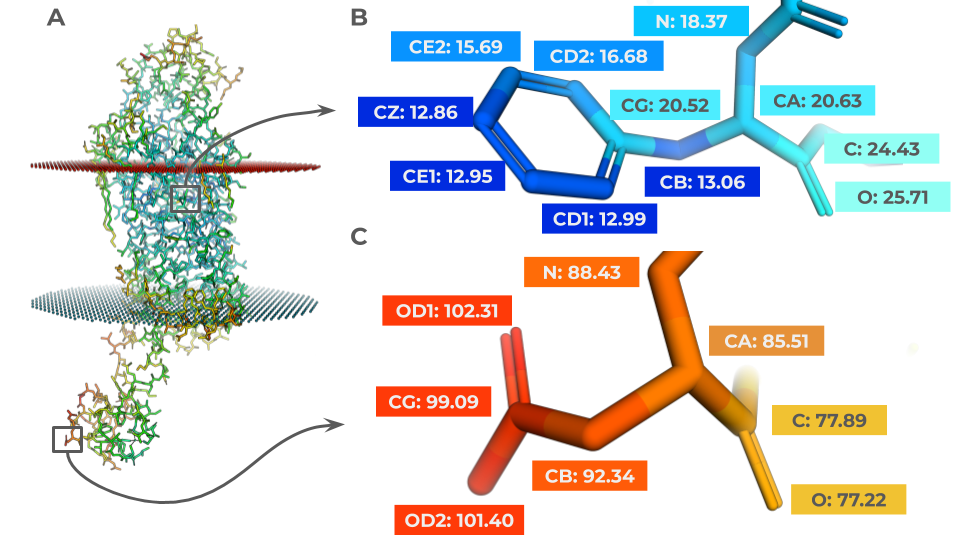
(A): Показана структура комплекса в мембране (слой красных сфер: граница, обращенная к внеклеточному пространству, слой синих сфер: граница, обращенная к внутренней цитоплазме клетки), вся структура приведена в палочной репрезентации и раскрашена по величине B-фактора (более теплые цвета соответствуют бóльшим значениям);
(B): Остаток Phe109 из рецептора, несущий атомы, попадающие в топ-10 обладающих самыми низкими значениями B-фактора;
(C): Остаток Asp1036 из рецептора, несущий атомы, попадающие в топ-10 обладающих самыми высокими значениями B-фактора;.
Наблюдаемая зависимость легко вписывается в понимание сути B-фактора: он отражает, насколько "растушевано и размазано" распределение электронной плотности из-за движений (колебаний) атома. Практически фиксированные в глубине трансмембранного участка атомы Phe109 скованы, в то время как атомы Asp1036, смотрящего в цитоплазму, имеют возможность смещаться (и, судя по величине В-фактора, очень активно этим занимаются).
[1] wwPDB Format
[2] 5uiw: OPM