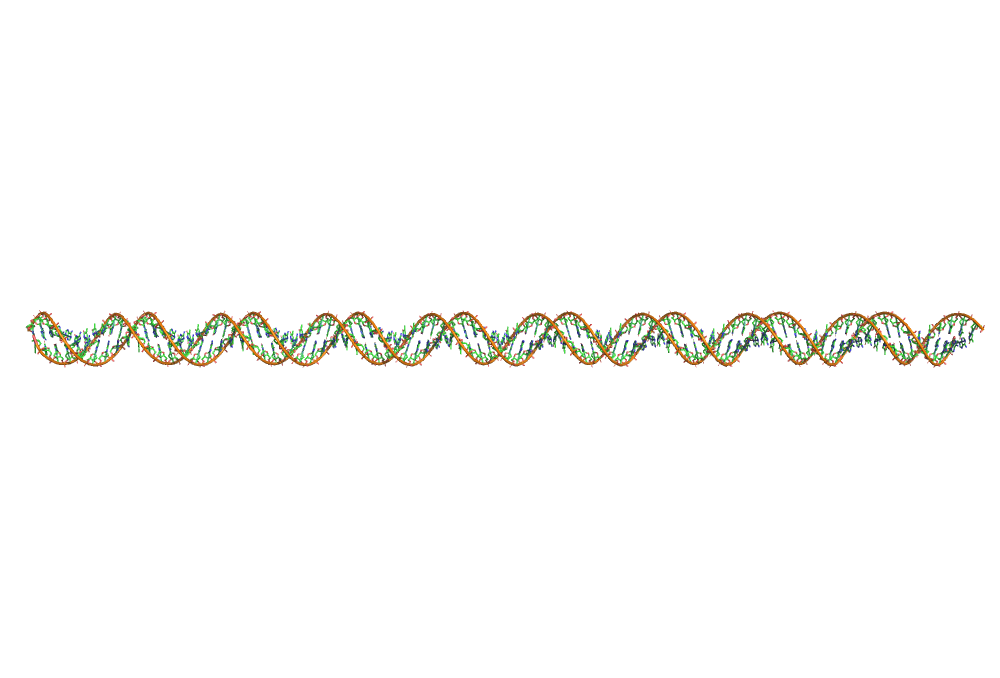Практикум 1¶
Задание 1. Знакомство с PyMOL¶
Импортируем нужное для запуска PyMOL.
import __main__
__main__.pymol_argv = ['pymol', '-x']
import pymol
pymol.finish_launching()
from pymol import cmd, stored
Начало¶
Скачиваем структуру 1cll, показываем линиями белок. Приближаем камеру к остаткам 4 и 5. Сейчас у нас есть одно состояние (state) — 1. Далее mset0 устанавливает соответствие между состояниями и кадрами анимации. mset 1 x1000 "растягивает" состояние 1 на тысячу кадров. Закрепляем это командой mview store.
cmd.do('''
fetch 1cll, async=0
as lines, n. C+O+N+CA
zoom i. 4+5
mset 1 x1000
mview store''')
Исследование итерации по остаткам¶
Проитерируемся по Cα-атомам остатков и сохраним их номера.
stored.r = []
cmd.iterate('1cll and n. CA','stored.r.append(int(resi))')
colors задает пространство R (от R — red) со 152 точками. Значения R равномерно распределены от 1 до 0.5.
G и B константны (0.5 и 0.75 соответственно).
В цикле мы проходим по всем остаткам и каждому задаем новый цвет отображения cartoon по очереди.
Включаем отображение cartoon для всего.
import numpy as np
length = len(stored.r)
colors = np.linspace(1,0.5, length)
for k,i in enumerate(stored.r):
cmd.set_color('col%d' %k, [colors[k],0.5,0.75])
print([1,1,colors[k]])
cmd.set('cartoon_color','col%d' % k ,'resi %d' % i)
cmd.show_as('cartoon','all')
Упражнение с movie¶
Выбираем каждый 10 кадр и приближаем камеру к остаткам с номерами i и i+7. Получается мини-фильм.
for i in range(length):
cmd.frame((10*i)+1)
cmd.zoom( 'n. CA and i. %d+%d' % (i,i+7))
cmd.mview('store')
А вот и результат!
from IPython import display
display.HTML('<img src="img/movie1.gif">')
Задание 2. Работа в PyMOL¶
Пункты 1-3¶
Подготавливаем PyMOL для создания анимационного ролика посложнее.
cmd.do('''reinitialize
set matrix_mode, 1
set movie_panel, 1
set scene_buttons, 1
set cache_frames, 1
config_mouse three_button_motions, 1''')
Загружаем структры: исходную и предварительно мутированную инструментом Mutagenesis
cmd.do('''bg_color white
set cartoon_side_chain_helper, 1
set cartoon_transparency, 0.1
load files/1lmp.pdb, orig
load files/1lmp_D52A.pdb, mut
translate [0,10,-30], mut
zoom all
center all
select wat, resn HOH
hide everything, wat
select lig_orig, orig and chain B
select lig_mut, mut and chain B
select prot_orig, orig and chain A and not wat
select prot_mut, mut and chain A and not wat
util.cbap prot_orig
util.cbam prot_mut
util.cbas lig*
select r, orig and resi 52
select r_mut, mut and resi 52
show sticks, r
show sticks, r_mut
deselect
''')
Вручную выбираем неплохой ракурс камеры и получаем его параметры через cmd.get_view(). Эти параметры копипастим и передаем далее в cmd.set_view(...)
cmd.set_view ('''\
0.237, 0.942, 0.238,\
-0.823, 0.065, 0.565,\
0.516, -0.329, 0.79,\
0.0, 0.0, -107.826,\
16.946, 66.04, 22.63,\
58.073, 157.58,-20.0''')
Задаем длину ролика в 700 кадров (фреймов). Создаем сцену 1, привязываем ее к фрейму 1, растягиваем на 30 кадров.
cmd.do('''
mset 1 x700
frame 1
mview store, object=orig
mview store, object=mut
scene 001, store, message="Original protein is in purple, mutated — in magenta."
mview store, scene=001
frame 30
mview store, object=orig
mview store, object=mut
mview store, scene=001
''')
На 150 кадре хотим видеть структуры, наложенные друг на друга. Создаем сцену 2, привязываем к кадру 150, интерполируем. Еще 30 кадров для небольшой паузы.
cmd.do('''frame 150
super mut, orig
mview store, object=orig
mview store, object=mut
scene 002, store, message="Now we overlap the two structures"
mview store, scene=002
mview interpolate, object=mut
''')
cmd.do('''frame 180
mview store, object=orig
mview store, object=mut
mview store, scene=002
mview reinterpolate, object=orig
mview reinterpolate, object=mut
''')
Задаем удачный вид, выбрав его вручную. Приближаем интересный нам остаток 52.В оригинальной структуре это аспартат (D, Asp), в мутированной — аланин (A, Ala).
cmd.do('''frame 210
set_view (\
0.24, 0.94, 0.24,\
-0.82, 0.07, 0.56,\
0.52, -0.33, 0.79,\
0.00, 0.00, -31.35,\
20.77, 55.81, 25.53,\
19.84, 42.86, -20.00 )
mview store, object=orig
mview store, object=mut
scene 003, store, message="Zoom to residue 52"
mview store, scene=003
mview reinterpolate, object=orig
mview reinterpolate, object=mut
''')
cmd.do('''frame 230
mview store, object=orig
mview store, object=mut
mview store, scene=003
''')
Трижды меняем отображение -- чередуются оригинальный белок и мутированный. В конце плавно возвращаемся в исходное состояние.
for i in range(3):
frame_scene_orig = 250 + i * 120
frame_scene_mut = 270 + i * 120
print(frame_scene_orig, frame_scene_mut)
cmd.do(f'''frame {frame_scene_orig}
hide everything, prot_mut
show cartoon, prot_orig
show sticks, r
mview store, object=orig
mview store, object=mut
scene 004, store, message="Original structure with Asp52"
mview store, scene=004
mview reinterpolate, obj=orig
''')
cmd.do(f'''frame {frame_scene_orig+120}
mview store, object=orig
mview store, object=mut
mview store, scene=004
mview reinterpolate, object=orig
mview reinterpolate, object=mut
''')
cmd.do(f'''frame {frame_scene_mut}
hide everything, prot_orig
show cartoon, prot_mut
show sticks, r_mut
mview store, object=orig
mview store, object=mut
scene 005, store, message="Mutated structure with Ala52"
mview store, scene=005
''')
cmd.do(f'''frame {frame_scene_mut+120}
mview store, object=orig
mview store, object=mut
mview store, scene=005
mview reinterpolate, object=orig
mview reinterpolate, object=mut
''')
cmd.do(f'''frame {frame_scene_mut+120}
show cartoon, prot_orig
show sticks, r
mview store, object=orig
mview store, object=mut
scene 006, store, message="Going back to initial state"
mview store, scene=006
''')
display.HTML('''
<div align="middle">
<video width="80%" controls>
<source src="img/movie_2.mp4" type="video/mp4">
</video></div>''')
Пункт 4. Присоединение метки¶
Загрузим структуры 1LMP и 6-Tamra. Белок, как обычно, в формате .pdb, метка TAMRA — в .sdf.
cmd.do('''reinitialize
bg_color white
set cartoon_side_chain_helper, 1
set cartoon_transparency, 0.1
load files/1lmp.pdb, 1LMP
load files/6-Tamra_3d.sdf, TAMRA
select wat, resn HOH
hide everything, wat
select lig, 1LMP and chain B
select prot, 1LMP and chain A and not wat
util.cbap prot
util.cbas lig
select r, orig and resi 52
show sticks, r''')
Выберем удачный вид и подпишем интересующие нас атомы метки TAMRA.
cmd.do('''set_view (\
0.01, -0.86, 0.5,\
0.45, 0.45, 0.77,\
-0.89, 0.22, 0.39,\
0.00, 0.00, -34.52,\
-0.34, 0.48, 0.00,\
25.98, 43.05, -20.00 )
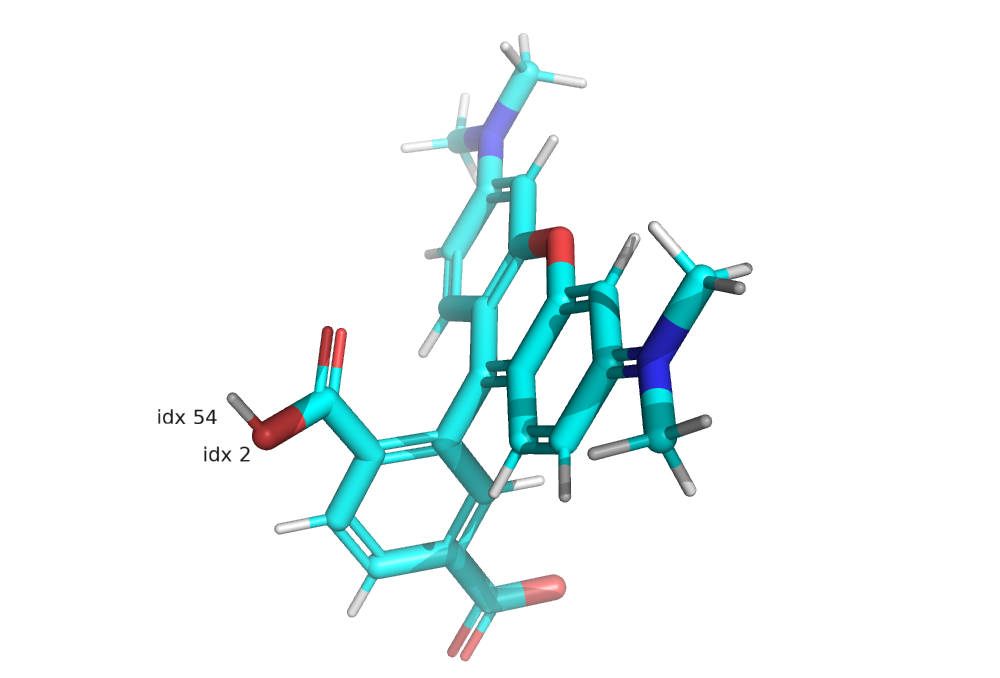
label TAMRA and idx 54, "idx 54"
label TAMRA and idx 2, "idx 2"
set label_position,(-1.15,-1.05,3)
''')
Вот что получилось:
display.HTML('<img width=50% src="img/tamra.png">')
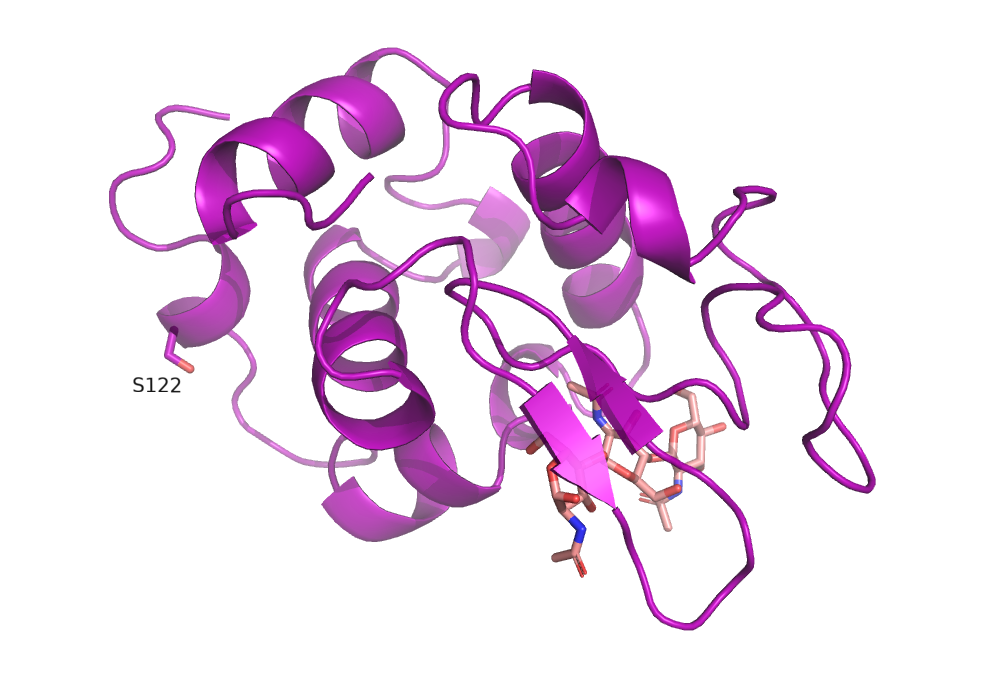
Теперь выберем удачный вид, чтобы показать остаток серина, к которому будет произведено прикрепление метки по механизму образования сложноэфирной связи.
cmd.do('''
set_view (\
-0.13, -0.43, 0.89,\
-0.02, -0.9, -0.44,\
0.99, -0.08, 0.11,\
0.00, 0.00, -103.14,\
14.93, 52.33, 21.15,\
75.44, 130.84, -20.00 )
select Ser, /1LMP//A/SER`122
show sticks, Ser
label Ser and n. OG, "S122"
deselect
''')
Видим подписанный остаток серина S122 в структуре 1LMP.
display.HTML('<img width=70% src="img/1LMP_S122.png">')
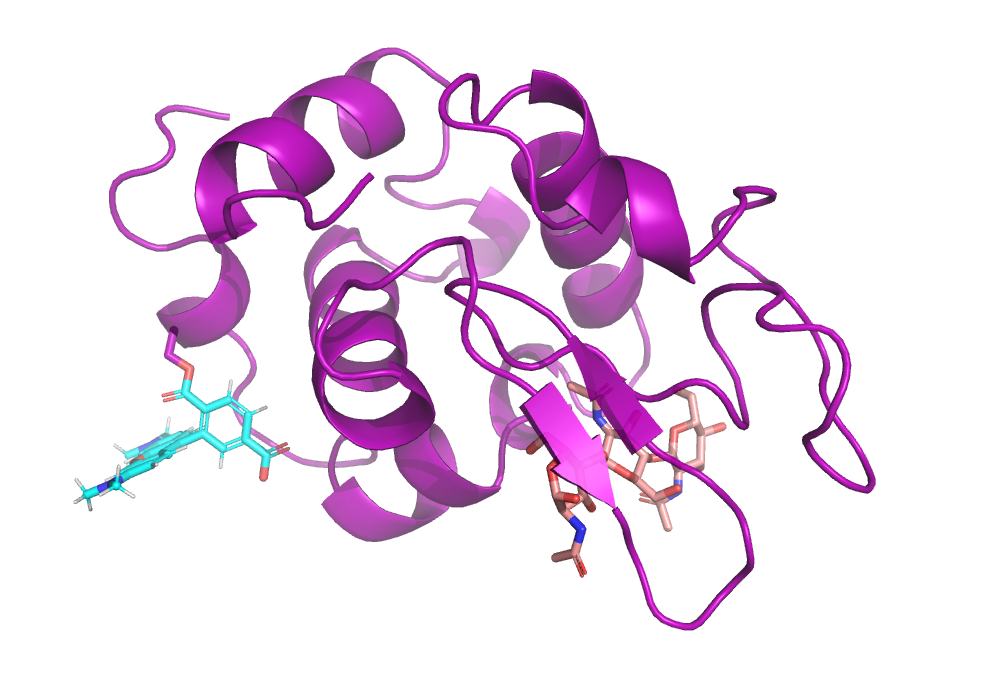
Присоединяем метку к кислородному атому OG серина 122 через атом углерода (TAMRA, idx 28), используя команды fuse и torsion.
cmd.do('''
remove TAMRA and idx 2+54
''')
cmd.fuse('TAMRA and idx 28', '/1LMP//A/SER`122/OG')
cmd.torsion(30)
cmd.hide('labels')
Получено изображение комплекса: фиолетовым — белок, розовым — его лиганд, бирюзовым — флуоресцентная метка TAMRA.
display.HTML('<img width=70% src="img/1LMP_tamra.png">')
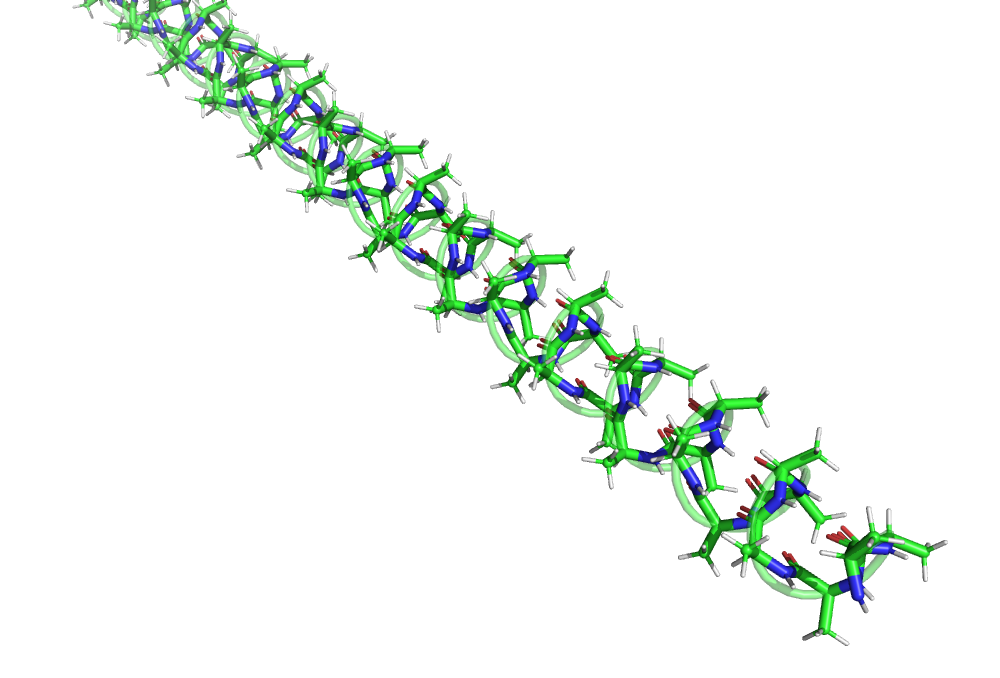
Пункт 5. Построение полиаланиновой α-спирали
α-спираль обладает характерными значениями торсионов (это определенная область на карте Рамачандрана, построенной без учета специфических случаев -- глицина и пролина):
φ = -64 (± 7)
ψ = -41 (± 7)
phi = -64
psi = -41
cmd.do('''reinitialize
bg_color white
''')
cmd.fragment('ala', 'helix')
for i in range(2, 101):
cmd.edit(f'helix & i. {i} & n. C')
cmd.editor.attach_amino_acid('pk1', 'ala')
for i in range(2, 101):
cmd.set_dihedral(f'i. {i} & n. N',
f'i. {i} & n. CA',
f'i. {i} & n. C',
f'i. {i + 1} & n. N',
phi)
cmd.set_dihedral(f'i. {i} & n. C',
f'i. {i + 1} & n. N',
f'i. {i + 1} & n. CA',
f'i. {i + 1} & n. C',
psi)
cmd.show('cartoon', 'helix')
cmd.set('cartoon_transparency', 0.5)
Результат работы — полиаланиновая α-спираль:
display.HTML('<img width=50% src="img/polyalahelix.png">')
Пункт 6. Построение B-формы ДНК¶
Из задания: заготовок для нуклеиновых кислот нет. Cуть подхода состоит в определении матрицы превращения при совмещении одной пары нуклеотидов с последующей. После чего вы можете размножать эту пару и применять к ней матрицу превращения.
Команды: cmd.pair_fit("pair","nextpair"), trans=cmd.get_object_matrix("pair"), create, cmd.transform_selection()
Найдем структуру B-ДНК. В этом поможет сервис Web 3DNA 2.0. PDB ID 355D подходит, в чем можно убедиться в выдаче сервиса.
Выберем из нее две пары нуклеотидов и сохраним в отдельные объекты. Первую пару также запишем в объект pair1, который послужит нам затравкой при генерации ДНК.
cmd.do('''reinitialize
bg_color white
load files/355d.pdb
create pair, /355d//A/DA`6 or /355d//B/DT`19
create pair1, pair
create nextpair, /355d//A/DT`7 or /355d//B/DA`18''')
Сделаем совмещение пар и получим матрицу трансформации trans.
cmd.pair_fit('pair', 'nextpair')
trans = cmd.get_object_matrix('pair')
trans
Удаляем ненужное, оставляя только затравку — объект pair1.
cmd.do('''
delete 355d
delete pair
delete nextpair
select pair1
show sticks
orient
deselect
alter chain A & resi 6, resi=1
alter chain B & resi 19, resi=100
''')
Вот наша пара
display.HTML('<img width=30% src="img/pair1.png">')
Продублируем пару сто раз, применяя к каждой новой паре трансформацию с помощью ранее полученной матрицы.
for i in range(1, 100):
cmd.create(f'pair{i+1}', f'pair{i}')
cmd.transform_selection(selection=f'pair{i+1}', matrix=trans)
cmd.alter(f'pair{i+1} & chain A & resi {i}', f'resi={i+1}')
cmd.alter(f'pair{i+1} & chain B & resi {100-i+1}', f'resi={100-i}')
cmd.create('B-helix', 'pair*')
cmd.delete('pair*')
Добавим фосфодиэфирные связи остова.
for i in range(1, 100):
cmd.bond(f"/B-helix//A/DA`{i}/O3'", f"/B-helix//A/DA`{i+1}/P")
cmd.bond(f"/B-helix//B/DT`{100-i}/O3'", f"/B-helix//B/DT`{100-i+1}/P")
cmd.orient('B-helix')
Такая получается спираль:
display.HTML('<img width=70% src="img/b-helix.png">')