Обзор генома и протеома бактерии Burkholderia stabilis
Резюме
В данной работе исследуется геном и протеом бактерии Burkholderia stabilis. Приведены данные о его составе, предположительно найдены точки начала и конца репликации, рассмотрено распределение генов по цепям ДНК (отдельно для рРНК), длины межгенных промежутков и статистика белков по категориям достоверности существования.
Введение
Burkholderia stabilis - это грамотрицательная бактерия, принадлежащая к порядку Burkholderiales, класс Betaproteobacteria. Данный вид был идентифицирован в 2000 году и обнаружен в крови, слюне, дыхательных путях человека [1]. Устойчивы к многим антибиотикам: β-лактамам, аминогликозидам и др. Вместе с несколькими близкими видами из рода Burkholderia входит в состав т.н. комплекса Burkholderia cepacia, являющегося условно-патогенным. Для других бактерий рода также показана патогенность [2]. Отмечались случаи обнаружения B.stabilis отдельно и в составе комплекса B.cepacia в продуктах, не предназначенных для питания, в т.ч. на медицинском оборудовании [3]. У представителей рода, в т.ч. B.stabilis, обнаружены гены, отвечающие за т.н. “чувство кворума” [4]. Геном бактерии состоит из трех хромосом и плазмиды, его общий размер - около 8.5 млн. пар оснований [5].
Материалы и методы
Все данные о геноме бактерии, использованные в работе, были взяты из базы данных NCBI. При работе с электронными таблицами Google Sheets использовались следующие функции: MIN и MAX (п. 3.2, 3.3), COUNTIFS (п. 3.3), COUNTIF (п. 3.4), статистические функции MODE, MEDIAN, AVERAGE (п. 3.3), BINOM.DIST (для оценки вероятности получить такое же или более сильное различие случайно в п. 3.3), а также встроенный инструмент электронных таблиц - фильтры, сочетания клавиш CTRL+F (в особенности для нахождения индексов при локальных максимумах/минимумах в интегральном значении GC skew в пункте 3.2), CTRL+X (вырезание), CTRL+C (копирование), CTRL+V (вставка). Скрипты, использованные при получении результатов в пунктах 3.1 и 3.2, написаны на языке программирования Python и находятся в сопроводительных материалах.
Результаты и обсуждение
Общая информация о геноме
B.stabilis в составе генома имеет 3 хромосомы и плазмиду (далее нумеруются римскими цифрами от I до IV, соответственно). Основные данные представлены в таблице 0. Общий размер генома больше среднего для бактерий. GC-состав соотносится с данными для других бактерий рода Burkholderia [6].
| i |
ii |
iii | vi |
|
| размер, mb | 3.705 | 3.499 | 1.23 | 0.071 |
| доля gc-пар | 66.59% | 66.67% | 64.67% | 62.17% |
3.2 Точки начала и конца репликации на основании параметра GC skew
GC skew (дословно “GC перекос”) - соотношение между количеством гуанина и цитозина в ДНК, которое позволяет определить место начала репликации (origin, или oriC) и конца репликации (terminator, или ter). Оно вычисляется следующим образом:
GC skew = (n(G) − n(C))/(n(G) + n(C))
Для построения графика значения GC skew в каждом текущем окне суммировались. На графике (рис.1) можем отметить несколько локальных максимумов и минимумов, каждый из которых последовательно соответствует oriC и ter для I-VI хромосом B.stabilis. В таблице 1 представлены данные значения.
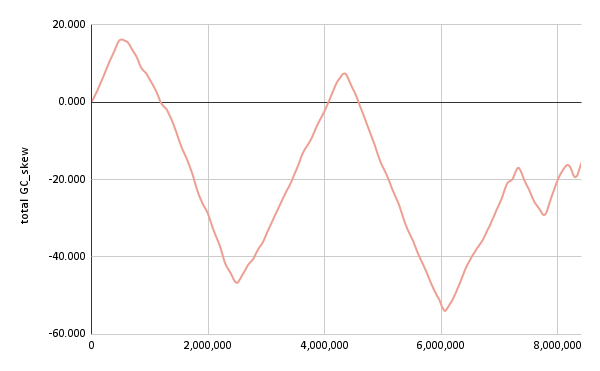
| i | ii | iii | vi | |
| oric | 512,000 | 4,341,000 | 7,323,000 | 8,172,000 |
| ter | 2,490,000 | 2,490,000 -2,491,000 | 7,760,000 -7,762,000 | 8,300,000 |
Полученные для точек OriC и ter значения не удалось сравнить с известными по причине отсутствия последних.
3.3 Распределение генов по цепям
Изучено распределение генов в геноме B.stabilis (см. таблицы 2, 3). Они расположены неравномерно: существенное различие (p-value < 0.05) наблюдается у белок-кодирующих генов на I и IV хромосомах. В первом случае на “-” цепи расположено больше подобных генов, а во втором - меньше.
| Хромосома | I | II | III | IV (плазмида) | ||||
| Класс гена/цепь | + | - | + | - | + | - | + | - |
| Белок-кодирующий | 1585 | 1821 | 1578 | 1517 | 528 | 544 | 59 | 26 |
| тРНК | 33 | 27 | 4 | 3 | 2 | 0 | 0 | 1 |
| псевдогены | 11 | 16 | 14 | 20 | 11 | 10 | 0 | 0 |
| рРНК | 6 | 6 | 0 | 3 | 3 | 0 | 0 | 0 |
| другие РНК | 2 | 2 | 0 | 0 | 0 | 0 | 0 | 0 |
| Хромосома/Класс гена | I | II | III | IV |
| Белок-кодирующий | 0.000056 | 0.281 | 0.647 | 0.00045 |
| тРНК | 0.519 | 1 | 0.5 | |
| псевдоген | 0.442 | 0.392 | 1 | |
| рРНК | 0.613 | 0.25 | 0.125 |
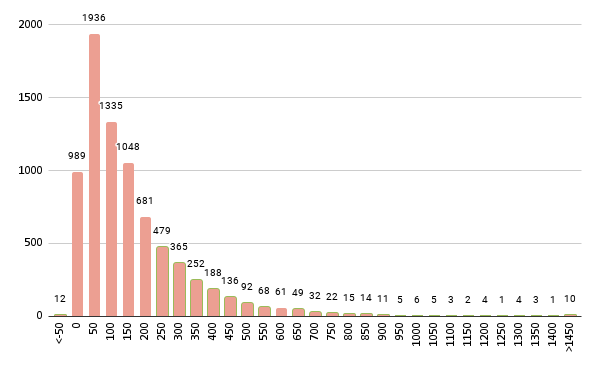
3.4 Анализ длин межгенных промежутков
Гистограмма длин межгенных промежутков приведена на рисунке 2. Наибольшее количество длин межгенных промежутков лежит в диапазоне 50-100 п.н. Мода равна -3, медиана — 86, среднее значение — 137,3. Минимальное расстояние отрицательное и равно -103 (хромосома I) , максимальное — 1969 (хромосома III). Данные значения единственны и соответствуют расстоянию между двумя генами белков, содержащих ABC-домен (ATP-binding cassette domain-containing protein), а также между геном белка инициации трансляции и гипотетическим белком, соответственно. Можно предположить, что в случае перекрывания аминокислотная последовательность, соответствующая общей части двух генов, входит в состав обоих белков. Следует отметить, что оба данных белка имеют совпадающие названия, поэтому обширное перекрывание объяснимо.
3.5 Гены рибосомальных РНК
Были найдены гены рибосомальных РНК и их расположение в геноме (см. таблицу 4). Мы выяснили, что рРНК размещены кластерами, т.е. группами на расстоянии до нескольких тысяч нуклеотидов на одной цепи, причем больше всего подобных кластеров расположено на хромосоме I. Отметим, что порядок расположения генов в них один и тот же: 16S, 23S, 5S РНК (5’ → 3’).
| Хромосома | Количество кластеров | Координаты начала кластера | Координаты конца кластера | Цепь |
| I | 4 | 2,090,399 | 2,095,507 | - |
| 2,311,251 | 2,316,512 | - | ||
| 2,586,723 | 2,591,922 | + | ||
| 3,644,325 | 3,649,586 | + | ||
| II | 1 | 2,011,331 | 2,016,592 | - |
| III | 1 | 684,293 | 689,492 | + |
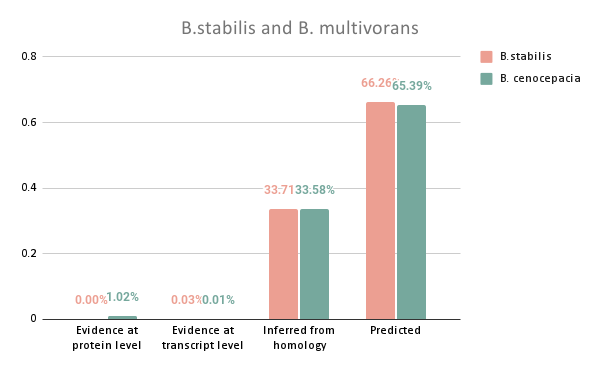
3.6 Статистика белков по категориям достоверности их существования
С использованием протеома B.stabilis, полученного из базы данных Uniprot, белки были сортированы по категориям достоверности их существования. Также была для сравнения значений выбрана другая более изученная бактерия из данного рода, Burkholderia cenocepacia (strain ATCC BAA-245 / DSM 16553 / LMG 16656 / NCTC 13227 / J2315 / CF5610). Она имеет полный протеом и большое количество записей с пометкой “Reviewed” в базе данных Uniprot. Заметим, что доли белков, существование которых предсказано (“Predicted”), основано на гомологии (“Inferred from homology”) или показано на уровне транскрипта (“Evidence at protein level”) различаются слабо (см. рис. 3). Однако у B.cenocepacia заметно выше доля белков, существование которых достоверно подтверждено (“Evidence at protein level”). Исходя из этого можем заключить, что протеом B.stabilis требует дальнейшего изучения.
Источики
- P. Vandamme, E. Mahenthiralingam, B. Holmes, et al. Identification and Population Structure of Burkholderia stabilis sp. nov. (formerly Burkholderia cepacia Genomovar IV) (2000). Journal of Clinical Microbiology, 38(3), 1042-1047
- Seth-Smith, H. M. B., Casanova, C., Sommerstein, R. et al. (2019). Phenotypic and Genomic Analyses of Burkholderia stabilis Clinical Contamination, Switzerland. Emerging Infectious Diseases, 25(6), 1084–1092. doi:10.3201/eid2506.172119
- Häfliger E, Atkinson A, Marschall J. Systematic review of healthcare-associated Burkholderia cepacia complex outbreaks: presentation, causes and outbreak control. Infect Prev Pract. 2020 Aug 13;2(3):100082. doi: 10.1016/j.infpip.2020.100082
- Lutter E, Lewenza S, Dennis JJ, Visser MB, Sokol PA. Distribution of quorum-sensing genes in the Burkholderia cepacia complex. Infect Immun. 2001, 69(7):4661-4666, doi: 10.1128/IAI.69.7.4661-4666.2001
- https://www.ncbi.nlm.nih.gov/assembly/GCA_900240005.1
- Mackiewicz, P. (2004). Where does bacterial replication start? Rules for predicting the oriC region. Nucleic Acids Research, 32(13), 3781–3791. doi:10.1093/nar/gkh699
- Сопроводительные материалы доступны по ссылке