Использование алгоритма поиска BLAST локально и через веб-интерфейс на примере контига из выбранной сборки эукариотического организма
Описание выбранного контига
В этом практикуме я продолжаю работать с сборкой генома для попугая какапо. Ee refSeq ID -- GCF_004027225.2. Был выбран скаффолд наименьшей длины, содержащий хотя бы 1 CDS. Он не помещен ни в одну из хромосом (unplaced scaffold). Его параметры:- На этом участке имеется 3 гена, среди которых 2 белок-кодирующих, причем у одного из белков 3 изоформы
- Идентификатор контига NW_022651074.1
- Длина: 21 488 b.p.
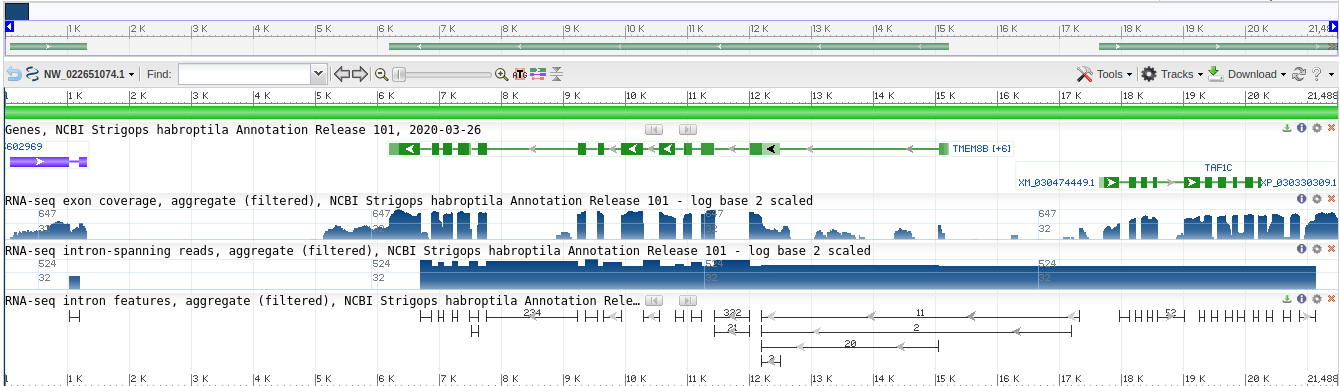
Скачать файл в fasta-формате можно по ссылке.
Поиск в BLAST
Далее проводился поиск последовательностей по выбранному контигу с использованием сервиса BLAST и алгоритмами blastn, megablast, blastx, tblastx и исключением некоторых таксонов. Параметры поиска и находки приведены в таблице 1.
Заметим, что попугаи какапо эволюционно обособлены от других видов птиц, и обычно сближаются с другими новозеландскими попугаями (род Nestor) в семейство Strigopidae [1][2]. Примечание: чаще всего количество находок не изменяется, т.к. у автора есть опасение, что как минимум один из белков, кодирующихся геном из данного скэффолда -- фактор, ассоциированный с TATA-box-связывающим белком -- присутствует у большого числа организмов в выбранных таксонах, что потребовало бы увеличения размеров выборки до допустимого максимума.
| Алгоритм поиска | Выбор таксонов | Длина слова | Количество находок | Таксономия находок | Состав находок | Пример использования алгоритма | |
| blastn | Исключен сам род Strigops, поиск среди птиц | 11 | 500/500 | Различные группы птиц: представители попугаеобразных (за исключением Cacatuidae), дневных хищных птиц, воробьинообразных, гусеообразных и др., а также бескилевых. | Найдены гомологи обоих белок-кодирующих генов, однако не найден ген, кодирующий ncRNA. Помимо этого 4 выранивания создано с целыми хромосомами (в данном случае это одна из половых хромосом -- Z). Это интересное наблюдение, т.к. рассматриваемый контиг не отнесен ни к одной хромосоме в рассматриваемой сборке. Находки с самым небольшим весом по размерам и местоположению совпадают с описанными в случае применения megablast для попугаеобразных без двух родов. | Поиск среди менее схожих (относительно megablast) последовательностей. Позволяет сравнивать геномы не-близкородственных видов, в том числе определять принадлежность генома или его фрагмента к конкретному организму. Также можно искать гомологи среди генов, не кодирующих белки. | |
| megablast (2 варианта - второй в скобках) | среди попугаев за исключением родов Strigops и Nestor (среди рода Nestor) | 16 (28) | 100/100 (7/100) | Psittacidae -- в результаты не включены два других семейства попугаеобразных (попугаи указанного рода) | У всех птиц найдены два участка в районе 8000 и 18 000 нуклеотидов, длиной около 200-300 b.p. Они принадлежат различным генам, в основном бета-фибриногену. При этом в оригинальном геноме такого гена нет, и найденные участки не соотносятся с границами генов в контиге. Возможно, это результат относительной эволюционной близости данных попугаев. (Хоть и фрагментарно, но найдены оба белок-кодирующих гена в контиге). Как итог, можно увидеть, насколько различается поиск среди более и менее близких видов. | Быстрый поиск среди очень похожих последовательностей -- подходит для особей того же или близкого вида. Может пригодиться для определения местоположения гена изучаемой последовательности гена | |
| blastx | В данном случае blastx не требуется использовать, т.к. гены уже известны, а для поиска гомологов предпочтительнее использовать blastp. И все же попробуем провести поиск с целью нахождения гомологов у других организмов -- были выбраны млекопитающие. | 3 | 100/100 | Разнообразные млекпитающие | Найден один из двух белков, кодируемых генами исследуемого контига. Это трансмембранный белок 8B. Заметим, что второй белок -- фактор, ассоциированный с TATA-box-связывающим белком -- найден не был, хотя поиск с использованием белка какапо показал наличие гомологов у млекопитающих | Аннотация генов и определение CDS | |
| tblastx | Попугаеобразные с исключением самих какапо | 3 | 100/100 | На этот раз найдены последовательности представителей всех трех семейств попугаеобразных | Повторяются результаты других поисков: найдены как гены из контига какапо, так и несвязанные с ними фрагменты -- участок гена бета-фибриногена и др. Мы предполагаем, что эти находки обусловлены эволюционной близостью попугаеобразных | Предсказание генов |
Поиск гомологов рРНК в геноме эукариот на основе гена E.coli
С помощью команды sudo apt install ncbi-blast+ установлена локальная версия BLAST. Создана локальная база данных на основе генома организма командой makeblastdb -in ../GCF_004027225.2_bStrHab1.2.pri_genomic.fna -dbtype nucl.
Командами
blastn -task blastn -evalue 0.05 -query RNA1.fa -db ../GCF_004027225.2_bStrHab1.2.pri_genomic.fna -out blast_rrna1 -outfmt 7
blastn -task blastn -evalue 0.05 -query RNA2.fa -db ../GCF_004027225.2_bStrHab1.2.pri_genomic.fna -out blast_rrna2 -outfmt 7
16S РНК входит в состав малой субъедиинцы рибосомы, а 23S -- большой.
Для 16S RNA найден 1 гомолог (с небольшим score и большим e-value -- 0.019), для 23S -- 5 (среди них 4 имеют e-value порядка как минимум 10^(-4); у лучшей находки e-value 2e-17, но небольшой score. Она находится внутри гена с CDS, продукт - неохарактеризованный белок XP_030347041.1). При этом в таблице особенностей не указано ни одной рРНК, кроме 5S или 5.8S. Поэтому сделать вывод о случайности находок трудно -- как минимум, мы имеем дело с неполной аннотацией генома.
Имеющиеся находки, если бы они были релевантными, соответствовали бы эукариотическим гомологам 16S и 23S rRNA -- это 18S и 28S rRNA. Эукариоты имеют большое количество копий этих РНК. Также могли быть найдены митохондриальные гомологи, но в данной сборке митохондриальный геном отсутствует.
- Wright TF, Schirtzinger EE, Matsumoto T, Eberhard JR, Graves GR, Sanchez JJ, Capelli S, Müller H, Scharpegge J, Chambers GK, Fleischer RC. A multilocus molecular phylogeny of the parrots (Psittaciformes): support for a Gondwanan origin during the cretaceous. Mol Biol Evol. 2008 Oct;25(10):2141-56. doi: 10.1093/molbev/msn160. Epub 2008 Jul 24. PMID: 18653733; PMCID: PMC2727385.
- https://www.bird-phylogeny.de/superorders/australaves/psittacifomes/
I don't know how to make footer properly. You may as well pretend you haven't seen this phrase!