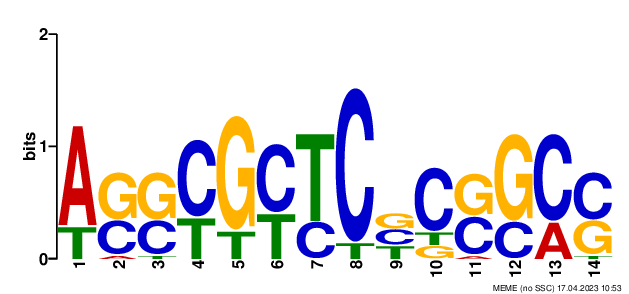Выбор входных последовательностей для MEME
В качестве входных последовательностей рассматривались опероны бактерии Deinococcus radiodurans (известна устойчивостью к радиоактивному излучению), сборка GCF_020546685.1 (референсный геном). Для дальнейшей работы использовалась хромосома 1. С помощью сервиса Operon Mapper были найдены координаты оперонов. Промотором считалалась область длиной 100 нуклеотидов перед опероном. Для составления тренировочной выборки выбирались гены домашнего хозяйства по следующим ключевым словам в аннотации: polymerase, gyrase, ligase, ATP synthase (бактерия - облигатный аэроб). Тестовая выборка составлялась из всех промоторов, отрицательный контроль - из случайно взятых последовательностей генома D.radiodurans аналогичной длины. Файлы с выдачей: train.fasta (33 последовательности), test.fasta (2640 последовательностей), control.fasta (33 последовательности).
Поиск мотивов с помощью MEME
Мотивы были найдены с помощью онлайн-сервиса MEME-suit. Попробовав разные минимальные числа встреч мотива в последовательностях, я остановилась на 15, так как меньшие значения не показывали новых мотивов. Мною было решено выбрать 2 мотив, так как он чаще обнаруживался в примерно одинаковых позициях у нескольких последовательностей и к тому же длиннее. Интересно, что при изменение параметра "How many sites must each motif have?" не помогло найти мотивы с хорошим e-value. Выдачу можно скачать: MEME.html.
| Параметр | Значение |
| Select the site distribution | 0 или 1 (zoops) |
| Select the number of motifs | 3 |
| Minimum and maximum width | 6 and 50 |
| Can motif sites be on both strands? (DNA/RNA only) | search given strand only |
| How many sites must each motif have? | min: 15 max: 33 (число последовательностей) |
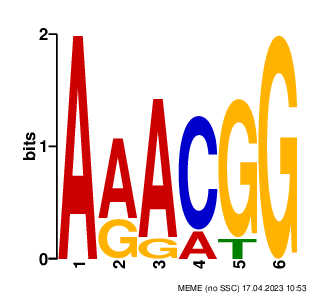
Проверка с помощью FIMO
Попробовав пороги e-value 10^(-4) (около 9000 находок), 10^(-3), 10^(-2) (около 250 находок), я выбрала 10^(-3) как самый оптимальный. Выдача FIMO для тестовых последовательностей может быть найдена здесь, для контрольной группы - тут (всего 33 последовательности). Точность, к сожалению, не очень высокая и в целом мотивы получились с очень большими значениями e-value. Думаю, это может быть связано с 1) неточностью предсказания оперонов 2) качеством самого генома 3) особенностями жизнедеятельности выбраннной бактерии - как было сказано в начале, она способна выдерживать большие дозы радиации. Предполагаемых способов защиты от нее несколько: геном D.radiodurans многокопийный (вероятно, для проведения гомологичной рекомбинации при образовании повреждений), также, у этой бактерии эффективные механизмы репарации повреждений. К тому же, в консенсусе мотива обильны гуанины и цитозины, и совпадения по случайным причинам могут быть связаны с повышенным GC-составом генома бактерии (в связи с экстремофильностью).
| Тренировочная | Тестовая | Отрицательный контроль | |
| "+"-сигнал | 20/33 | 1101 (1576 находок)/2640 | 23 (47 находок)/33 |
| "-"-сигнал | 13/33 | 1539 | 10/33 |
Благодарности
Выражаю благодарность Георгию Муравьеву за предоставленный скрипт. Он использовался с небольшими модификациями и поэтому не был приложен.
I don't know how to make footer properly. You may as well pretend you haven't seen this phrase!