Поиск консервативных мотивов в выравнивании гомологичных белков
Для работы были выбраны белки семейства HSP70 (AC PF00012). Они являются шаперонами, присутствующими повсеместно: как у бактерий, так и у эукариот (грибы, растения, многоклеточные животные и др). У этого семейства seed-выравнивание содержит 27 последовательностей. В выравнивании обнаружилось несколько участков с высокой консервативностью аминокислотных остатков. У белков семейства HSP70 есть нуклеотид-связывающий домен [1]. Интересно, что консервативность остатков выше в первой половине выравнивания, на которую приходится нуклеотид-связывающий домен. Могу предположить, что он должен содержать консервативные положительно заряженные аминокислоты - аргинин/лизин/гистидин в меньшей степени (исходя из структуры NTP), поэтому было решено выбрать данный участок с мотивом T[VIL]..[AILV]KR, т.к. в нем содержалось две сохраняющиеся позиции с положительно заряжеными аминокислотами. Границами мотива я решила сделать колонки с почти 100% идентичностью. По данному паттерну в исходном выравнивании находится 17 из 27 аминокислот. С помощью сервиса MyHits был выполнен поиск белков с таким мотивом в базе данных Swiss-Prot. В выдаче оказалось много белков (3395). Помимо гомологов HSP70, ниже перечислены некоторые интересные находки: D-аминоацил-тРНК-деацилаза (связывает тРНК, состояющую из нуклеотидов, следовательно, есть возможность неслучайной находки), белок LepA (согласно странице в Uniprot, производит гидролиз ГТФ => находка имеет отношение к нуклеотид-связывающему домену), метилтрансфераза RlmN (связывает тРНК), большая цепь карбамоилфосфатсинтазы (гидролизует ATP), потенциальная киназа At5g38990 (связывает АТФ) и др. Таким образом, находятся гомологи, белки, имеющие отношение к связыванию нуклеотидов, но есть и предположительно не связанные находки -- что объяснимо, так как мотив небольшой и в нем есть два неспецифичных аминокислотных остатка.
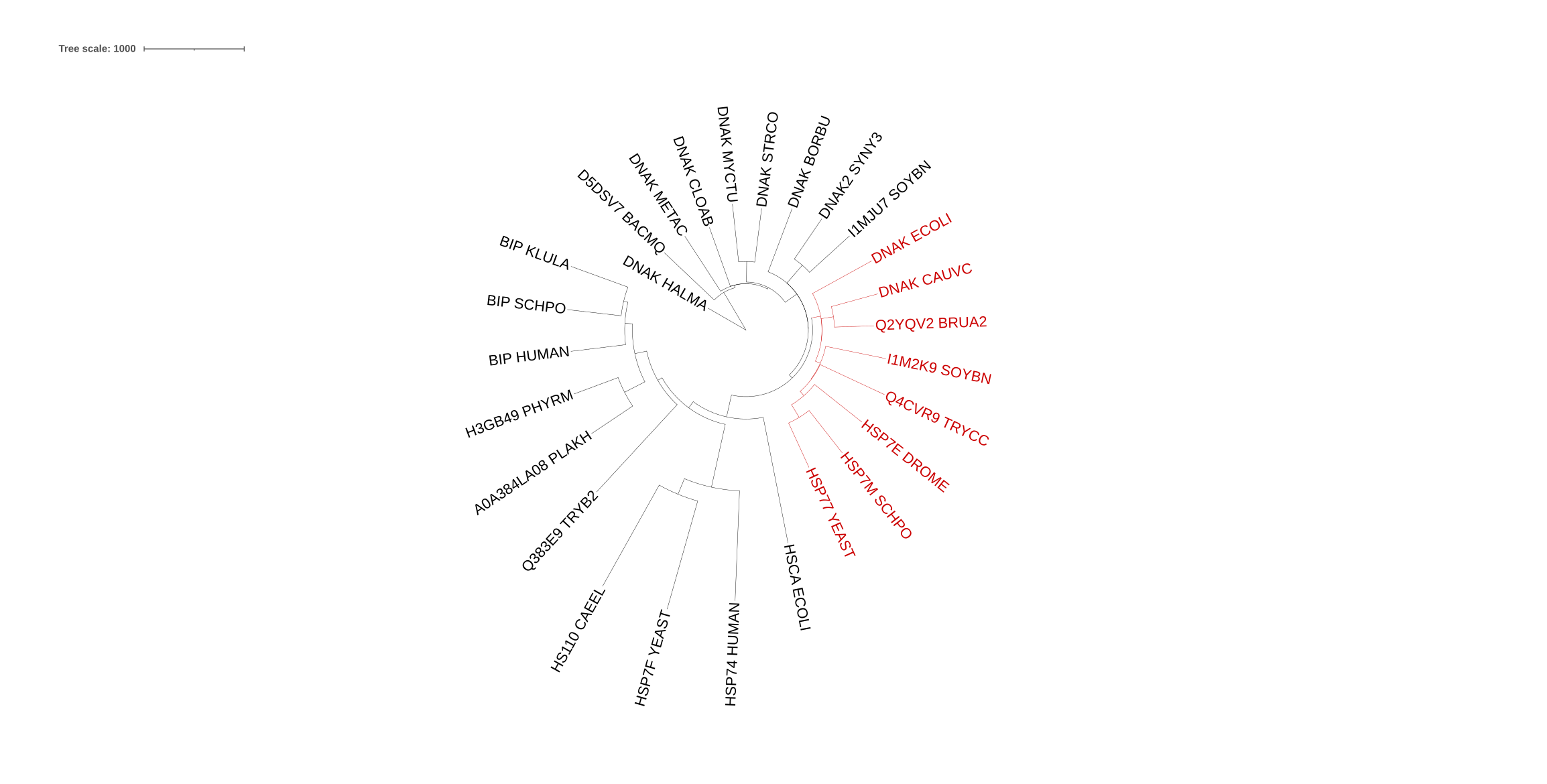
Специфичный мотив для клады одного дерева
Я выбрала кладу, отмеченную красным на данном дереве (построено методом Neighbour-Joining). Она содержит 8 последовательностей. Был обнаружен паттерн из четырех аминокислот: GDAW, появляющийся во всех последовательностях данной клады, но не находящийся в любых других группах. Несмотря на его небольшую длину, в нем содержится триптофан - редкая аминокислота, не находимая в этой позиции в белках из других клад. Скачать выравнивание этой клады можно тут.
Построение семейства гомологов на основании PSI-BLAST
Для работы был выбран белок P17265 (Ribosome hibernation promotion factor) - рибосомный фактор гибернации из агробактерии Rhizobium meliloti. С помощью алгоритма PSI-BLAST составлялось семейство гомологов данного белка. e-value для работы алгоритма было установлено ниже стандартного на порядок (0.005). После второй итерации число находок перестало увеличиваться. Заметим, что разница между находками до и после порога при этом составляет около 6 порядков, что свидетельствует о неплохой обособленности представленного семейства белков.
| Номер итерации | Число находок выше порога (0.005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1 | 17 | P0A147.1 | 7e-04 | O05886.4 | 0.046 |
| 2 | 28 | P33987.1 | 2e-08 | P9WMA8.1 | 0.009 |
| 3 | 28 | P33987.1 | 2e-08 | P9WMA8.1 | 0.009 |
Число TA-участков в геноме Deinococcus radiodurans
Для данного задания я выбрала бактерию, с которой работала в практикуме 7 -- Deinococcus radiodurans (GCF_020546685.1). Подсчитывать число нуклеотидов будем только в хромосоме 1. Длина хромосомы: 2,644,543. Подсчитав процент A и T нуклеотидов в геноме, вычислила ожидаемое число АТ-сайтов: 71636. Реальное же число таких сайтов: 35241, оказалось меньше ожидаемого более чем в 2 раза. Z-тест подтверждает статистическую значимость разницы: p-value << 0.
Источники:
- https://en.wikipedia.org/wiki/Hsp70
I don't know how to make footer properly. You may as well pretend you haven't seen this phrase!
