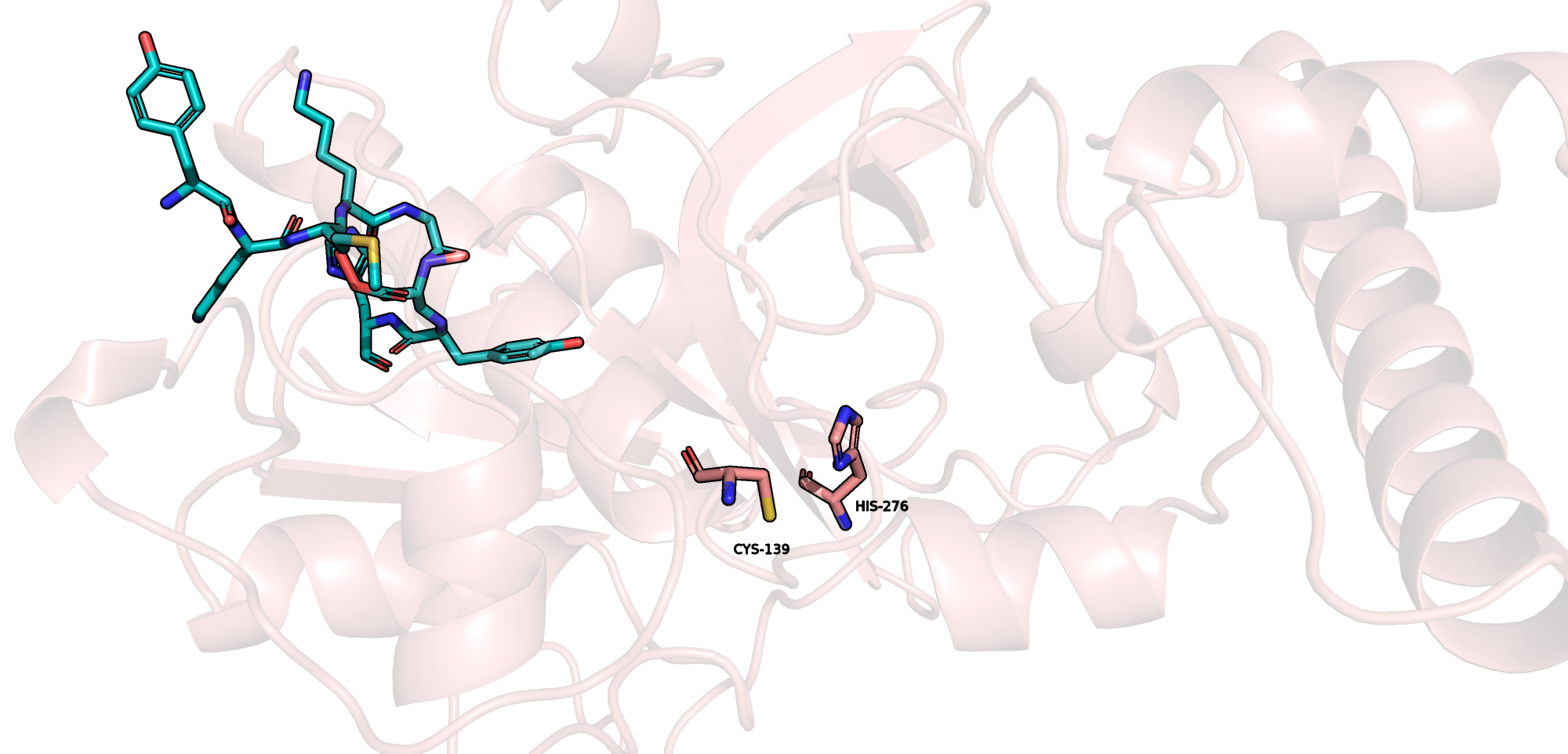Работа с AlphaFold2 (ColabFold)
A. Amyloids
Для данного задания я взяла пептидную послеовательность SVQIVYKPVDLSKVTSKCGSLGNIHHKPGGGQVEVKSEKLDFKDRVQSKIGSLDNITHVPGGGNKKIETHKLTFR длины 75, принадлежащую тау-белку, ассоциированному с микротрубочками (Microtubule-associated protein tau, MAPT). Накопление агрегатов тау-белка характерно для болезни Альцгеймера (т.н. "нейрофибриллярные клубочки", NFT). Интересно, что агрегаты существуют в разных формах: в виде парных спиральных филаментов (PHF), прямых филаментов (SF), лентовидных филаментов [1][2]. В базе данных Uniprot указано, что тау-белка имеется несколько изоформ с варьирующейся длиной (более 300 аминокислот), т.е. или при агрегации происходит частичный протеолиз белка (тау-белок может разрезаться, например, различными каспазами и это может быть связано с амилоидогенезом [1][2]), либо исследователи рассматривали только участвующую в образовании агрегатов часть белка.
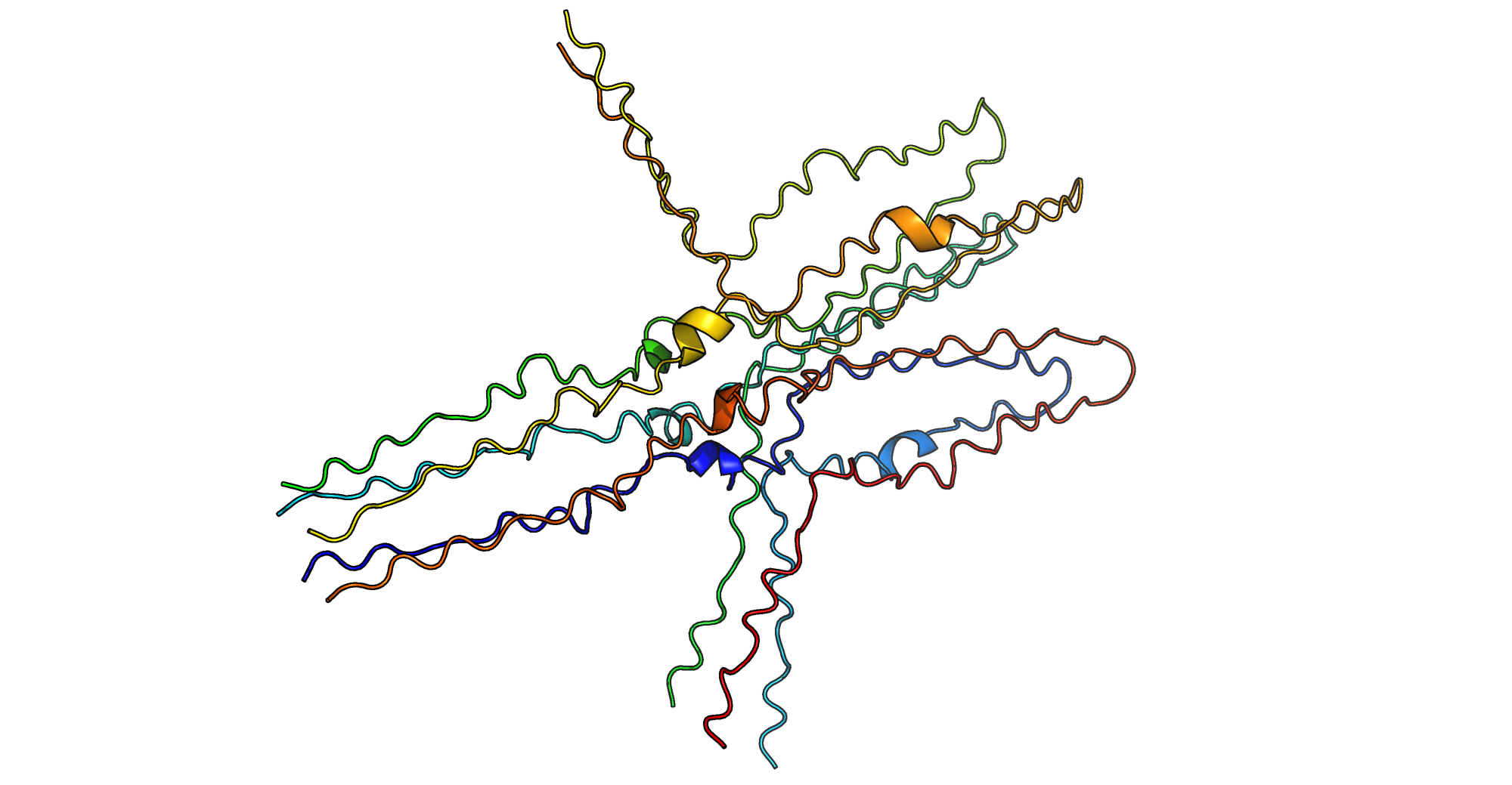
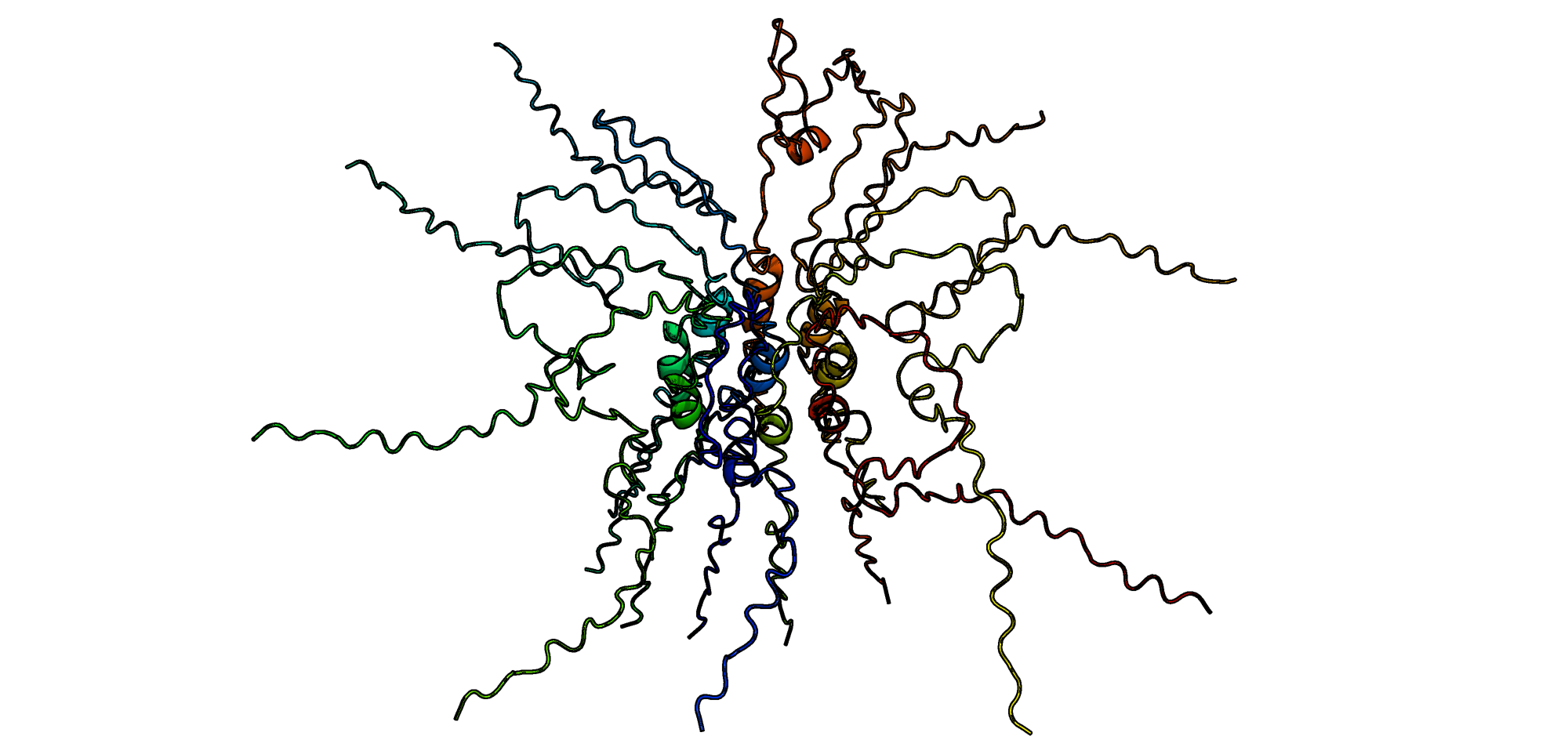
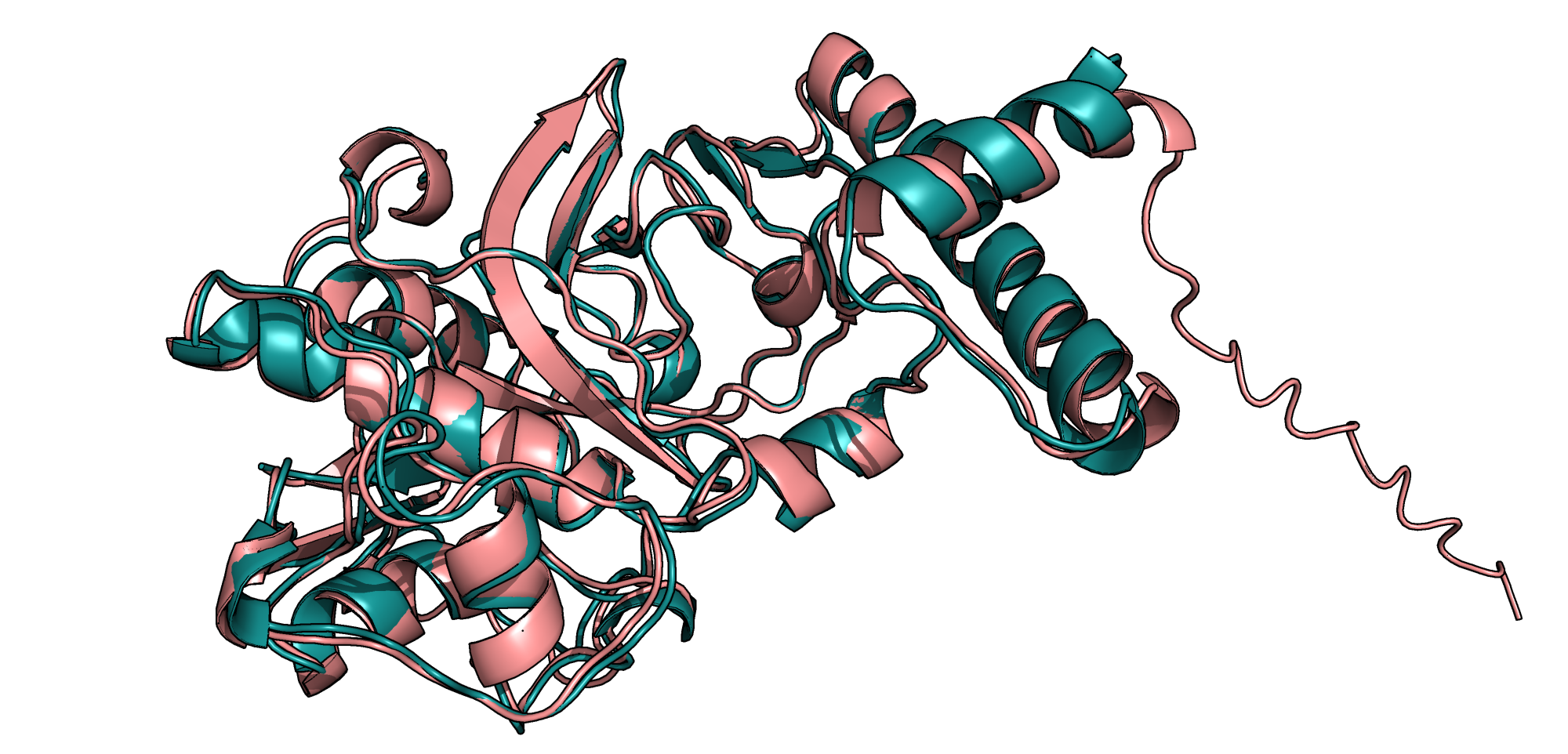
Рассмотрим резульаты предсказания структуры с помощью ColabFold для моно-, пента- и декамера (в каждом из случаев взяты структуры с наивысшим рангом). Стоит отметить, что участок тау-белка, с которым мне предстояло работать, содержит в себе тубулин-связывающий домен, в котором находятся ключевые для агрегации тау-белка участки, поэтому не было ясно, какой результат получится. Для сравнения я решила использовать структуру 2MZ7 для мономера фрагмента тау-белка, схожего с используемым в задании (в неагрегированной форме).
Картинки открываются в полном размере при нажатии.
Как оказалось, структура 2MZ7 совсем не похожа на сгенерированную. В полученном с помощью ColabFold фрагменте элементов с регулярной вторичной структурой совсем не наблюдается, в отличие от взятой из PDB. Однако в процессе поиска структур в PDB мною была обнаружена запись 8AZU, в которой содержится структура агрегата-гексамера, где аминокислотная последовательность мономера совпадала с предложенной. Если выровнять этот мономер с сгенерированной структурой, результат также окажется неудовлетворительным, но "на глаз" эта структура схожа с выдачей нейросети в большей степени, нежели 2MZ7.
Пента- и декамеры я также сравнила со структурами, найденными в PDB по аминокислотной последовательности и количеству белковых цепей в структуре (5, 10). Выровненные реальная и сгенерированная структуры не приведены, так как в рассматриваемом случае это довольно бесполезно. К сожалению, для пентамеров характерной структуры агрегатов не получилось (для всех 5 сгенерированных структур).
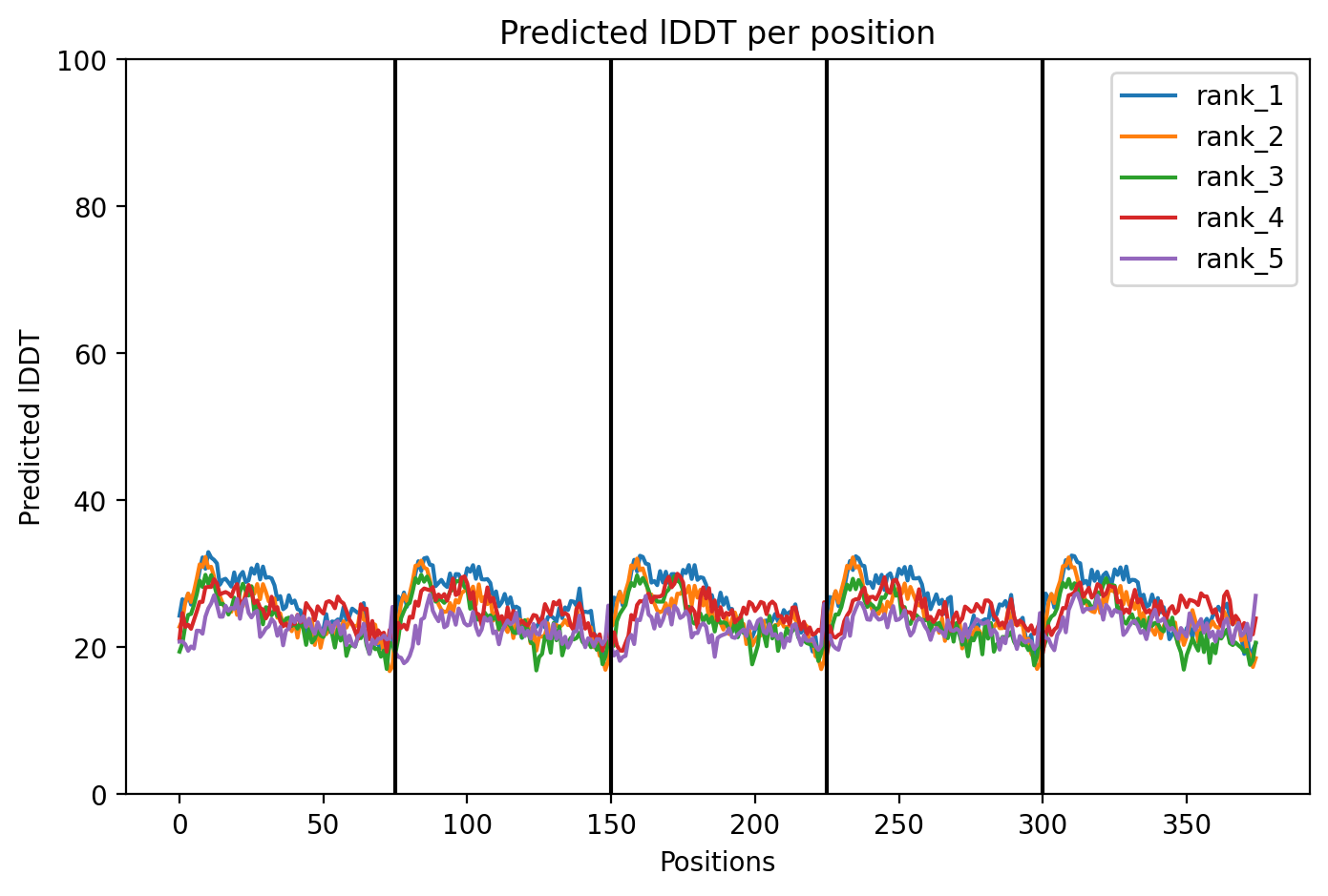
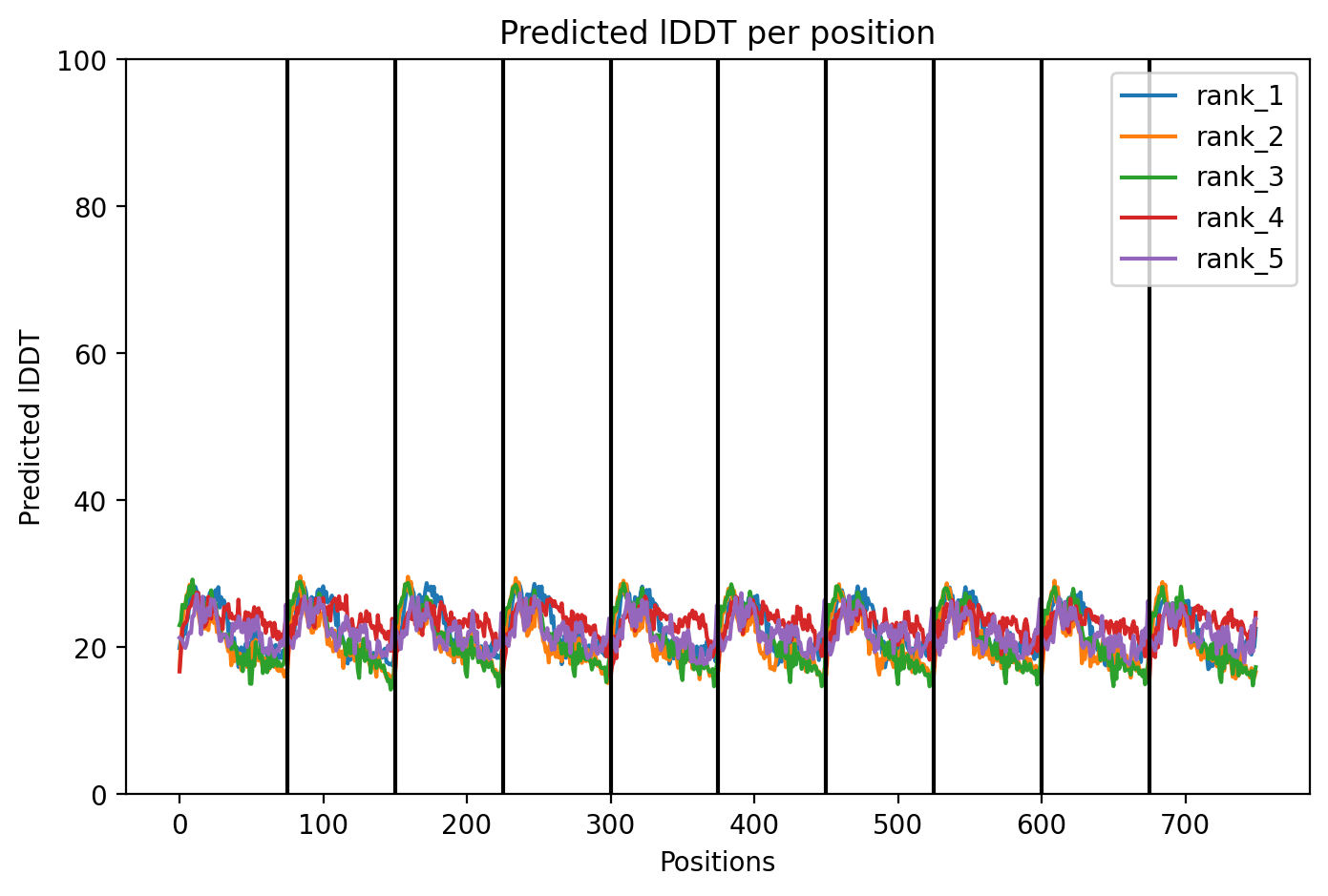
Подводя итоги, можно сказать, что ColabFold не справился с поставленной задачей. Скорее всего, дело в отсутствии такого же, как для глобулярных белков, разнообразия для подобных агрегированному тау-белку белков (как минимум фибриллярных), т.е. можно было бы ожидать для схожих белков. Ранее было упомянуто, что агрегаты тау-белка имеют несколько форм, поэтому при сравнении с экспериментальной структурой стоит это учитывать при генерации хоть сколько-нибудь реалистичной структуры. Стоит отметить, что полученный результат не был неожиданным: достаточно посмотреть на значения predicted IDDT :)
C. Proteases
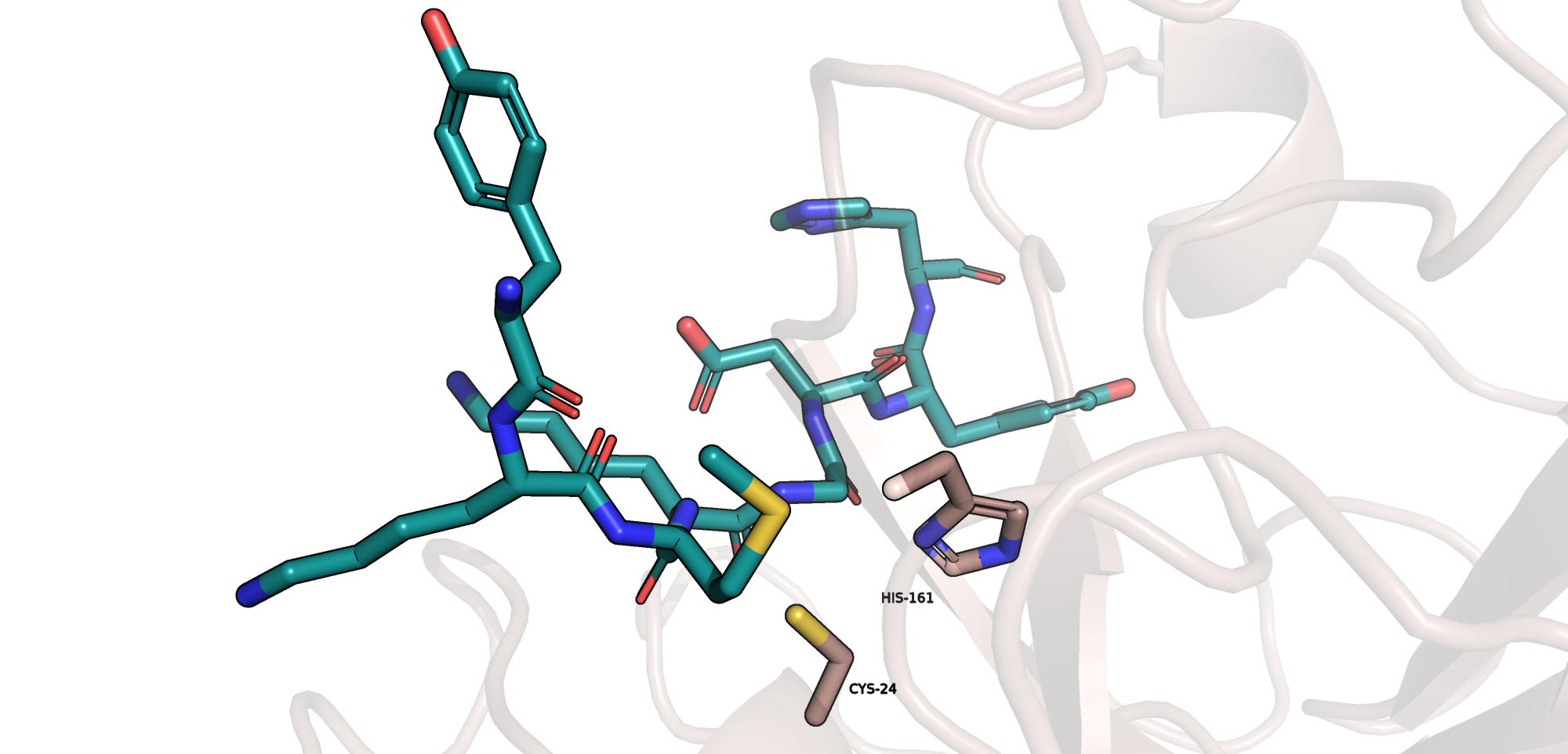
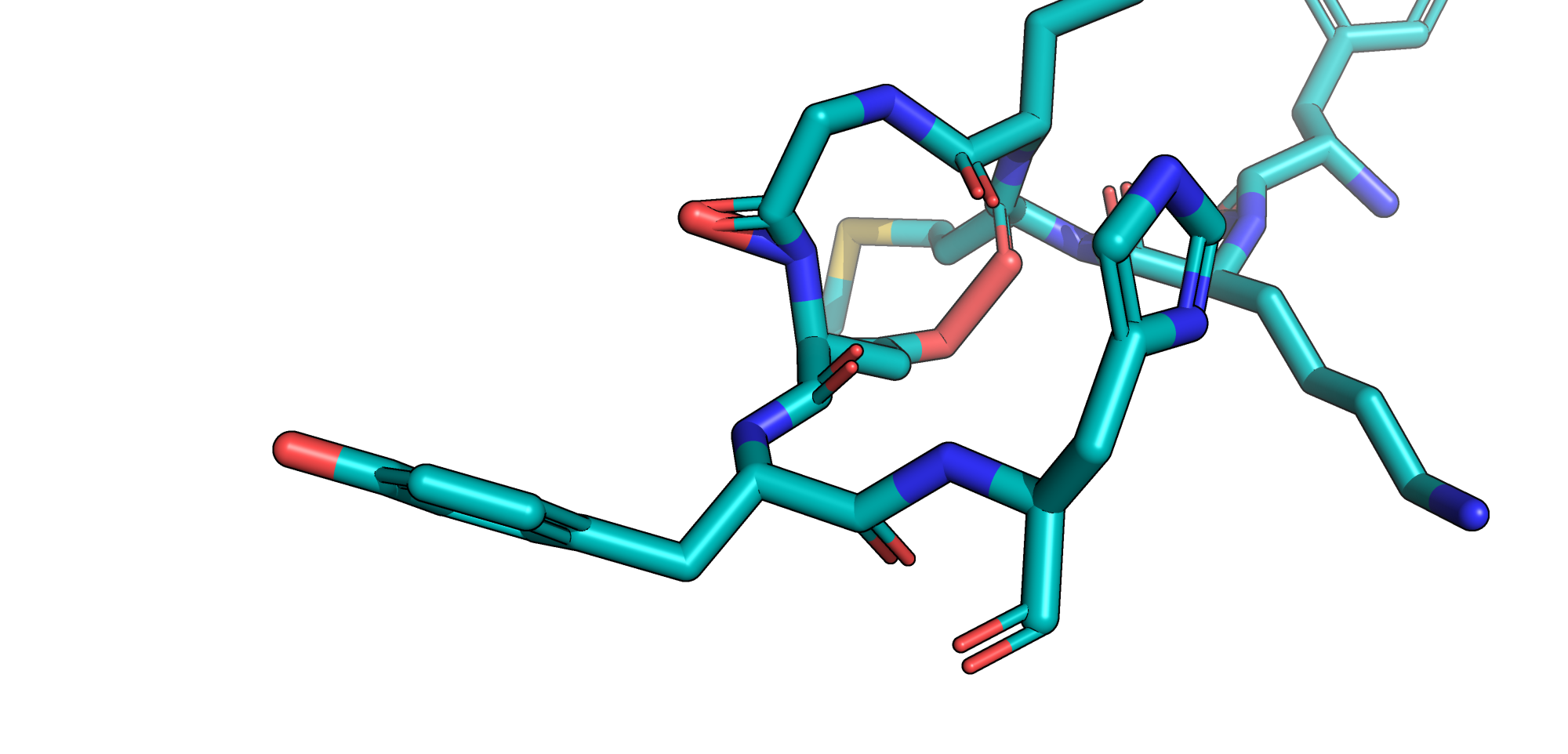
В данном задании я работала с комплексом "протеаза-субстрат". В качестве протеазы была взята цистеиновая протеаза катепсин K (Uniprot ID P43235), в качестве субстрата - пептид YKMKGDYH. В Uniprot отмечено, что представленная последовательность представляет собой прокатепсин (т.е. зимоген - неактивную форму фермента).
Опираясь на литературные данные и информацию в соответствующей записи в Uniprot, я отметила каталитическую диаду катепсина C. Субстрат в предсказанной структуре расположен совсем в другом месте, т.е. результат неудачный. Отметим также, что использованная экспериментальная структура не содержит ориентировочно 1-19 аминокислоты. В них входит сигнальный пептид, отрезаемый, видимо, раньше, чем пропептид.
Поскольку запуск ColabFold с проферментом был неудачен, было решено использовать активную форму фермента (согласно данным в Uniprot, 1-114 аминокислоты соответствуют сигнальному пептиду и пропептиду, т.е. вырезаются). В таком случае пептид, используемый в качестве субстрата, оказывается расположен близко к каталитической диаде.
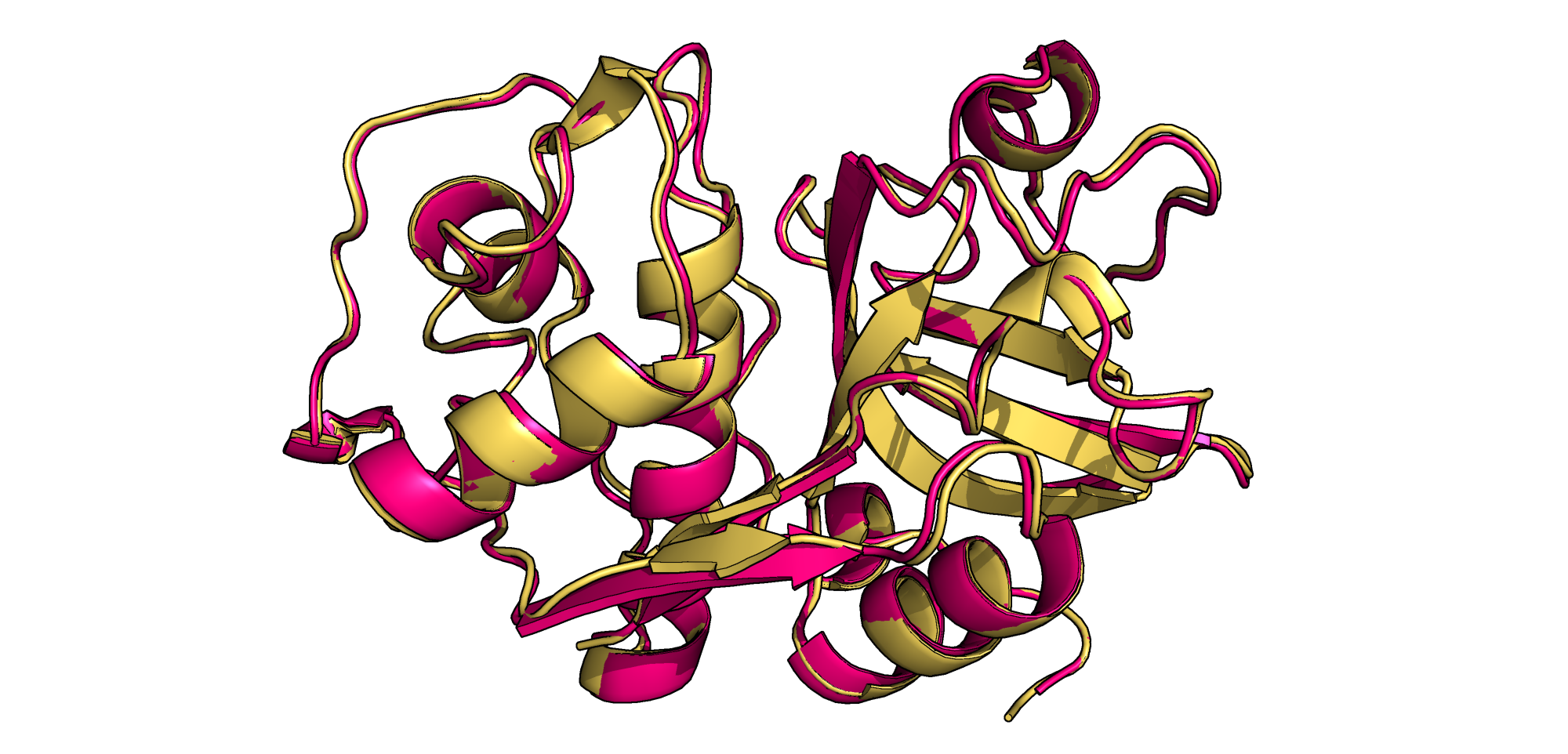
В этом задании, поскольку генерация структуры типичного глобулярного белка в целом успешна, можем посмотреть на сгенерированные структуры с рангом 1-5 и на расположение субстрата относительно активного центра в них. Видим, что положение аминокислот в активном центре практически не меняется, чего нельзя сказать про расположение субстрата. Сильные отличия заметны для структуры с рангом 5: его остов отдалился от активного центра, да и сама структура выглядит неестественно.

По результатам этой генерации можно предположить, что для активной формы пептидазы AlphaFold2 может расположить субстрат в нужном месте, но нужно внимательно проверять полученную структуру. Что касается экспериментальных контролей, можно сравнивать полученную структуру с реальной, если такая имеется, причем желательно использовать субстрат большей длины (дабы не получить то, что показано ниже).
Чуть не упущен забавный, но ожидаемый момент: структура субстрата в комплексе с зимогеном вызывает очень много вопросов, в то время как субстрат в комплексе с активным ферментом при беглом взгляде не вызывает особых проблем.
PyMOL-сессию можно скачать по ссылке.
Литература
- Nizynski B, Dzwolak W, Nieznanski K. Amyloidogenesis of Tau protein. Protein Sci. 2017 Nov;26(11):2126-2150. doi: 10.1002/pro.3275. Epub 2017 Sep 13. PMID: 28833749; PMCID: PMC5654847.
- Šimić G, Babić Leko M, Wray S, Harrington C, Delalle I, Jovanov-Milošević N, Bažadona D, Buée L, de Silva R, Di Giovanni G, Wischik C, Hof PR. Tau Protein Hyperphosphorylation and Aggregation in Alzheimer's Disease and Other Tauopathies, and Possible Neuroprotective Strategies. Biomolecules. 2016 Jan 6;6(1):6. doi: 10.3390/biom6010006. PMID: 26751493; PMCID: PMC4808800.
I don't know how to make footer properly. You may as well pretend you haven't seen this phrase!