Отчет за практикум 8
Задание 1. Выбор сборки генома эукариотического организма
Для выполнения задания я выбрала контиг NW_024570312, его длина - 8977 bp.
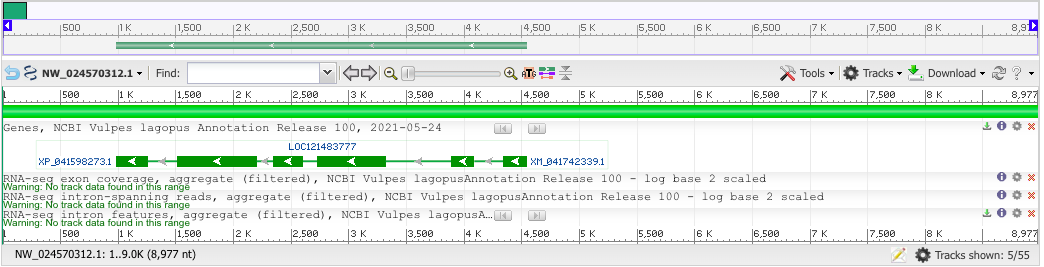
Зелеными прямоугольниками обозначены экзоны, их 6 штук. Между ними - интроны.
Результаты поиска BLAST:
Изначально все алгоритмы запускались с параметрами по умолчанию и с исключением таксона Chordata.
blastn: Очень много находок, достигнут максимальный размер выдачи - 5000 последовательностей. После уменьшения параметра Expect threshold до 0.001 и увеличения длины слова до 15 количество находок изменилось - 3688 последовательностей.
Blastn используют для поиска некодирующих гомологичных последовательностей. Он принимает на вход нуклеотидную последовательность и сравнивает ее с нуклетидной базой данных.
megablast: Обнаружено 17 находок, 15 из которых бактерии и 2 - вирусы.
Принцип работы megablast аналогичен blastn, однако алгоритм находит только сильно схожие последовательности, поэтому его выдача значительно меньше и работает он быстрее.
blastx: Достигнут максимальный размер выдачи (5000), причем у первых 100 находок E-value равен нулю. Я попробовала исключить другие таксоны повыше, но результат был тем же даже после исключения из поиска Эукариот.
Blastx принимает на вход нуклеотидную последовательность, транслирует ее в аминокислотную и затем сравнивает ее с белковой базой данных. С помощью данного алгоритма можно узнать, например, какие мутации могут быть в гене.
tblastx: Алгоритм не нашел ничего даже после того, как я убрала какие-либо ограничения по таксонам.
Tblastx так же как blastx принимает на вход нуклеотидную последовательность и переводит ее в аминокислотную, но сравнивает ее с транслированной базой данных секвенированных нуклеиновых кислот.
Задание 2.
Я установила BLAST+ на свой компьютер и проиндексировала последовательности генома Vulpes lagopus с помощью следующей команды:
makeblastdb -in Documents/vulpes_genomic.fna -dbtype nucl
Было необходимо провести поиск для некодирущих (рРНК) последовательностей в нуклеотидной базе данных, поэтому был выбран алгоритм blastn:
blastn -task blastn -query Documents/16S_rRNA_ecoli.fna -db Documents/vulpes_genomic.fna -out 16S_rRNA_ecoli.out
blastn -task blastn -query Documents/23S_rRNA_ecoli.fna -db Documents/vulpes_genomic.fna -out 23S_rRNA_ecoli.out
Полученные файлы: 16S_rRNA_ecoli.out; 23S_rRNA_ecoli.out
Среди найденных для 16S рРНК гомологов последние 5 явно ими не являются из-за большого e-value (5.8). На участках первых двух находок с очень высоким Score (NC_054830.1(131464660-131463120) и NW_024571137.1(1-759)) не аннотировано никаких генов. Аннотацию имеет только одна из находок - NC_054827.1(864115-864073). Этот участок кодирует ген, с которого экспрессируется 18S рРНК (и она действительно является гомологом 16S рРНК)
Из найденных гомологов для 23S рРНК можно исключить последние 2 из-за большого e-value. Интересно, что первые две находки совпадают с первыми двумя для 16S рРНК - NC_054830.1 и NW_024571137.1 (и никаких генов там тоже не аннотировано). На одном из найденных гомологичных участков (NC_026529.1(2338-2537)) аннотирован ген, с которого экспрессируется 18S рРНК.