Отчет за практикум 12
1. Базa данных OPM
В базе данных OPM я нашла трансмембранный белок с β-листами: Invasin. Он находится во внешней мембране бактерии Yersinia pseudotuberculosis.
| Идентификатор PDB | 4e1t |
| Идентификатор Uniprot | INVA_YERPS |
| Тип | Трансмембранный |
| Класс | Трансмембранный бета-бочонок |
| Суперсемейство | Автотранспортер |
| Семейство | Автотранспортер-3 |
| Вид | Yersinia pseudotuberculosis |
| Локализация | Внешняя мембрана грамотрицательных бактерий |
| Толщина гидрофобной части белка в мембране | 25.6 Å |
| Число субъединиц | 1 |
| Координаты трансмембранных участков | 1(150-157), 2(166-175), 3(183-191), 4(197-206), 5(211-220), 6(229-238), 7(241-249), 8(270-277), 9(287-292),10(313- 321),11(328- 333),12(343- 351) |
| Среднее количество остатков в одном β-тяже | 8-9 |

2. DeepTMHMM: Предсказание трансмембранных элементов по последовательности белка
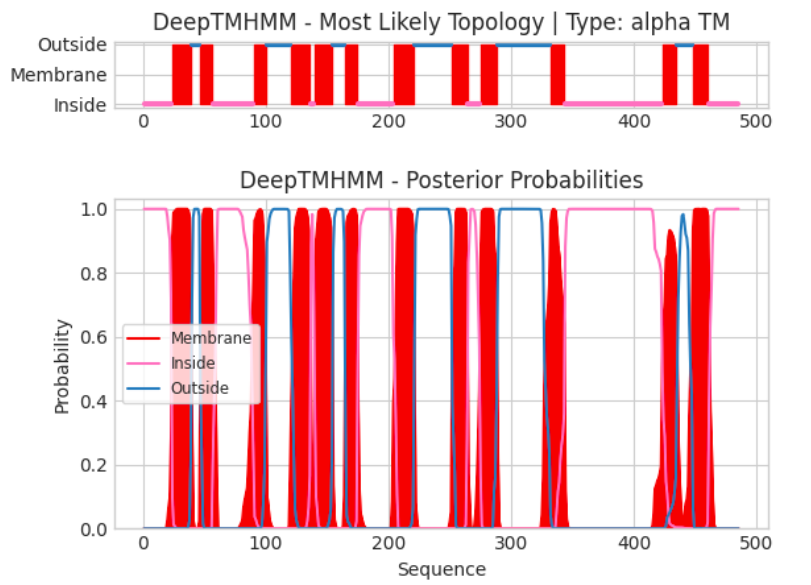
Мне был выдан белок с индентификатором ARCD_HALS3. Это аргинин/орнитиновый антипортер ArcD из археи Halobacterium salinarum. Я скачала его последовательность в формате FASTA и трехмерную структуру. Затем я запустила сервис DeepTMHMM.
Текстовая выдача для α-спирального белка: A_results.md. Графический результат можно увидеть на Рис 2.
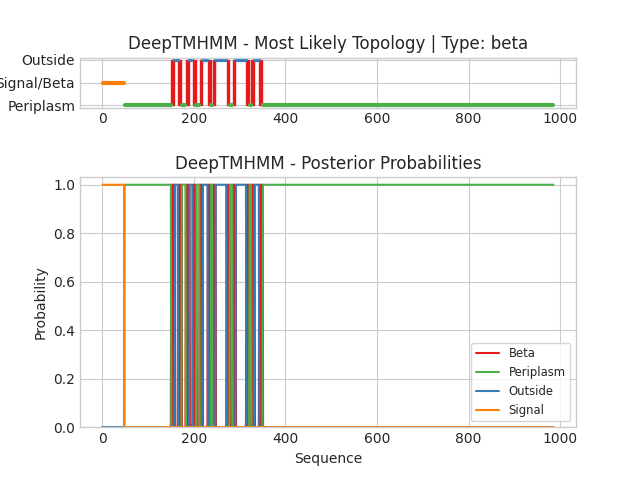
Также я запустила DeepTMHMM для найденного ранее мной β-листового белка INVA_YERPS. Его последовательность можно увидеть здесь. Текстовая выдача: B_results.md. Графический результат можно увидеть на Рис 3.
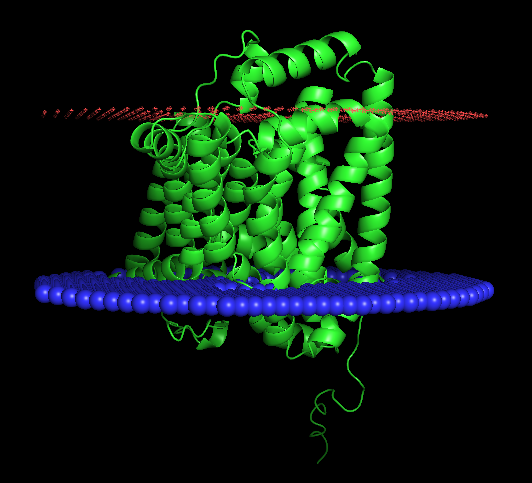
3. PPM: Предсказание положения выданного белка в мембране
Я запустила сервис PPM для выданного мне белка со следующими параметрами: Number of Membranes = 1, Type of membrane = Archaebacteria cell membrane (т.к. белок из археи), Allow curvature = no, Topology (N-ter) = in (из результата работы DeepTMHMM).
Результат:
| Толщина гидрофобной части | 28.8 ± 1.0 Å |
| Энергия Гиббса переноса | -82.6 kcal/mol |
| Угол изгиба | 20 ± 0° |
| Координаты трансмембранных участков | 1( 20- 41), 2( 45- 64), 3( 74- 103), 4( 117- 137), 5( 138- 154), 6( 161- 179), 7( 203- 224), 8( 252- 267), 9( 271- 290),10( 323- 344),11( 412- 426),12( 444- 464) |
| Среднее количество остатков в одной a-спирали | 20 |
4. Сравнение алгоритмов предсказания трансмембранных участков
Результаты работы DeepTMHMM для белка ARCD_HALS3 схожи со структурной информацией из БД OPM и предсказанием сервера PPM. Верно предсказано 12 трансмембранных участков, концы белка локализованы во внутренней части. В AlphaFold большая часть спиралей имеет высокую достоверность предсказания, вероятно поэтому результаты работы программ совпали.
Для β-листового белка INVA_YERPS результат предсказания программой DeepTMHMM тоже совпал с данными из OPM, были предсказаны все 12 трансмембранных участков.