Отчет за практикум 4
1. Составление списка гомологичных белков, включающих паралоги
Я скачала протеомы, отобранных мной ранее бактерий, и объединила их в один файл. Затем проиндексировала их и с помощью программы blastp провела среди них поиск гомологов белка CLPX_ECOLI.
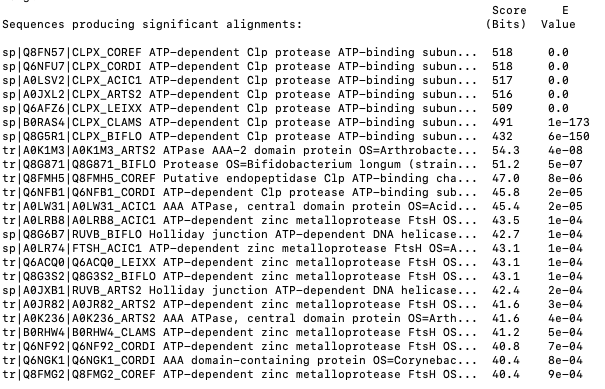
2. Реконструкция и визуализация
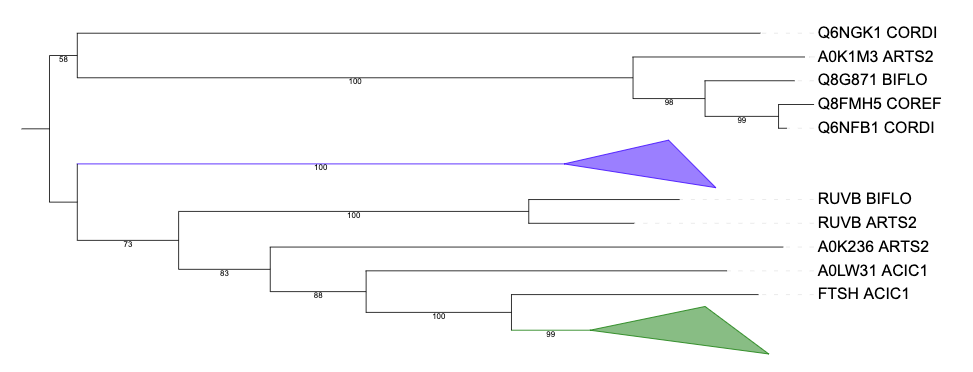
Затем с помощью сайта NGPhylogeny.fr я реконструировала дерево найденных гомологов программой FastME со следующими параметрами: "Gamma distributed rates across sites" — No, "Starting tree" — BIONJ, "No refinement", 100 бутстреп реплик.
Дерево в Newick формате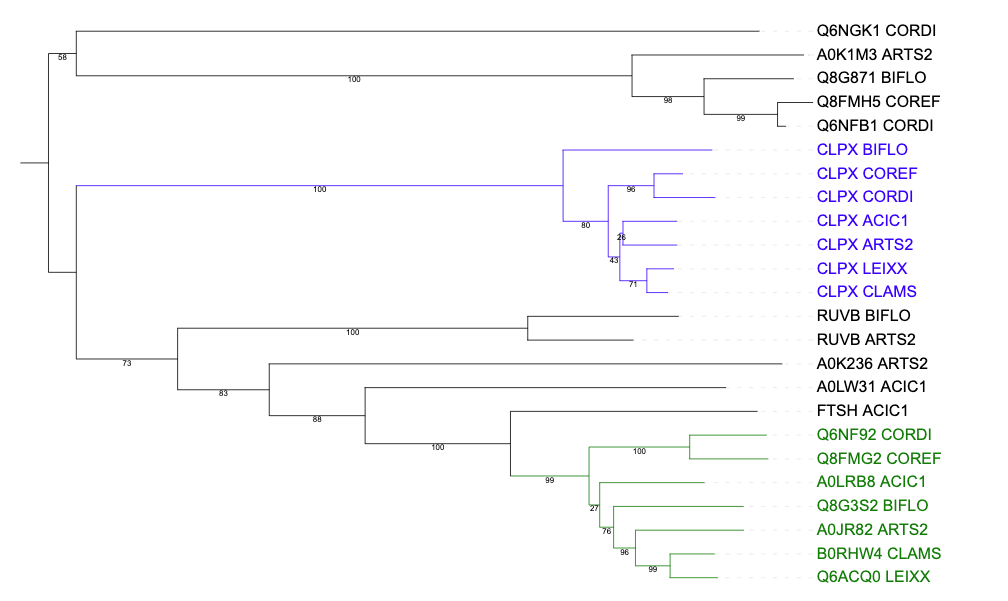
Считая, что дерево реконструировано верно, привожу примеры пар ортологов и паралогов.
Ортологи: CLPX_BIFLO и CLPX_COREF, RUVB_BIFLO и RUVB_ARTS2, Q6NF92_CORDI и Q8FMG2_COREF.
Паралоги: FTSH_ACIC1 и A0LRB8_ACIC1, Q6NGK1_CORDI и Q6NFB1_CORDI, Q8G871_BIFLO и CLPX_BIFLO.