3. Карта локального свойства двух белков с разной доменной архитектурой
1. Случайным образом я выбрала семейство доменов LIFR_N (PF18207)
2. Семейство доменов: LIFR_N (PF18207)
ID: PF18207
Это семейство доменов N-концевых рецепторов факторов ингибирования лейкимии.
Функция домена: это рецептор на поверхности клеток, выступающий посредником активности факторов ингибирования лейкимии и других Интерлейкинов-6 (ИЛ-6) через формирование сигнальных комплексов с гликопротеином 130(gp130)[1].
Число последовательностей (full) — 198. Число последовательностей в выравнивании seed (Alignment) — 33
Число доменных архитектур с этим доменом — 6, две из которых представлены 102 белками (структуры LIFR_N, LIFR_D2, fn3 ) и 87 (структуры LIFR_N, LIFR_D2) белками соответственно.
3d структура известна для 3х белков содержащих домен семейства: LIFR_HUMAN (человеческий рецептор факторов ингибирования лейкимии, цепь А), LIFR_MOUSE мышиный рецептор факторов ингибирования лейкимии, цепи А и С)
Домены из этого семейства встречаются только у 137 видов позвоночных.
HMM профиль выравнивания был создан в 2015 году, и содержит 79 позиций.

ссылка на скачивание проекта Jalview
1-я немногочисленная подгруппа обозначена лазурным цветом
Ее особенности:
в 12ом элементе выравнивания находится - Q,
13ом - N,
15ом - T,
19ом - H,
24ом - V,
27ом - T,
29ом - T,
39, 40 - DD,
45ом - Y,
47ом - F,
49ом - Y,
52ом - S,
61ом - S,
66ом - E,
67ом - K,
71ом - T,
72ом - F,
75ом - L,
76ом - I,
78ом - F,
79ом - D,
85ом - I,
87ом - T,
88ом - F,
89ом - N,
90ом - S.
Вторую группу (отмечена синим цветом) можно охарактеризовать отсутствием вышеперечисленных элементов (аминокислотных остатков в соответствующих позициях) и следующими “колонками” :
72 - I (присутствует в большинстве последовательностей. Cтоит учесть что последовательностей во второй группе значительно больше, поэтому полностью совпадающие колонки ак остатков тут встречаются реже)
73 - P (не полностью совпадающий участок)
84 - T (встречается примерно столько же раз, сколько и P)
100 - F
Uniprot выдал больше белков с доменом, чем указано в Pfam в выравнивании full (в full - 198, в uniprot - 452) т.к. выравнивание full основано на сравнении с "демонстрационными протеомами"(representive proteoms) для порога сходства в 15%, 35%, 55%, 75%.
Процент белков, чье существование надежно доказано в Swiss-prot составляет 100%(4 белка из 4х. По отношению ко всем 452 белкам это лишь 0,885%):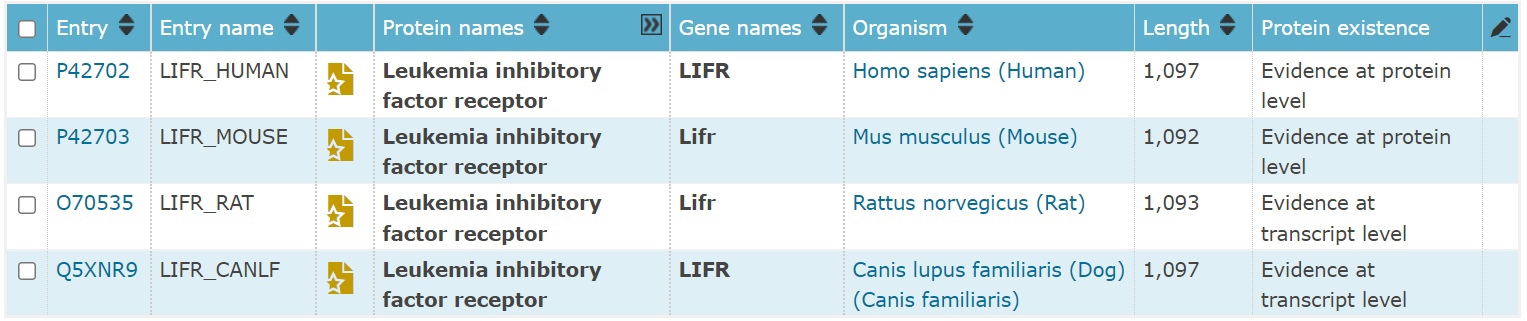
1 . Huyton T, Zhang JG, Luo CS, Lou MZ, Hilton DJ, Nicola NA, Garrett TP;, Proc Natl Acad Sci U S A. 2007;104:12737-12742.: An unusual cytokine:Ig-domain interaction revealed in the crystal structure of leukemia inhibitory factor (LIF) in complex with the LIF receptor.