Практикум 12.
Для выполнения заданий использовались: последовательность белка YYBK_BACSU (Неохарактеризованный белок YybK из организма Bacillus subtilis, пренадлежащий к суперсемейству атф-связывающих кассет) в формате FASTA YYBK_BACSU.fasta .
Предсказание трехмерной структуры, полученное с помощью AlphaFold AF-P37493-F1-model_v4.pdb .
Задание 1.
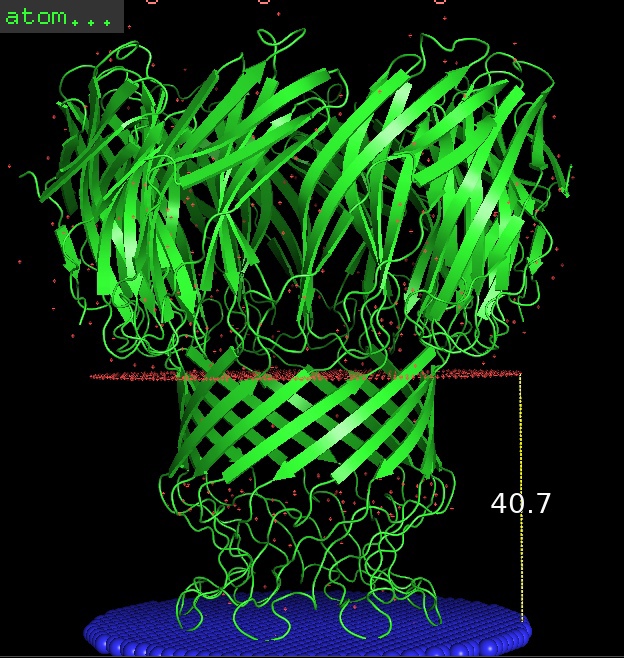
В базе данных OPM был найден белок являющийся основным порином в организме Mycobacterium smegmatis (микобактерия, штаммы которой используют как модельный организм). Белок образует заполненный водой канал, который способствует проникновению катионов, аминокислот, железа Fe3+ и менее эффективно фосфатов. Играет роль в транспорте бета-лактамазы и гидрофильных фторхинолоновых антибиотиков, таких как норфлоксацин, а также хлорамфеникола.
В трансмембранной части белка содержится β-бочонок из 16 β-листов.
Идентификатор PDB: 1uun
Идентификатор Uniprot: MSPA_MYCS2
Для этого белка была определена некоторые параметры:
| Толщина гидрофобной части белка в мембране | 40.7 Å |
|---|---|
| Координаты трансмембранных участков. | 1(74-83),2(111-119) 1(74-83),2(111-119) 1(74-83),2(111-119) 1(74-83),2(111-119) 1(74-83),2(111-119) 1(74-83),2(111-119) 1(74-83),2(111-119) 1(74-83),2(111-119) (белок симметричный => координаты одинаковы для всех субъединиц) |
| Среднее количество остатков в одном β-тяже белка. | 17(9+8) |
| Мембрана, в которую встроен белок. | Мембрана грамположительных бактерий |

Задание 2.
С помощью сервиса DeepTMHMM были предсказаны положение 2х белков в мембране: бета-листовогого (MSPA_MYCS2) и YYBK_BACSU - альфа-спирального.

Текстовая выдача программы: YYBK_BACSU.gff3

Текстовая выдача программы: MSPA_MYCS2.gff3
На рис.3 видно, что все 6 трансмембранных элементов белка YYBK_BACSU были предсказаны верно. С предсказанием траснмембранных элементов MSPA_MYCS2 DeepTMHMM не справился. Большинство бета-бочонковых трансмембранных белков, с которыми DeepTMHMM не справляется - не могут образовывать пору самостоятельно, и выполняют свою функцию только либо связываясь с другими белками, либо образуя мультимеры с дополнительными копиями тех же белков, например, путем тримеризации[1]. И, видимо, MSPA_MYCS2 можно отнести к "проблемным" белкам.
Задание 3.
Алгоритм PMM былы запущен со следующими параметрами:
Number of Membranes: 1
Type of membrane: Gram-positive bacteria inner membrane
Allow curvature: no
Topology (N-ter): in (согласно результату DeepTMHMM, см выше)

| Толщина гидрофобной части белка в мембране | 30.5 ± 0.9 Å |
|---|---|
| Координаты трансмембранных участков со вторичной структурой | 1( 3- 21), 2( 44- 65), 3( 88- 116), 4( 131- 157), 5( 158- 179), 6( 186- 193), 7( 217- 237) |
| Среднее количество остатков в одном трансмембранном сегменте | 20.14 |
| Угол изгиба | 2 ± 1° |
Задание 4.
Про MSPA_MYCS2 и DeepTMHMM см. задание 2.
Что касается белка YYBK_BACSU, то DeepTMHMM предсказывает 6 трансмембранных участков, а PPM - 7. Но в основе PPM лежит предсказанная AF структура. При ее расмотрении на странице Uniprot видно, что помимо 6 предсказанных спиралей со средней достоверностью, есть 7я, с низкой достоверностью (рис.6). Скорее всего ее PPM также предсказывает как трансмембранный участок. Таким образом можно сказать, что достоверность модели AF оказывает влияние на результат PPM .

Источники.
1. Bernhofer, M., Rost, B. TMbed: transmembrane proteins predicted through language model embeddings. BMC Bioinformatics 23, 326 (2022). https://doi.org/10.1186/s12859-022-04873-x