Основные параметры BPLASTp
BLASTp - это программа, позволяющая находить гомологичные участки белковых последовательностей. Разберемся, что же нужно знать, чтобы поиски проходили успешно.
Сначала блок "Enter Query Sequence". Здесь мы можем в поле ввода вбить аминокислотную последовательность белка (особенно актуально, если его нет в нужной нам базе данных), либо его AC, либо подгрузить файл с последовательностью. "Query subrange" позволяет задать диапазон аминокислот для поиска. Отсюда с помощью "Align two or more sequences" можно перескочить в сравнение двух и более белков.
Следующий блок "Choose Search Set" дает возможность указать базу данных для поиска "Database", организм "Organism" (используются таксономические идентифокаторы), последовательности которых нужно рассматривать, а можно и исключить последовательности этого организма с помощью галочки в поле "exclude". Есть здесь и отдельный пункт "Exclude", с помощью которого можно исключать группы по другим принципам.
Блок "Program Selection" предлагает нам выбрать алгоритм поиска.
Дальше - интереснее. С помощью "Algorithm parameters" настроим параметры поиска, т. е. выполним более тонкую отладку параметров. Здесь видим 3 блока.
Блок "General Parameters":
→ "Max target sequences" позволяет установить лимит на количество показываемых результатов;
→ "Short queries" может включать автоматическую настройку параметров под короткие последовательности;
→ "Expect threshold" принимает максимально допустимое E-value для находок;
→ "Word size" - длина затравки, по которой строится локальное выравнивание нашей последовательности и найденных;
→ "Max matches in a query range" ограничивает число совпадений на одном (рассматриваемом) участке последовательности.
Блок "Scoring Parameters":
→ "Matrix" - выбираем матрицу весов;
→ "Gap Costs" - цена гэпа (штрафы за гэпы);
→ "Compositional adjustments" - варианты корректировки матрицы для получение более точного E-value (борьба с участками малой сложности).
Блок "Filters and Masking"
→ "Filter" - можно замаскировать участки малой сложности;
→ "Mask" - первое поле: маскировка участков малой сложности на этапе поиска затравок; второе поле: маскировка строчных букв.
Результаты поиска можем открыть как в том же окне (по умолчанию), так и в новом, если поставим галочку в поле "Show results in a new window".
Задание 1. Поиск гомологов белка DFX_DESB2 в БД Swissprot
Проведен поиск белков, гомологичных десульфоферродоксину, в BLASTp. На основании найденного - составлена таблица о находках. Ее можно скачать здесь: табличка.
Построение выравнивания
скачать проект выравнивания
Для построения выравнивания было взято 10 белков, из них 2 гомологичны десульфоферродоксину [query cover > 80%], 2 - негомологичны совсем [query cover ~30-40%], а 6 - "средние".
Подтверждением гомологичности всех последовательностей считается участок выравнивания
→ начинающийся и заканчивающийся абсолютно (100%) консервативной позицией
→ длиной более 6 колонок
→ без колонок с гэпами
→ в котором высокая плотность консервативных позиций
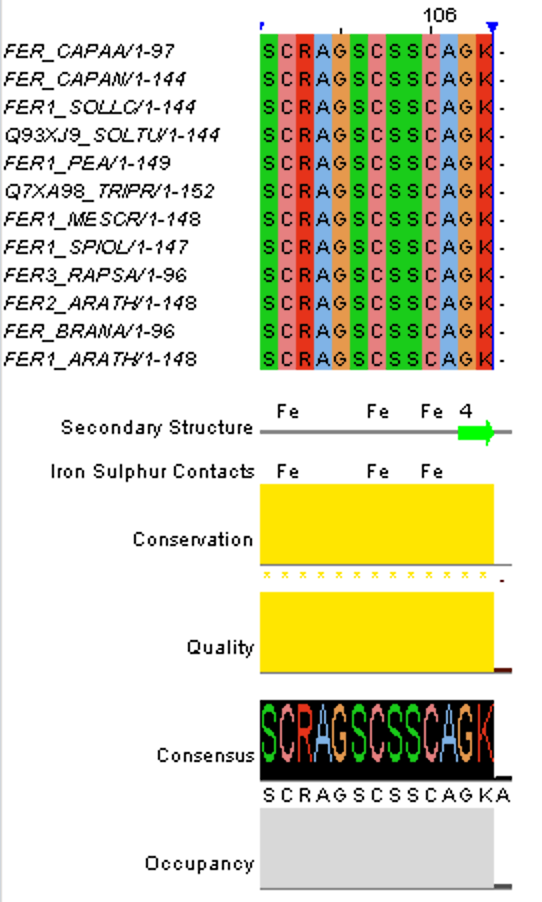
Казалось бы, белки были взяты разной степени "гомологичности", но, когда я построила выравнивание (см. выравнивание), то по написанным выше критериям все выбранные мной белки оказались гомологичны (см. Рис. 1).
Рис. 1. Гомологичность выбранных белков
Задание 2. Объяснение карты сходства двух белков
В BLASTp при выборе опции "Align two or more sequences" с помощью Dot Matrix (см. Рис. 2) построено выравнивание белков M2R9A4_CERS8 [по горизонтали, из первой группы] и D3AZP5_POLPP [по вертикали, из третьей группы]. Длины белков составляют соответсвенно 252 и 593 аминокислотных остатков. У _POLPP относительно _CERS8 наблюдается транслокация участка 110-117 на 580-593.
Рис. 2. Карта локального сходства белков
Задание 3. Игры с BLAST
Поиск по "случайной" последовательности в Swiss-Prot
Я позаимствовала последовательность у Иосифа Александровича Б.: "NOW IS WINDY AND THE WAVES ARE CRESTING OVER". БД - SwissProt, максимальное число находок - 1000, длина затравки - 3. О чудо, всего одна находка! Правда, E-value=3.0. Тем не менее, процент идентичности составляет 61,54%. Здесь можно посмотреть информацию о находках.
Поиск моего белка DFX_DESB2 при изменении wordsize
Wordsize = 2. Число находок: 12019Wordsize = 3. Число находок: 12019
Wordsize = 6. Число находок: 12016
Это в целом подтверждает ту закономерность, что чем меньше wordsize, тем больше находок, но у моего белка для wordsize 3 и 6 это значения не имеет.
Поиск схожих с OPSD_APIME белков, не встречающихся у членистоногих
OPSD_APIME - родопсин пчелы (воспринимающий свет с большой длиной волны).В пункте "Organism" с помощью опции "exclude" были исключены членистоногие (taxon ID: 6656), длина затравки - 3, БД - SwissProt, порог числа находок - 20000. Всего нашлось 16513 последовательностей, при этом минимальный E-value составил 0,90, что очень немало, особенно если сравнивать с поисками, проводимыми мной ранее.
