3. A,B,Z-формы ДНК
Цель работы - узнать основные характеристики и научиться различать A,B,Z-формы ДНК.
Задание 1. Команда fiber пакета 3DNA
Скачать структуру дуплекса ДНК а А-форме
Скачать структуру дуплекса ДНК а В-форме
Скачать структуру дуплекса ДНК а Z-форме
Использованные команды
fiber -h - для справки о том, что команда умеетfiber -a(b,z) -seq=ATGCATGCATGCATGCATGC gatc-a(b,z).pdb - строим дуплексы и записываем их в pdb файл в рабочей директории
Задание 2. Сравнение сгенерированной структуры Z-формы ДНК со структурой той же формы полученной экспериментальными данными
С помощью команды fiber получен pdb-файл с посделовательностью z-формы ДНК (состоящей из 10 повторов GC - по дефолту). Визуально определены большая и малая бороздки.
Дальше случилось непредвиденное. При визуализации в JMol оказалось, что z-форма "в чистом виде" состоит только из GC- пар. Оказалось, что устойчивая z-форма может образоваться при изменении условий в участках b-ДНК, богатых GC парами.
Выделена пара GC, С - красный, G - синий.
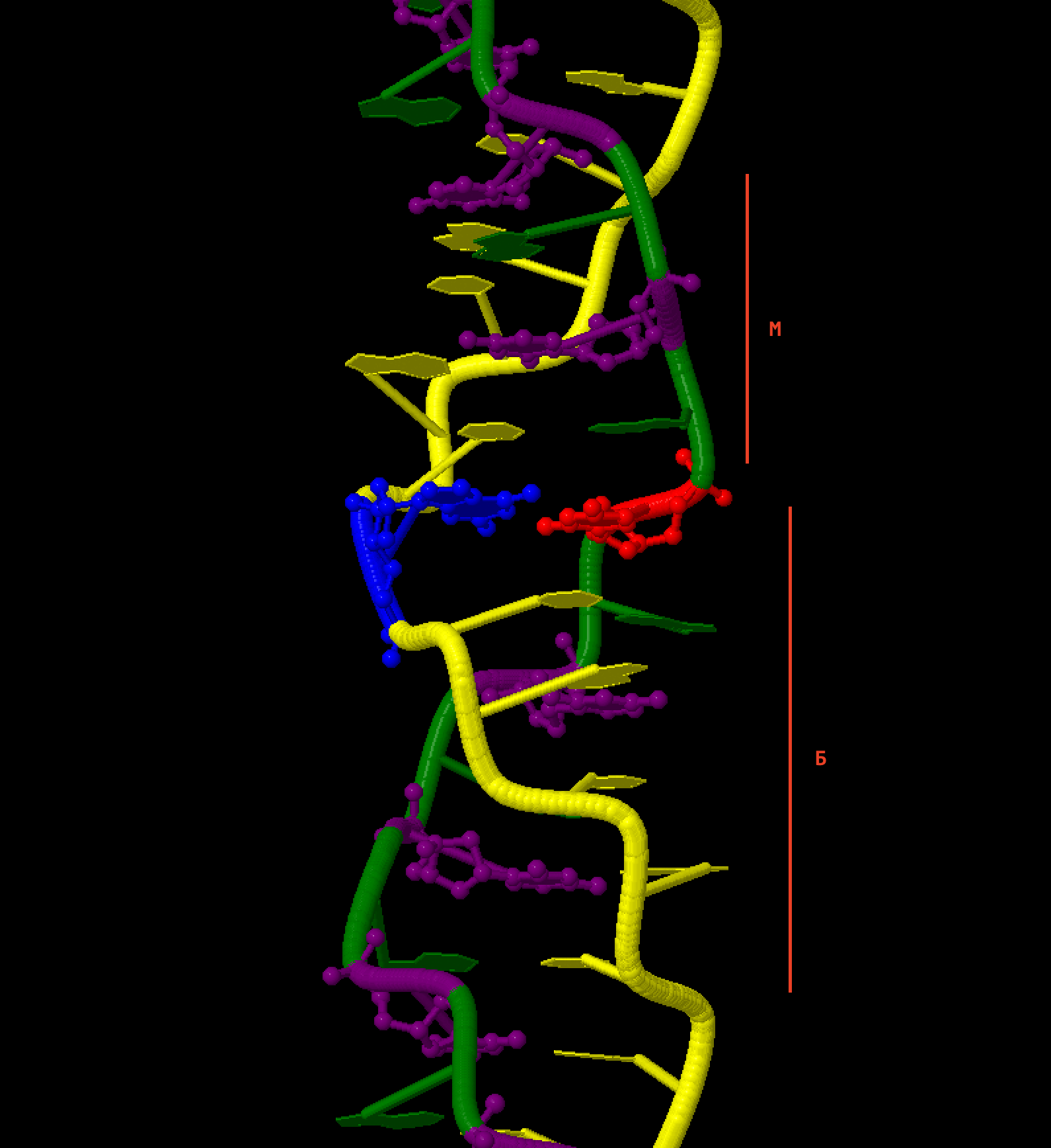
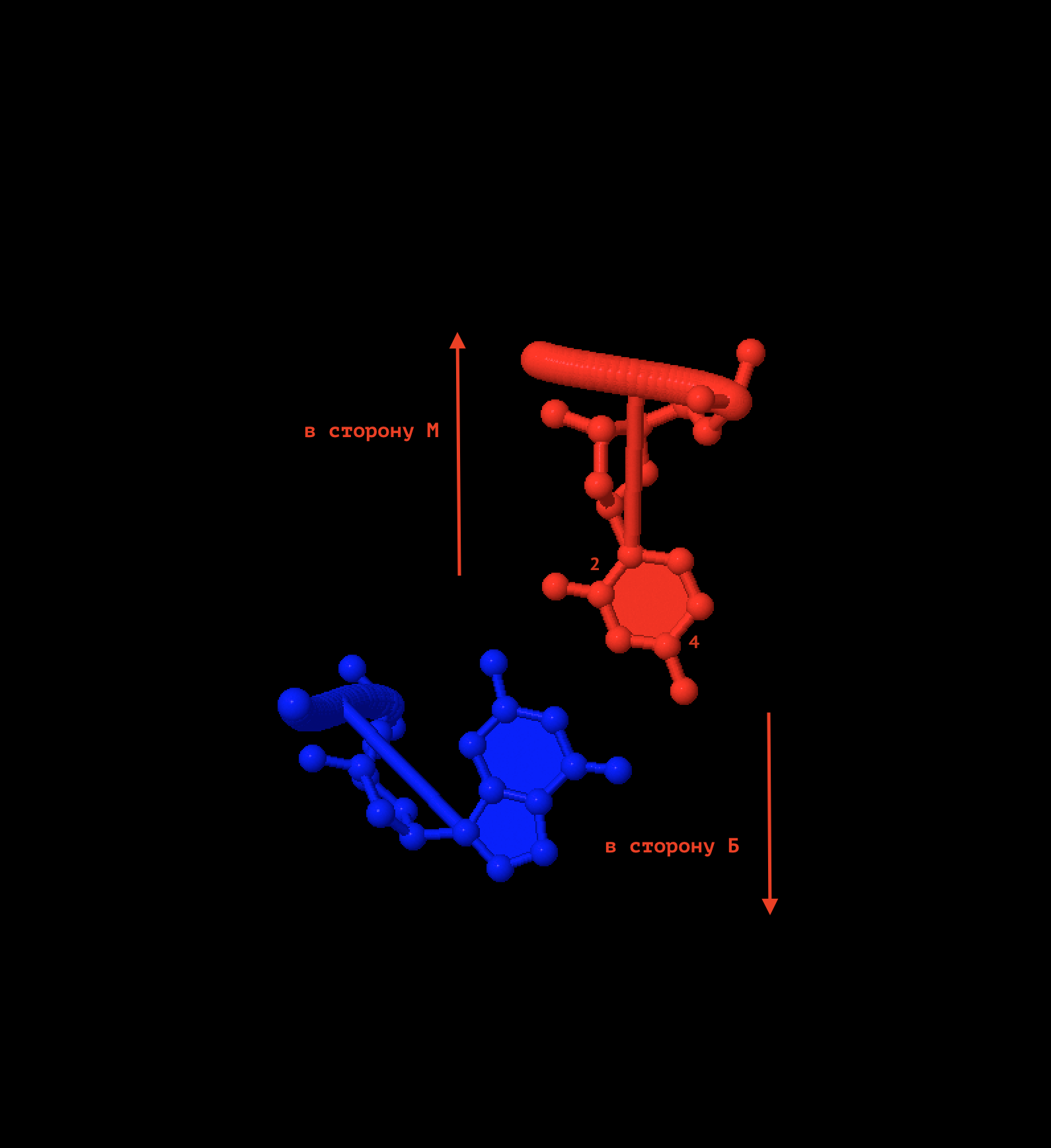
Так как GC-насыщенность z-формы была мной открыта уже в процессе работы, а определенное мне основание - тимин, я решила описывать далее цитозин (тоже пиримидин, по крайней мере).
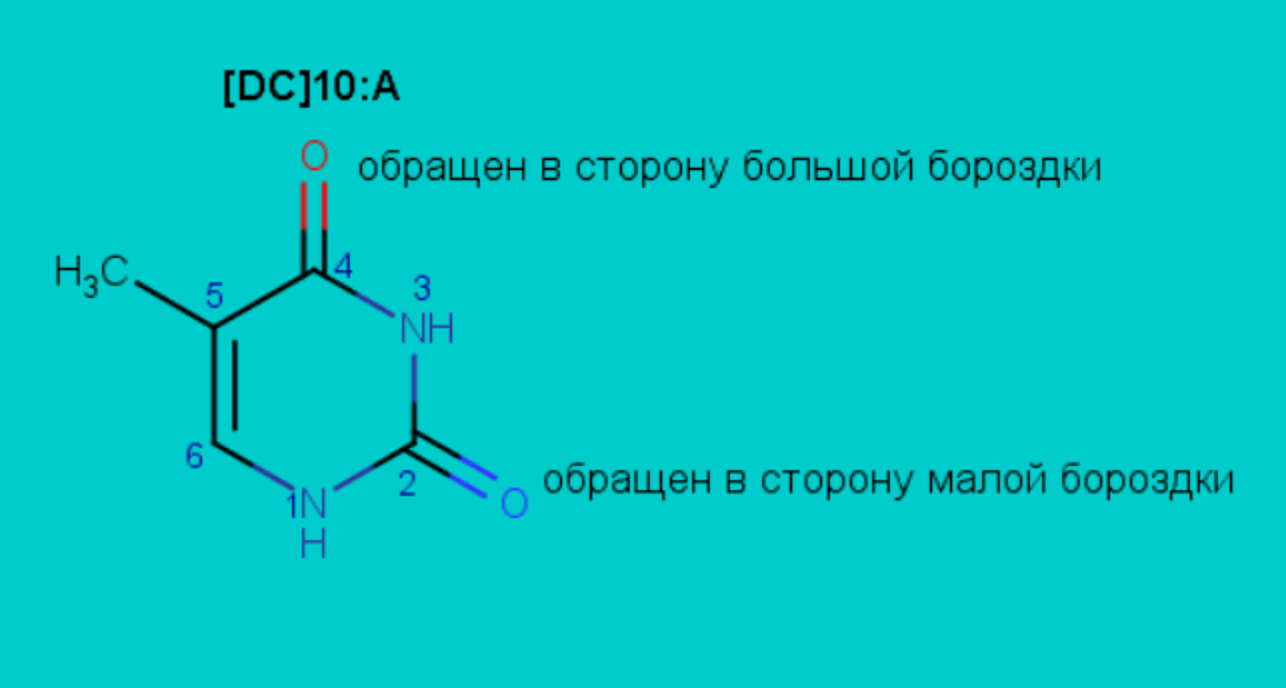
• В сторону большой бороздки обращен атом [DG]31:B.n4
• В сторону малой бороздки обращен атом [DG]31:B.n2
| A-форма | B-форма | Z-форма | |
|---|---|---|---|
| Тип спирали (правая или левая) | правая | правая | левая |
| Шаг спирали (Å) | 28.03 | 33.75 | 43.5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 16.81 [DG]37:B.P - [DT]6:A.P | 17.21 [DC]8:A.P - [DC]30:B.P | 18.30 [DC]6:A.P-[DC]32:B.P |
| Ширина малой бороздки | 7.98 [DC]8:A.P - [DA]27:B.P | 11.7 [DT]28:B.P - [DA]17:A.P | 7.20 [DG]29:B.P - [DG]15:A.P |
Задание 3. Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA
Упражнение 1
Ситуация: пакет 3DNA не умеет работать с новым форматом pdb. Воспользуемся программой remediator, чтобы перевести наши pdb в старый формат: remediator --old ''XXXX.pdb'' > ''XXXX_old.pdbЦели:
• Научиться определять торсионные углы нуклеотидов.
• Определить значения торсионных углов в заданной структуре тРНК; определить, на какую из форм ДНК больше всего похожи тяжи этой структуры.
Взята тРНК (PDB ID = 1GTS). На изображениях ниже представлены значения торсионных углов для данной тРНК.
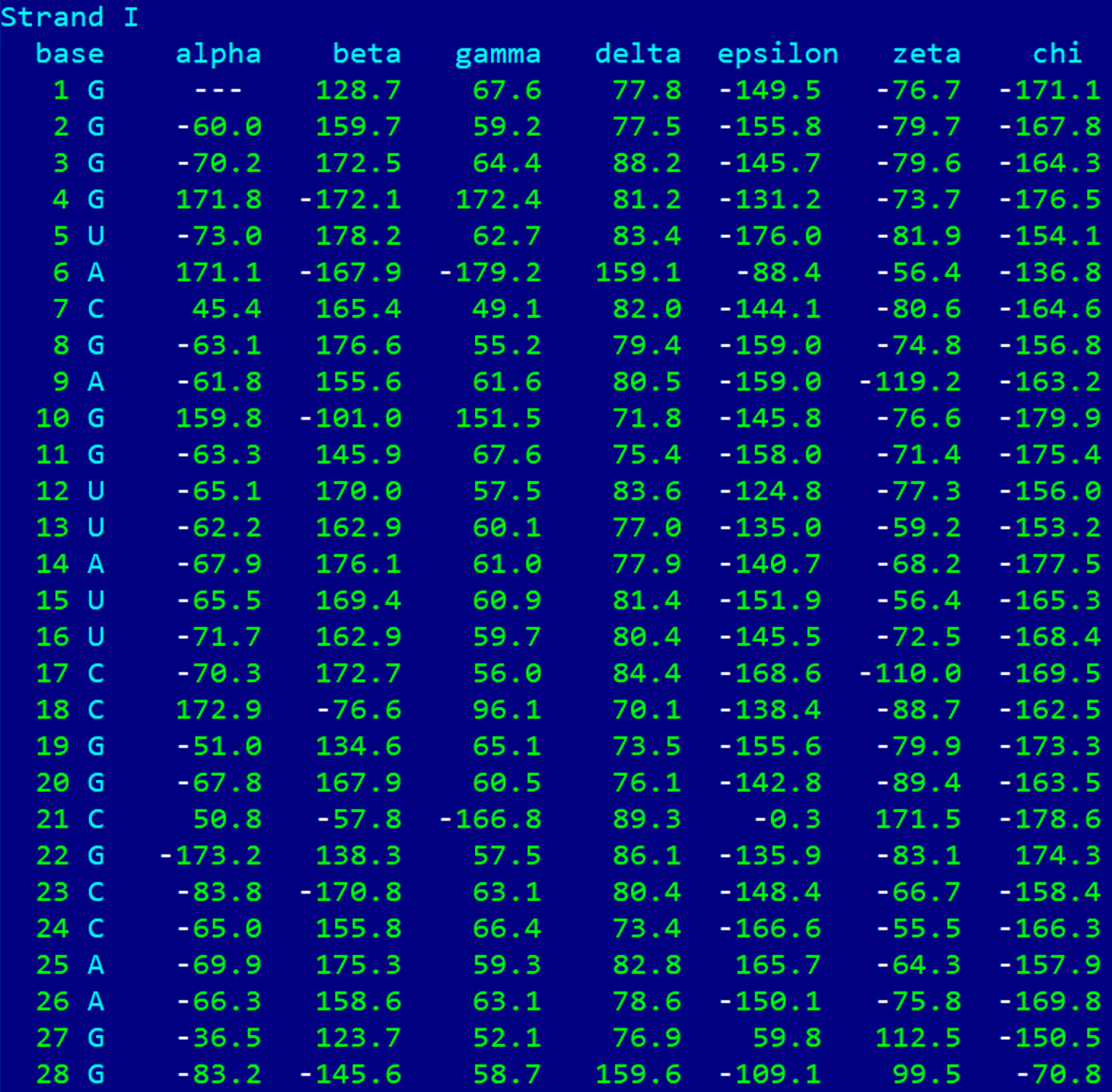
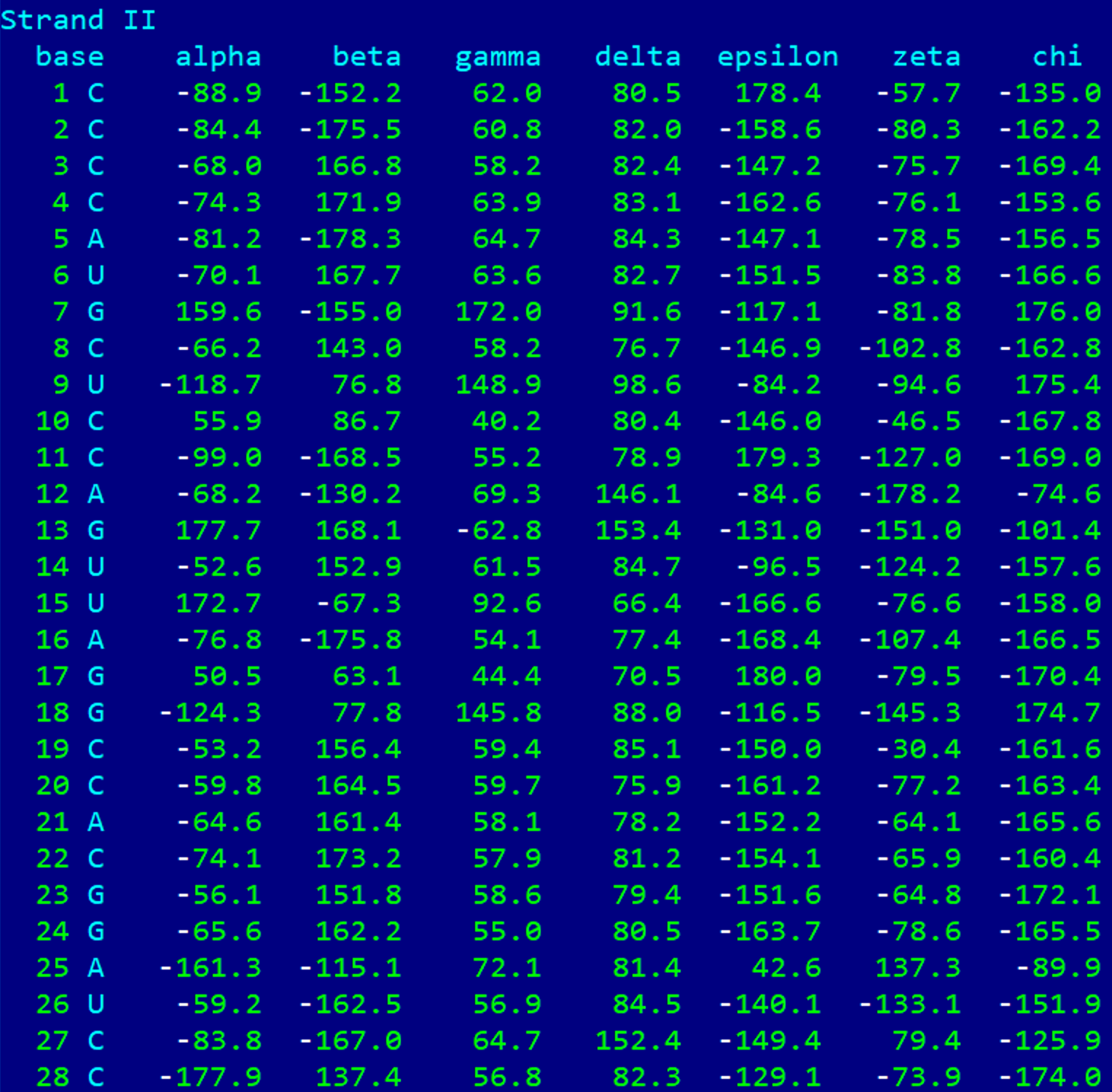
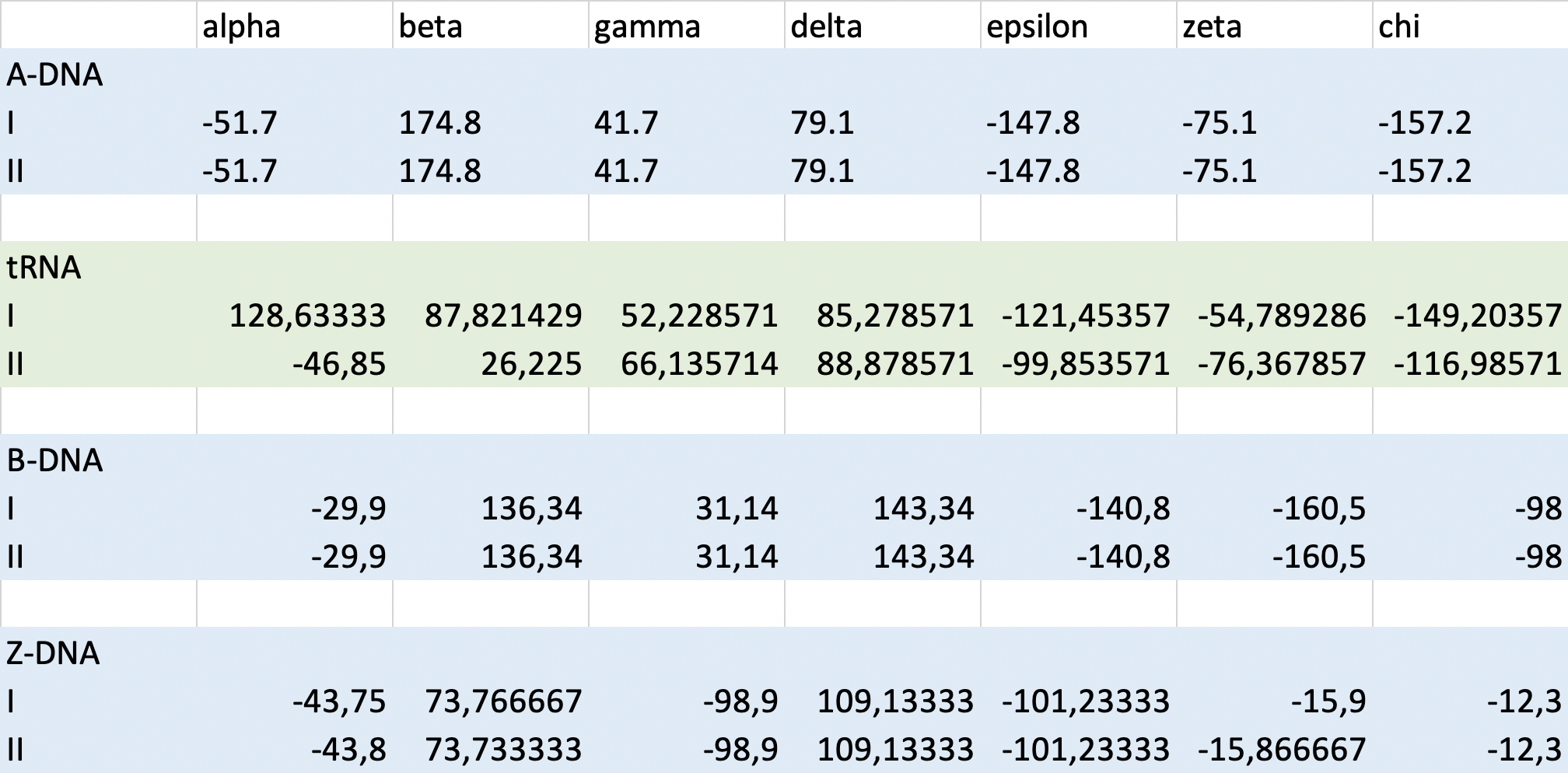
Ниже можно увидеть усредненные значения торсионных углов для каждой из форм ДНК и для тРНК. По значениям углов тРНК ближе всего к B-форме ДНК.
Упражнение 2
Цели:• Научиться определять структуру водородных связей между основаниями.
• Определить номера нуклеотидов, образующих стебли(stems) во вторичной структуре заданной тРНК.
• Определить неканонические пары оснований в структуре тРНК.
• Определить, есть дополнительные водородные связи в тРНК, стабилизирующие ее третичную структуру.
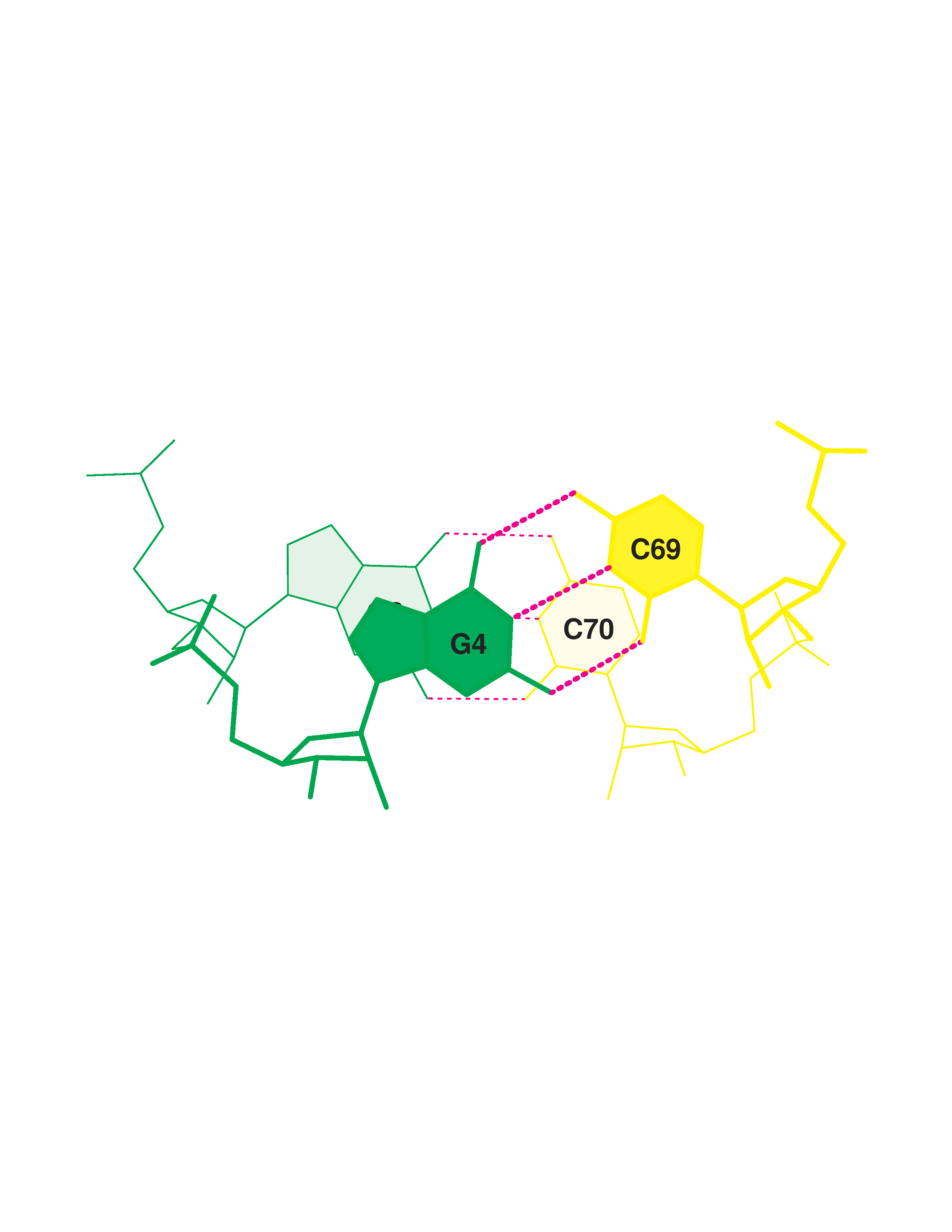
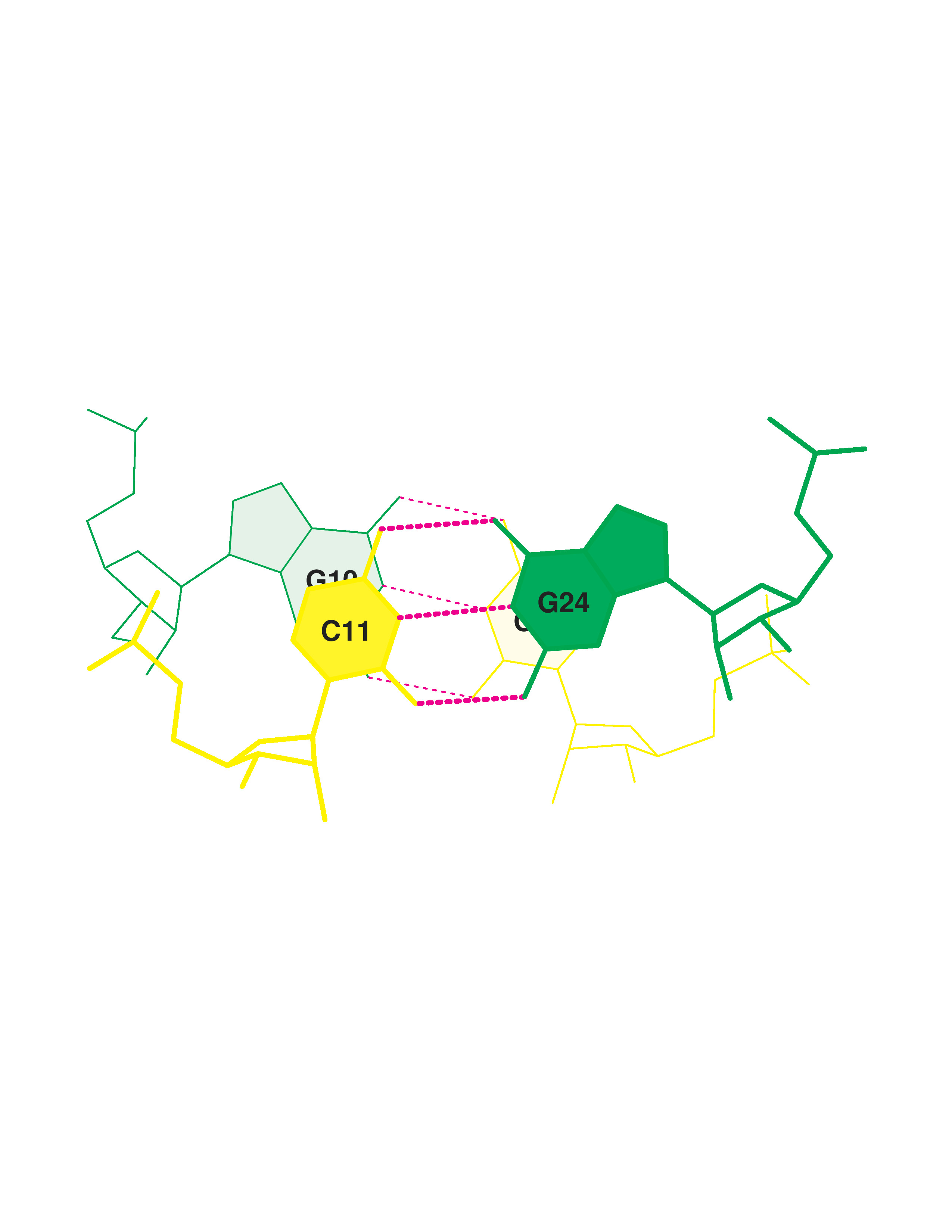
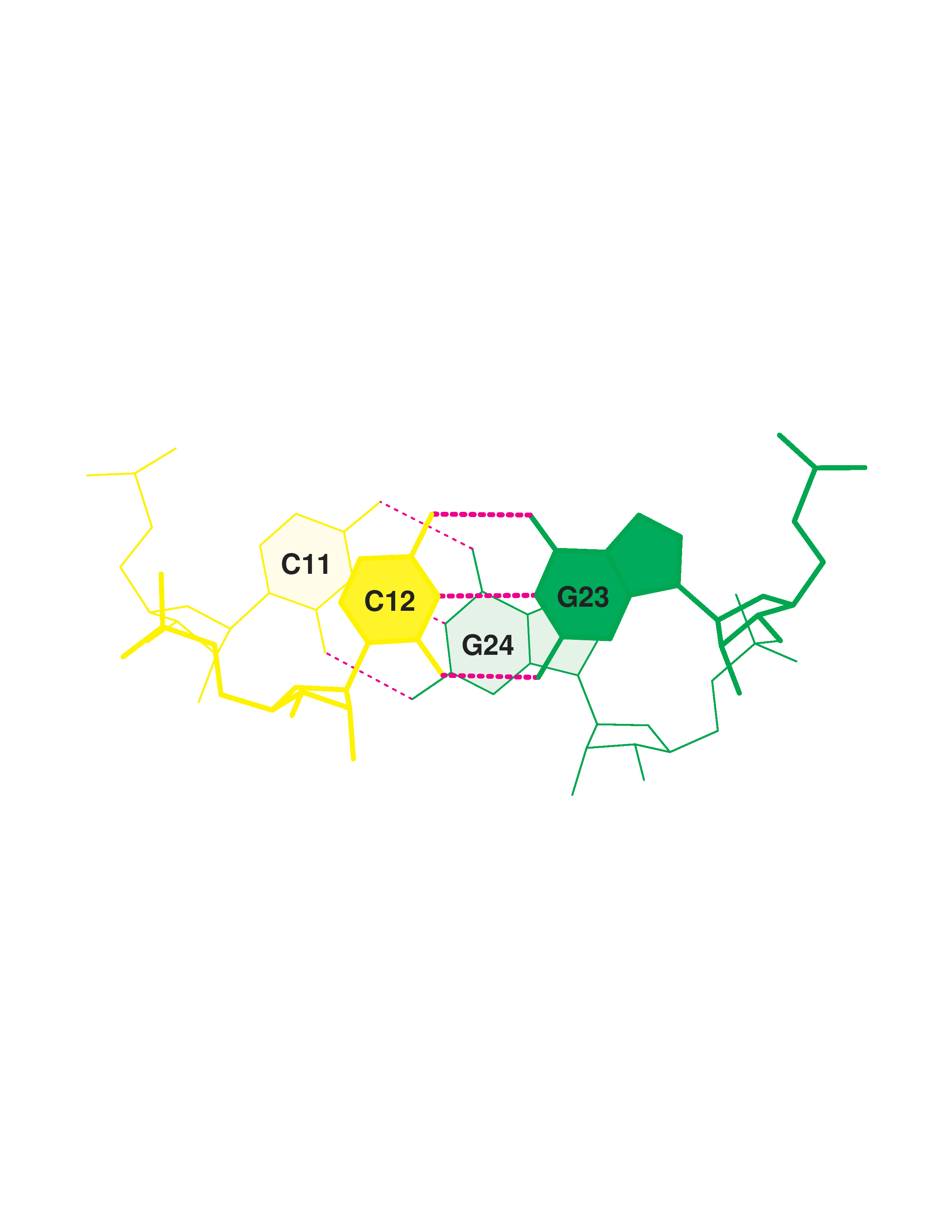
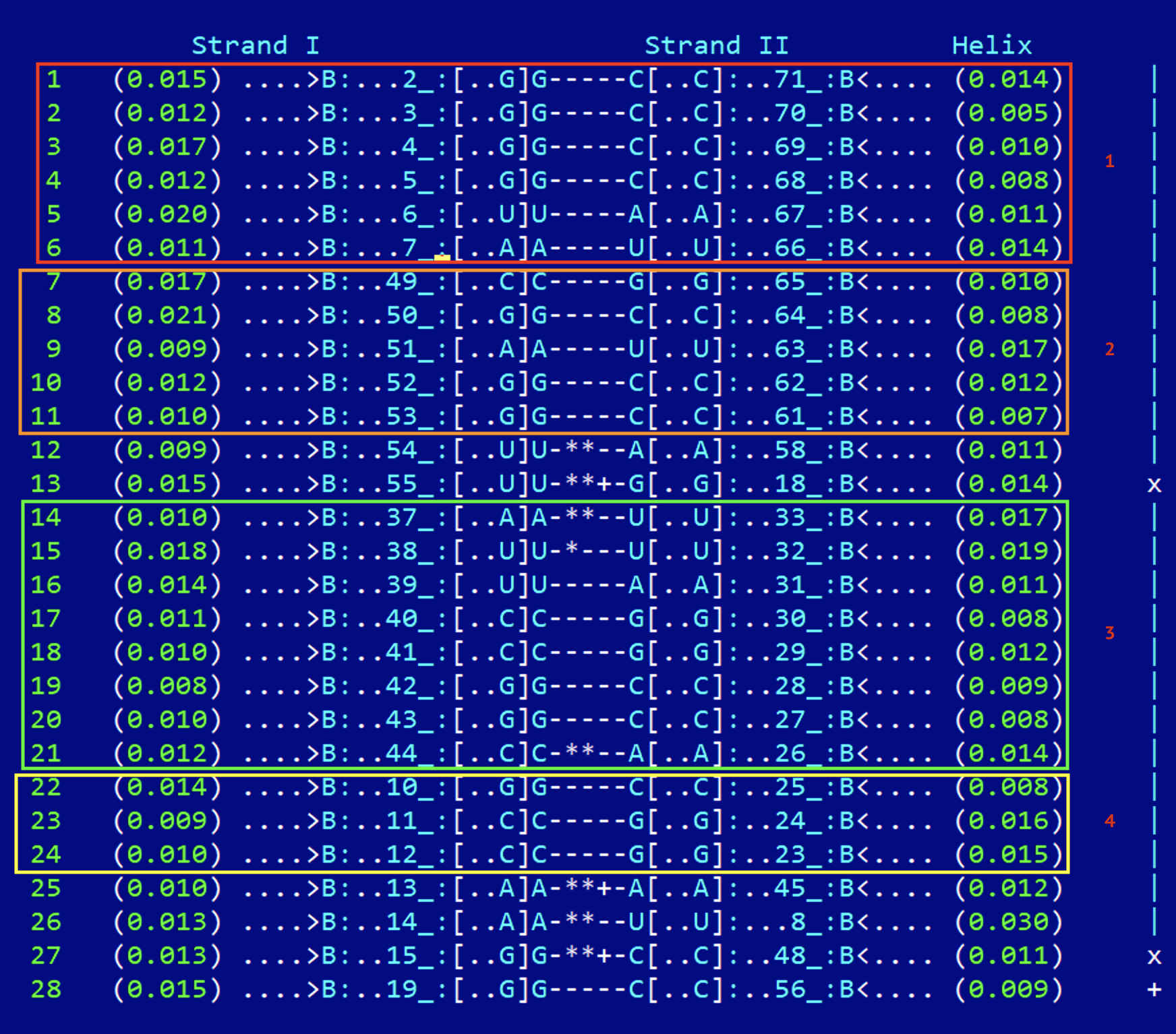
У данной тРНК - 4 стебля:
1. Нуклеотиды 2-7 (strand I) / нуклеотиды 66-71 (strand II)
2. Нуклеотиды 49-53 (strand I) / нуклеотиды 61-65 (strand II)
3. Нуклеотиды 37-44 (strand I) / нуклеотиды 26-33 (strand II)
4. Нуклеотиды 10-12 (strand I) / нуклеотиды 23-25 (strand II)
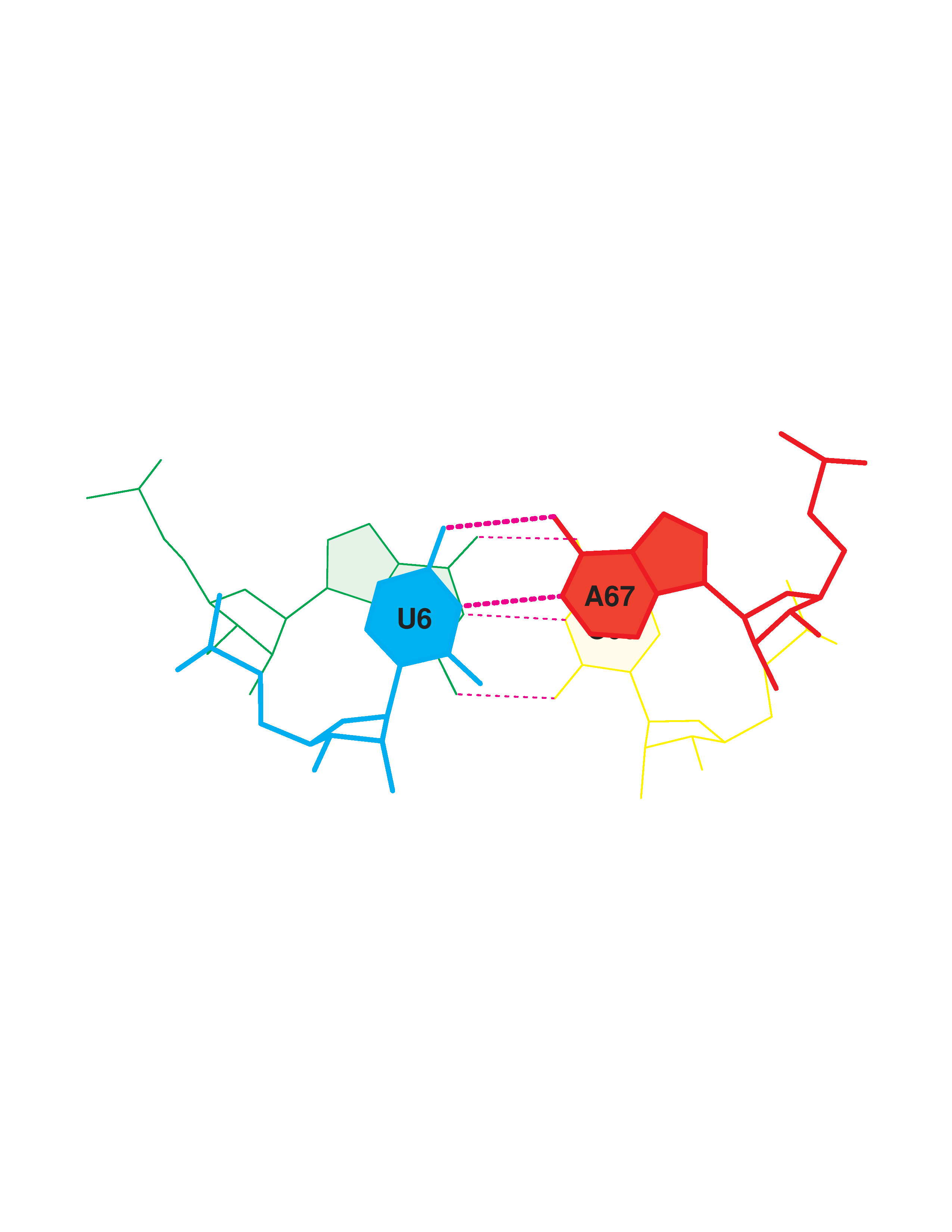
Неканонические взаимодействия:
[..U]U-**--A[..A]
[..U]U-**+-G[..G]
[..A]A-**--U[..U]
[..U]U-*---U[..U]
[..C]C-**--A[..A]
[..A]A-**+-A[..A]
[..A]A-**--U[..U]
[..G]G-**+-C[..C]
Дополнительная водородная связь, стабилизирующая третичную структуру, присутствует между нуклеотидами 19 (strand I) и 56 (strand II).
Упражнение 3
Цель - научиться находить возможные стекинг-взаимодействия. Это было мучительно, но результативно. Использовались секции 2, 4, 22 и 23.