Disclaimer!
Bсе вычисления, апплет, визуализация в JMol выполнены для комплекса с PDB ID = 1PP8. Nucplot применялся для комплекса PDB ID = 1PP7.
Задание 1. Предсказание вторичной структуры заданной тРНК
Упр. 1 и 2. Предсказание вторичной структуры тРНК путем поиска инвертированных повторов. Предсказание вторичной структуры тРНК по алгоритму Зукера
Скачать последовательность тРНК (PDB ID = 1GTS) в формате fasta
C помощью программы einverted из пакета EMBOSS найдены инвертированные участки в последовательности данной тРНК. Их оказалось 2 - они комплементарны друг другу. По факту они составляют один из трех стеблей тРНК.
Было бы, наверное, здорово увидеть больше инвертированных учатсков, поскольку применение команды RNAfold к данной структуре показало наличие у нее наличие трех петель, в то время при выполнении einverted даже при минимальном значении порога score (равному 1) можно судить о наличии у тРНК1 лишь одного стебеля (и это навевает сомнения).
Мне это показалось интересным, поэтому я еще визуализировала вторичную структуру этой тРНК в программе mfold, изменив значение параметра P с 5 на 15. Параметр P говорит, насколько структуры могут отличаться по своей энергии от вычисленного оптимума. Результат вычисления представлен на картинке ниже - эта структура была третьей по счету.
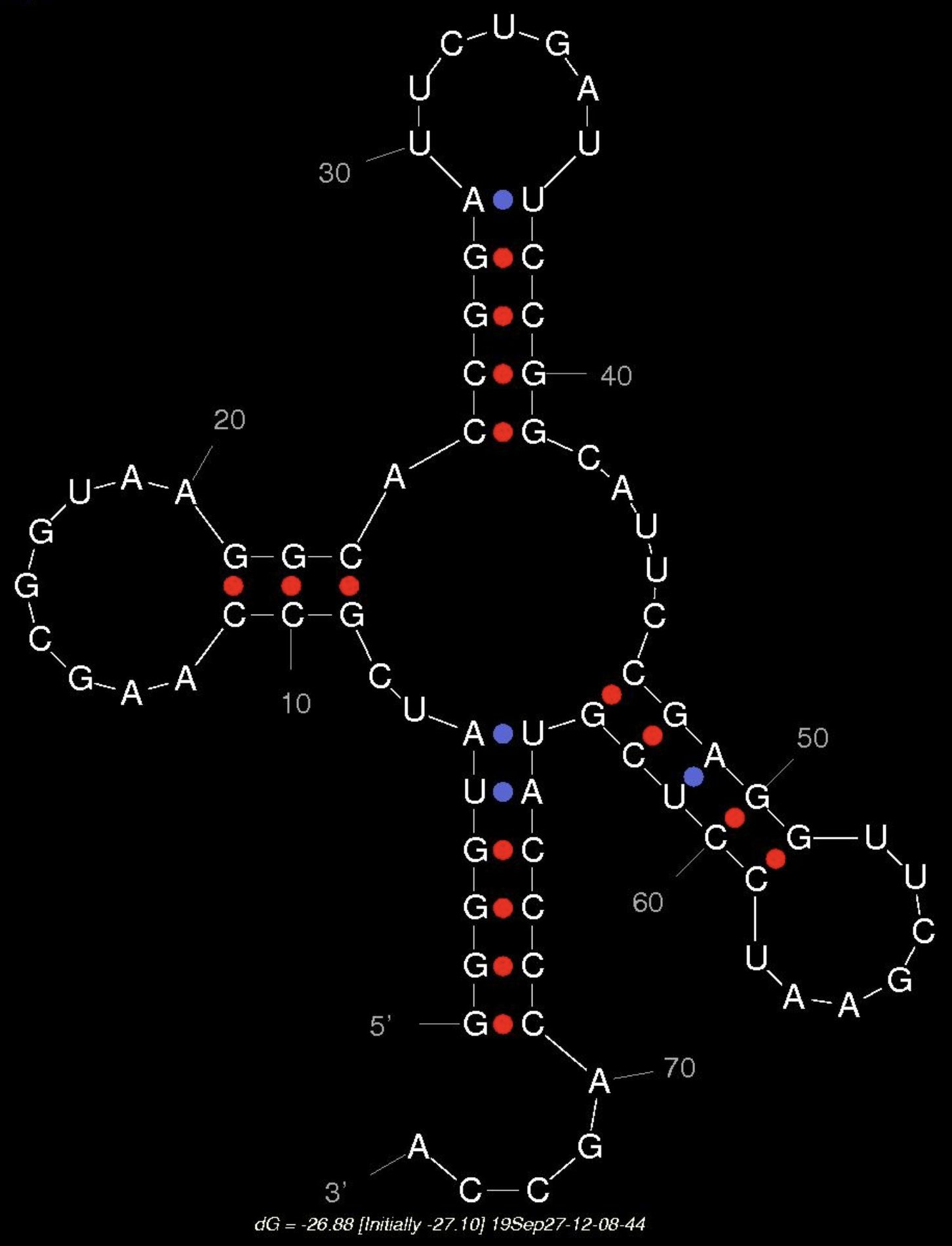
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
|---|---|---|---|
| Акцепторный стебель | 2-7, 66-71 | 2-7, 66-71 | 2-7, 66-71 |
| D-стебель | 10-12, 23-25 | 0 пн | 10-12, 23-25 |
| T-стебель | 49-53, 61-65 | 0 пн | 49-53, 61-65 |
| Антикодоновый стебель | 27-31, 39-43 | 0 пн | 27-31, 39-43 |
| Общее число канонических пар нуклеотидов | 19 пн | 6 пн | 19 пн |
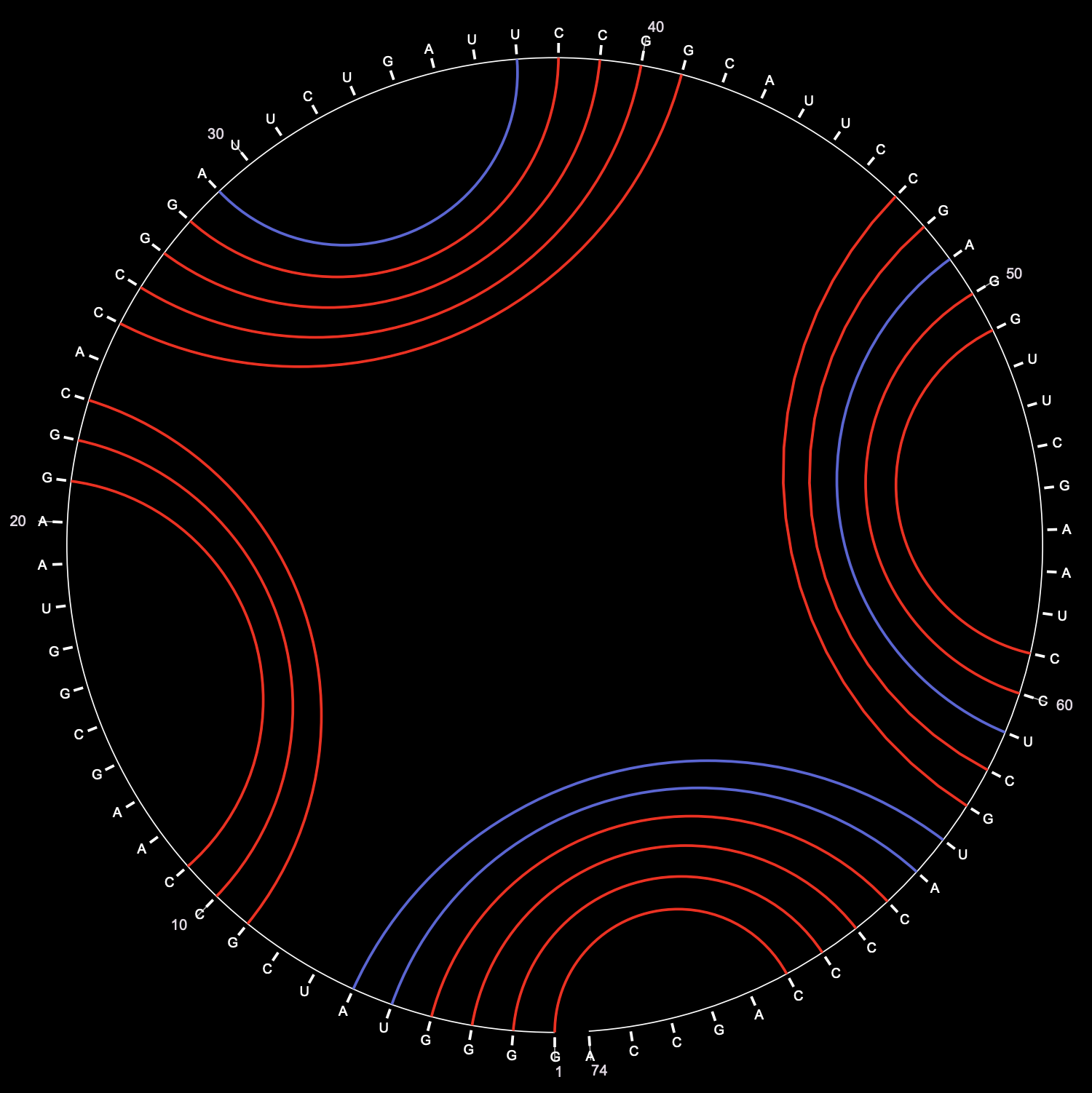
Ниже можно посмотреть на круговую диаграмму структуры 3 (тоже mfold).
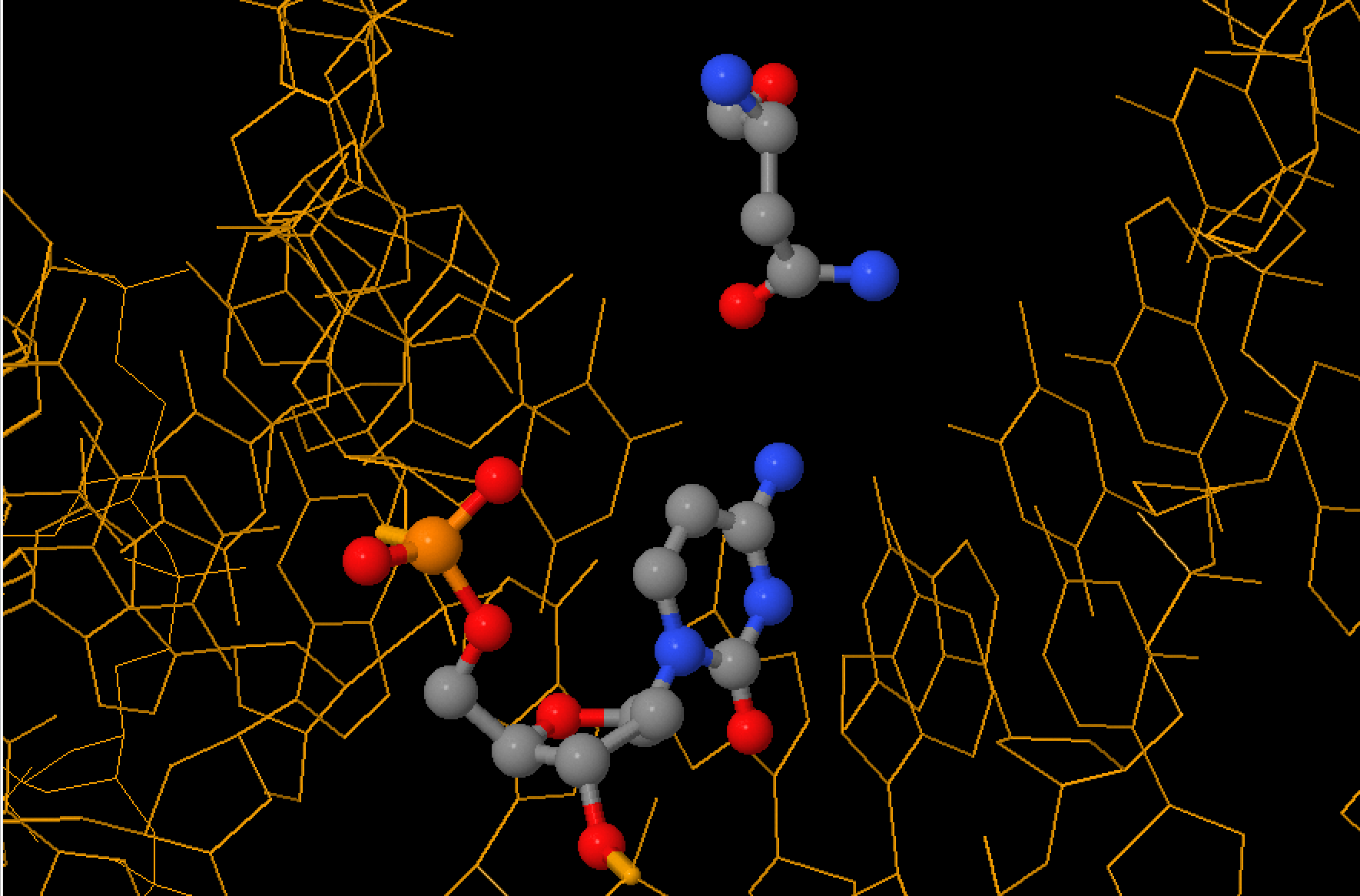
Задание 2. Поиск ДНК-белковых контактов в заданной структуре
Упр. 1. Define и компания
Скачать проекты по define и изображениям структур
Упр. 2. ДНК-белковые контакты
Скачать скрипт
В таблице 2 приведены данные о контактах разного типа в данном комплексе. Контакты были посчитаны с помощью скрипта для JMol и операции select within.
Полярными считались атомы кислорода и азота, а неполярными - атомы углерода, фосфора и серы.
Полярный контакт - ситуация, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5Å. Аналогично, неполярный контакт - пара неполярных атомов на расстоянии меньше 4.5Å.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
|---|---|---|---|
| остатками 2'-дезоксирибозы | 1 | 11 | 12 |
| остатками фосфорной кислоты | 8 | 14 | 22 |
| остатками азотистых оснований со стороны большой бороздки | 2 | 5 | 7 |
| остатками азотистых оснований со стороны малой бороздки | 1 | 0 | 1 |
Поскольку кусок ДНК в комплексе представлен в В-форме, азотистые основания находятся ближе к центру спирали, то больше всего контактов образуется с остатками фосфорной кислоты (белку проще с ними взаимодействовать). Неполярные контакты преобладают по количеству над полярными, потому что неполярных атомов в белке больше, чем полярных, а также потому, что неполянрые контакты длиннее (по определению, см. выше).
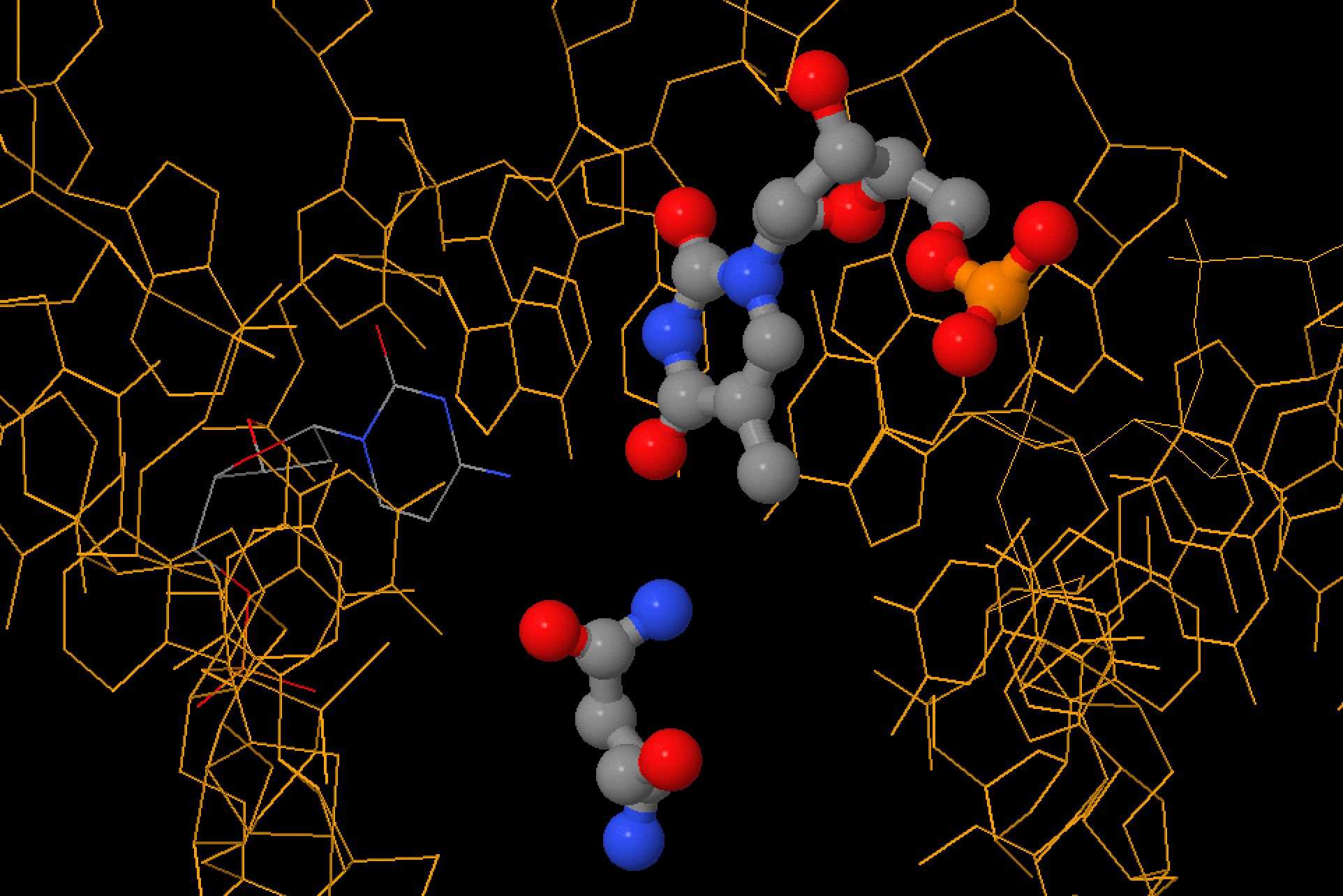
Упр. 3. Nucplot
С помощью nucplot было получено изображение ДНК-белковых контактов в структуре PDB ID = 1PP7 (эта структура близка к данной мне, но с визуализацией 1PP8 при выполнении тех же команд по неведомым мне причинам не получалось - "segmentation fault").
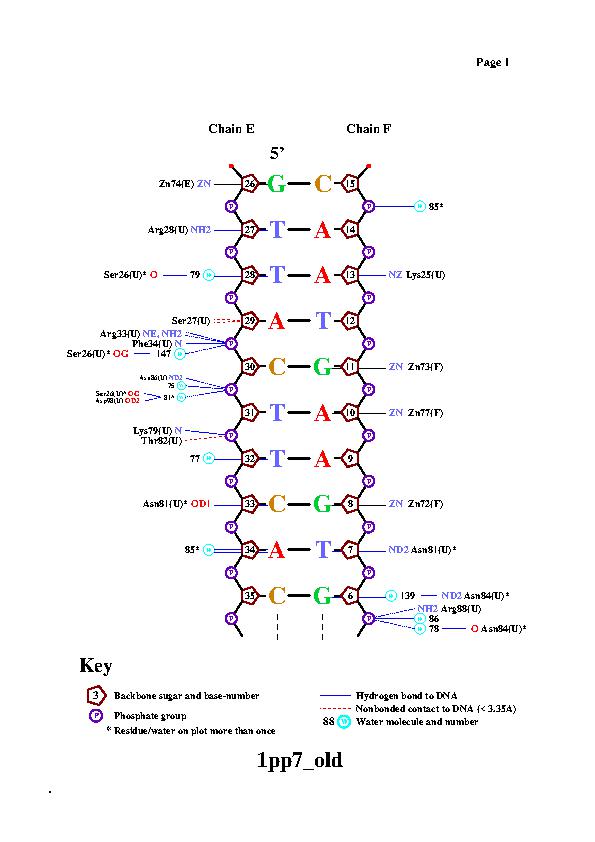
Упр. 4. "Выбрать на схеме"
Asn81 и Asn84 имеют наибольшее число контактов с ДНК (по 2). При этом, как мне кажется, наиболее важным является контакт Asn81 и С33, потому что с Asn81 более одного контакта, и контакт Asn81 и С33 довольно специфичен, поскольку в данном комплексе в принципе редки контакты аминокислотных остатков непосредственно с азотистыми основаниями (а этот контакт именно такой).